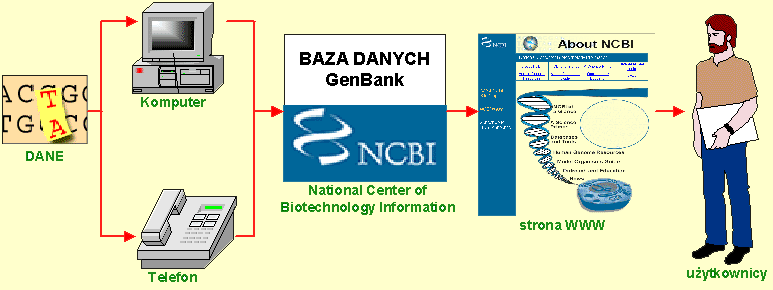
Dziedzina ta narodziła się na początku lat osiemdziesiątych, wraz z utworzeniem w USA bazy danych nazywanej GenBankiem. Amerykański Departament Energii opracował ten program, aby zbierać i przechowywać informacje o sekwencjach krótkich odcinków DNA, które naukowcy zaczęli właśnie sekwencjonować, badając różne organizmy. Na początku istnienia GenBank rzesze techników wpisywały sekwencje DNA używając klawiatur składających się z czterech klawiszy A, C, T oraz G. Z czasem opracowano protokoły przekazywania danych. Badacze mogli zadzwonić do GenBank i bezpośrednio wprowadzić do komputera dane o sekwencji kolejnych odcinków genomu. Administrację GenBank przeniesiono do Państwowego Centrum Informacji Biotechnologicznej (NCBI - National Center for Biotechnology Information) w National Institutes of Health. Po uruchomieniu serwisu WWW w Internecie naukowcy z całego świata uzyskali bezpłatny dostęp do danych zgromadzonych w GenBank. Internet fundamentalnie zmienił sposób przekazywania danych między ośrodkami naukowymi [24;80]. Kiedy w 1990 roku ruszył oficjalnie Projekt Poznania Ludzkiego Genomu, liczba informacji o zapisie genetycznym napływających do GenBanku zaczęła rosnąć w postępie wykładniczym (rys.1). Po wprowadzeniu w latach dziewięćdziesiątych metod wysoko wydajnego sekwencjonowania z wykorzystaniem robotyki, automatycznych sekwencjonerów i komputerów proces ten nabrał zawrotnego tempa. Do dnia, w którym oddano do druku lipcowy numer "Scientific American", GenBank zdążył zgromadzić dane o kolejności zapisu ponad 7 mld jednostek DNA [25].
Mniej więcej w tym samym czasie, gdy rozpoczęto realizację Projektu Poznania Ludzkiego Genomu, podobnymi badaniami zajęły się firmy prywatne i stworzyły własne obszerne bazy danych. Obecnie korporacje takie jak Incyte Genomics z Palo Alto w Kalifornii są w stanie odczytać w ciągu jednego dnia zapis genetyczny liczący około 20 mln par zasad DNA. Natomiast przedstawiciele firmy Celera Genomics oznajmili, że uzyskali wstępną wersję pełnej sekwencji genomu ludzkiego i zgromadzili na ten temat 50 TB (tetra bajtów) danych. Taka ilość informacji zajęłaby 80 tys. płyt kompaktowych [25]. Inne uzupełniające i wysokowydajne technologie, takie jak np. mikromacierze DNA (ang. microarray), są również źródłem tysięcy danych na minutę. Ponadto na Ziemi występuje ponad milion gatunków owadów, i liczba ich ciągle wzrasta. Wiele z nich jest ekonomicznie istotna. Znaczenie wielu danych biologicznych wzrasta ze względu na wysoką efektywność sprzętu laboratoryjnego i technologii analizy [24;80]. GenBank i wyżej wymienione firmy są jednak zaledwie częścią bioinformacji. Istnieją inne publiczne i prywatne bazy danych zawierające informacje o ekspresji genów (czyli o tym, kiedy i gdzie geny są włączane), tzw. polimorfizmie sekwencji nukleotydowych (czyli drobnych różnicach genetycznych między poszczególnymi osobnikami), budowie różnych białek, oraz schematach ich wzajemnych oddziaływań.
| 1843 | Richard Owen opisał różnicę między homologią i analogią |
| 1858 | Darwin & Wallace opublikowali referaty o teorii ewolucji |
| 1859 | Charles Darwin opublikował "The Origin of Species" |
| 1864 | Ernst Häckel - zarys współczesnych elementów zoologicznej klasyfikacji |
| 1865 | Gregory Mendel ustanowił prawa dziedziczności w pracy "Badania nad mieszańcami roślin...". |
| 1868 | Friedrich Miescher - odkrycie kwasu nukleinowego |
| 1902 | Chromosomowa teoria dziedziczności zaproponowana przez pracujących niezależnie Suttona i Boveri'ego |
| 1930 | Badania nad naturą kwasu nukleinowego |
| 1936 | Alan Turing - wynalezienie uniwersalnej maszyny obliczeniowej tzw. Turing maschine |
| 1941 | Beadle & Tatum - "Genetyczna kontrola reakcji biochemicznych" -
pierwszy naukowy dowód na hipotezę jeden-gen-jeden-enzym |
| 1948 | Chude Shannon - jeden z twórców teorii informacji |
| 1951 | Pauling & Corey proponują strukturę alfa-spirali i beta-kartki |
| 1953 | Watson & Crick model podwójnej helisy DNA |
| 1953 | Sanger F., Thompson E., Tuppy H. oznaczenie aminokwasowej sekwencji łańcuchów A i B insuliny |
| 1970 | Opublikowano szczegóły dotyczące algorytmu Needleman-Wunsch służącego do wyrównywania sekwencji |
| 1971 | Tomlinson - wynalezienie programu e-mail |
| 1972 | Paul Berg i współpracownicy stworzyli pierwszą zrekombinowaną cząsteczkę DNA |
| 1975 | Southern E. M.publikuje eksperymentalne dane dotyczące techniki Southern Blot dla specyficznych sekwencji DNA |
| 1977 | Maxam A., Gilbert W., Sanger F. - metody sekwencjonowania DNA |
| 1978 | Założono połączenie Usenet pomiędzy Duke i Uniwersytetem w Północnej Karolinie |
| 1980 | Opublikowano pierwszy gen sekwencji organizmu - FX174 |
| 1981 | Wprowadzenie na rynek pierwszego komputera PC |
| 1981 | Opublikowano algorytm Smith-Waterman |
| 1983 | Mullis K. wynalezienie reakcji PCR, metody umożliwiającej klonowanie fragmentów DNA |
| 1985 | Mullis K. i współpracownicy - opisanie reakcji PCR |
| 1986 | Utworzenie bazy danych SWISS-PROT na Uniwersytecie w Genewie i EMBL (European Molecular Biology Laboratory) |
| 1988 | Założono Narodowe Centrum Informacji Biotechnologicznej (NCBI) |
| 1988 | Person i Lupman - algorytm FASTA do porównywania sekwencji |
| 1988 | Założono HUGO - Human Genom Organization - Projekt Poznania Ludzkiego Genomu |
| 1990 | Altschul, Gish, Miller, Myers, Liman - wdrożono program BLAST (Basic local alignment search tool) |
| 1990 | Opublikowano specyfikacje HTTP 1.0. Berners-Lee opublikował pierwszy dokument HTML |
| 1991 | Opisano utworzenie i użycie sekwencji ESTs |
| 1993 | Oficjalne przeniesienie bazy danych GenBank pod administrację NCBI - National Center for Biotechnology Information |
| 1995 | Zsekwencjonowano genom Heamophilius influenzea |
| 1995 | Zsekwencjonowano genom Mycoplasma genitalium |
| 1996 | Zsekwencjonowano genom drożdży piekarniczych Saccharomyces cerevisiae |
| 1997 | Zsekwencjonowano genom drożdży piekarniczych Saccharomyces cerevisiae |
| 1997 | Klonowanie pierwszego ssaka (owieczki Dolly) w Instytucie Roślin w Szkocji |
| 1997 | Opublikowano genom E.coli |
| 1997 | Opublikowano algorytm PSI - BLAST - Altschul, Madden, Zhang, Miller, Liman |
| 1998 | Opublikowano kompletny genom drożdży piekarskich Caenorhabditis elegans |
| 1998 | Kompletne genomy ukazują rozległość duplikakcji gen/białko sekwencja/struktura |
| 1999 | PSIMAP (Protein Structural Interactome Map) zawierający pierwszy, pełny system interakcji genomów -
pierwszy system filogenetycznych interakcji, pierwsza mapa utworzona przy użyciu domen białek, pierwszy globalny system interakcji |
| 1999 | Marcotte, Pellegrini, Rice, Yeates, Eisenberg - Rozpoznanie funkcji białek oraz oddziaływania białko-białko
na podstawie sekwencji genomowych |
| 2000 | Opublikowano genom Pseudomonas aeruginosa |
| 2000 | Zsekwencjonowano genom A. thaliana |
| 2000 | Zsekwencjonowano genom D. melanogaster |
| 2001 | Opublikowano genom człowieka |
| 2002 | Opublikowano pełną sekwencję genomu myszy domowej |
| 2004 | Opublikowano szkic sekwencji genomu brązowego szczura norweskiego, Rattus norvegicus |