Chromosomowa lokalizacja kilku genów uznana jest za skorelowaną ludzkim genem BRCA1 związanym z rakiem piersi, wyróżniony za pomocą zapytania Map Viewer "BRCA1" (http://www.ncbi.nlm.nih.gov/mapview/map_search.cgi?chr=hum_chr.inf&query).
Książki
NCBI zawiera linki do PubMed oraz innych zasobów, gdzie oferuje biomedyczne książki oraz monografie, które umieszczone są we współpracy z autorami i wydawcami. Autoryzowane przez NCBI zasoby są dostępne w tej bazie danych tak jak w: NCBI Handbook (internetowy podręcznik NCBI - link http://www.ncbi.nlm.nih.gov/books/bv.fcgi?call=bv.View..ShowTOC&rid=handbook.TOC&depth=2a), Coffee Break (zbiór krótkich raportów najnowszych biologicznych odkryć) oraz Geny i choroby (zbiór artykułów które omawiają geny oraz choroby z nimi związane).
Bookshelf - półka z ksiązkami, zawierająca powiększającą sie kolekcję książek biologicznych, które mogą być odnalezione bezposrednia przez wpisanie koncepcji-słowa klucza w "textbox" oraz wybranie "Go".
Czasopisma
Wyszukiwarka czasopism w PubMed, razem z linkami do rekordów w tych czasopismach.
- Wyszukiwanie przez temat, tytuł czasopisma lub skrót, ISSN (Miedzynarodowy Znormalizowany Numer Wydawnictwa Ciaglego), lub wyszukiwanie przez składnik tematu (http://www.nlm.nih.gov/bsd/journals/subjects.html).
- Wyszukiwanie z ograniczeniami w czasopismach PubMed oraz/lub aktualnie indeksowanych.
(http://www.ncbi.nlm.nih.gov/journals/Limits)
- Lista wszystkich czasopism Entrez (http://www.ncbi.nlm.nih.gov/entrez/citmatch_help.html#JournalLists) z linkami do dostępnych stron internetowych pełno tekstowych.
MeSH
JEst słownikiem kontrolowanym przez NLM's (Narodowa Bibioteka Medyczna Stanów Zjednoczonych) do indeksowania artykułów. Terminologia MeSH dostarcza logicznych dróg do uzyskania informacji, które mogą być użyte w innej terminologii dla tych samych koncepcji (projektów).
NLM Katalog - Narodowa Bibioteka Medyczna Stanów Zjednoczonych
Katalog dający dostęp do danych bibliografii NLM dla czasopism, książek, filmów (audiowizualizacje), oprogramowania komputerowe, zasobów elektronicznych oraz innych materiałów. Linki do zbiorów biblioteki są w LocatorPlus, dostępnym online katalogu NLM
OMIA - ta baza danych jest katalogiem genów, dziedzicznych zaburzeń oraz cech gatunkowych innych niż ludzkie i mysie autorem, których jest prof Frank Nicholas z Uniwersytetu w Sydney, Australia.
Dostępne w sieci Mendlowskie dziedziczenie u zwierząt (OMIA) jest bazą danych genów, dziedzicznych zaburzeń oraz cech gatunkowych (innych niż ludzkie i mysie) autorem, których jest prof Frank Nicholas z Uniwersytetu w Sydney, Australia, oraz wiele innych osób które pomogały mu w tym przez wiele lat. BAza danych zawiera informacje tekstowe oraz bibliografie, również linki do istotnych rekordów z bazy danych OMIM, PubMed, Gene, i niedługo do fenotypowej bazy danych NCBI. OMIA jest ręcznie tworzona. Jeśli widzisz jakiś błąd albo chciałbyś brać udział w pracy, skontaktuj się z OMIA.
Przewodnik wyszukiwarki OMIA
Wejdż w jeden lub więcej termin: arthris (http://www.ncbi.nlm.nih.gov/sites/entrez?Db=omia&Cmd=DetailsSearch&Term=arthritis%5BAll+Fields%5D) albo chicken (http://www.ncbi.nlm.nih.gov/sites/entrez?Db=omia&Cmd=DetailsSearch&Term=chicken%5BAll+Fields%5D)
OMIM
Ta baza danych jest katalogiem genów człowieka oraz zaburzeń genetycznych, których autorem i redaktorem jest dr Victor A.McKusick oraz jego współpracownicy z Uniwesytetu Johna Hopkinsa oraz z innych miejsc, które rozwijane są na stronie przez NCBI.
Dostępne w sieci Mendlowskie dziedziczenie u człowieka. OMIM jest podstawowym, wiarygodnym oraz aktualnym kompendium ludzkich genów i genetycznych fenotypów. Pełno tekstowy, przegląd bibliografii w OMIM zawiera informacje wszystkich znanych mendlowskich zabrzueń genetycznych oraz ponad 12 tys genów.OMIM skupia się na relacjach pomiedzy fenotypem a genotypem. Jest ona uaktualniana codziennie, oraz zawartość zawiera łącza do innych genetycznych zasobów.
Ta baza danych była zainicjowana we wczesnych latach 60 przez Dr Victora A.McKusicka jako katalog mendlowskich cech oraz zaburzeń, nosiła ona tytuł Mendlowskie zaburzenia u czlowieka (MIM). Dwanaście edycji MIM były publikowane miedzy rokiem 1966 oraz 1998. Wersja online - OMIM- powstała w roku 1985 przez współpracę pomiędzy Medyczna Biblioteką Narodową oraz Medyczną Biblioteką Wiliama H. Welch'a na Uniwersytecie Johna Hopkinsa. Ogólnie dostępną na internecie została od roku 1987. W 1995, OMIM była rozwijana przez www dla NCBI - Narodowe Centrum Informacji Biotechnologicznej.
OMIM powstaje pod dyrekcją dr Ada Hamosh na Instytucie Medycyny Genetycznej McKusick-Nathans oraz na Uniwersytecie Johna Hopkinsa.
OMIM jest prowadzony głównie dla użytku lekarzy oraz innych profesjonalistów skoncentrowanych na genetycznych zaburzeniach przez badaczy genetycznych oraz przez studentów zaawansowanych w naukach ścisłych i medycynie. Odkąd OMIM jest dostępne publiczne, odbiorcy poszukujący informacji na temat osobistej medycyny oraz warunków genetycznych mają możliwość konsultacji z wykwalifikowanym lekarzem w specjalizacji diagnostyki oraz uzyskania odpowiedzi na osobiste pytania.
PubMed
Serwis Narodowej Biblioteki Medycznej (USA), która pozwala na dostęp do ponad 17mln cytatów z MEDLINE (światowej bazy piśmiennictwa medycznego) i dodatkowo czasopism nauk naturalnych. PubMed zawiera linki do wielu stron zawierających pełnotekstowe artykuły i inne pokrewne zasoby.
Aby rozpocząć prace w PubMed wprowadź jedno lub więcej poszukiwanych terminów. Poszukiwanymi terminami mogą być tytuły, autorzy lub czasopisma.
Dostęp do NIH (Narodowy Instytut Zdrowia (USA)) może dotyczyć też ciebie. Czy NIH znalazło twoja prace? Wtedy Twój maszynopis musi być dostępny w Centrali PubMed. Jak? Jeśli publikujesz w jednym z tych czasopism http://publicaccess.nih.gov/submit_process_journals.htm one zadbają o cały proces. Jeśli publikujesz gdziekolwiek indziej zdeponuj swój maszynopis w Centrali PubMed korzystając z jednej z opcji opisanej na stronie http://publicaccess.nih.gov/
PubMed to serwis Narodowej Biblioteki Medycznej (USA), który obejmuje ponad 18 milionów cytatów z MEDLINE i innych biomedycznych czasopism (artykuły od 1948). PubMed zawiera linki do pełnotekstowych artykułów i innych pokrewnych zasobów.
PubMed Central - PMC - jest U.S. Narodowym Instytutem Zdrowia dostępnych zarchiwizowanych cyfrowo czasopism biomedycznych oraz nauk naturalnych.
Aby otrzymywać powiadomienie o nowych czasopismach oraz innych większych aktulizacjach produktu PMC należy: dołączych do PMC News listy mailowej lub zapisać się do PMC News RSS Feed.
Wszystkie artykuły w PMC są bezpłatne (czasami na odkładanie podstaw). Niektóre czasopisma nie są dostępne dla Open Access. Dowiedz sie co to oznacza: - http://www.pubmedcentral.nih.gov/about/openftlist.html
Narzędzia PMC zawierają OAI serwis, który dostarcza XML z pełną treścią niektórych artykułow, funkcję wyszukiwania skryptów PMC i linki do konkretnych artykułów PMC z Twojej witryny, oraz więcej...
Szukasz nowoczesnego czasopisma DTD? Rzuć okiem na Journal Publishing XML DTD and schema. (http://dtd.nlm.nih.gov/publishing/)
Chodzi o zachowanie i dostępność: cyfrowy do wszystkich poprzednich numerów wielu czasopism PMC. (http://www.pubmedcentral.nih.gov/about/scanning.html)
W PMC lista czasopism składa się z czasopism depozytowego materiału w PMC
Książki
We współpracy z wydawcami książek NCBI oferuje cyfrowe książki biomedyczne i monografie z linkami do PubMed – biomedycznej bibliograficznej bazy danych. http://www.ncbi.nlm.nih.gov/sites/entrez?db=books
‘Rakowe chromosomy’
Zawiera cytogenetyczne, kliniczne i polecane informacje zaczerpnięte ze zintegrowanych danych NCI (NAtional Cancer Institute - USE) Mitelman Database of Chromosome Aberrations in Cancer, NCI Recurrent Aberrations in Cancer database, oraz bazy danych NCI/NCBI SKY/M-FISH & CGH.
Trzy bazy danych, baza danych NCI/NCBI SKY/M-FISH & CGH, NCI Mitelman Database of Chromosome Aberrations in Cancer, oraz NCI Recurrent Aberrations in Cancer , są teraz zintegrowane w systemie NCBI Entrez jako Chromosomy Rakowe.
Szukaj cytogenetycznych, klinicznych i polecanych informacje. Zapytania są realizowane tak jak w innych bazach Entrez np. PubMed czy nukleotydowej. Szukaj trzema metodami: okno zapytania w Entrez, proste wyszukiwanie lub zaawansowane wyszukiwanie.
Przejrzyj publikację http://www.ncbi.nlm.nih.gov/pubmed/15934046?dopt=Abstract o tej bazie danych i o bazie danych SKY/M-FISH & CGH.
Ostatnia aktualizacja bazy danych
- dane Mitelman - 27 maja 2008
- dane SKY/M-FISH & CGH 9 czerwca 2008
Przewodnik wyszukiwania dla okna zapytania w Entrez
Wprowadz zapytanie w pole (na szarym tle) na górze strony i naciśnij „Go”
Przykład:
ISCN (International System for Human Cytogenetic Nomenclature) Symbol anormalnej krótkiej formy chromosomu, zgodny z ISCN np. t(9;22)(q34;q11) też może być wprowadzony.
Aby składać zapytania bardziej zaawansowania używaj także tej samej składni, skorzystaj z Preview/Index lub wyszukiwania zaawansowanego: http://www.ncbi.nlm.nih.gov/sites/entrez?db=cancerchromosomes&cmd=ShowAdvQryPage
CDD
Baza danych zachowanych domen (domen konserwatywnych), kolekcja uliniowień (porównań) i profili reprezentujących konserwatywne w ewolucji molekularnej domeny białek. Wybierz „Domeny” z rozwijanej listy menu w Entrez.
Wskazówki do znajdowania domen
Nowa pomocna aplikacja
CDTree przeglądarką NCBI hierarchii domen białkowych. Jako pomocna aplikacja dla Twojej przeglądarki internetowej, pozwala na interaktywny pokaz sekwencji klastrów, taksonów, domen i architektury. CDTree jest dostosowany do Windowa. Wychodzi w pakiecie z Cn3D, molekularną strukturą i przeglądem alignmentów, a także funkcjonuje jako samodzielne narzędzie do analizy rodzin białek. Więcej: http://www.ncbi.nlm.nih.gov/Structure/cdtree/cdtree.shtml
O bazie danych:
Conserved Domain Database (CDD) zawiera modele domen białkowych importowane ze źródeł zewnęrznych, takich jak Pfam i SMART, i zarządzana przez NCBI. CDD zawiera ponad 12,000 takich modeli i posiada linki do innych baz danych NCBI, zawierających sekwencje proteinowe, bibliografie cytatów i taksonomy.
Nukleotydowe serce (sekwencje nukleotydowe)
Zawiera wszystkie nukleotydowe sekwencje niezawarte w podzbiorach EST lub GSS.
Nukleotydowa baza danych Entrez to kolekacja sekwencji z kilku źródeł włączając GenBank, RefSeq i PDB. Liczba baz w tych bazach danych ciągle rośnie w wykładniczym tempie.
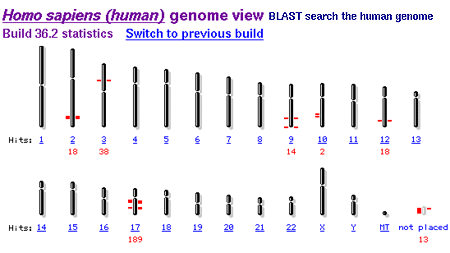
Genom człowieka
Badania zasobów genomu ludzkiego http://www.ncbi.nlm.nih.gov/genome/guide/ lub przeszukiwanie sekwencji genomu ludzkiego przy użyciu Map Viewer http://www.ncbi.nlm.nih.gov/mapview/ .
Budowa genomu ludzkiego
Sekwencja DNA genomu ludzkiego została poznana w kwietniu 2003. Aktualna wersja jest is listed as a build number na stronie Genome View http://www.ncbi.nlm.nih.gov/mapview/map_search.cgi?taxid=9606 i zawiera dodatkowo zestaw statystyk http://www.ncbi.nlm.nih.gov/mapview/stats/BuildStats.cgi?taxid=9606&build=36&ver=2 i not publikowanych http://www.ncbi.nlm.nih.gov/genome/guide/human/release_notes.html.

Chromosomowa lokalizacja kilku genów uznana jest za skorelowaną ludzkim genem BRCA1 związanym z rakiem piersi, wyróżniony za pomocą zapytania Map Viewer "BRCA1" (http://www.ncbi.nlm.nih.gov/mapview/map_search.cgi?chr=hum_chr.inf&query).
Domeny trójwymiarowe (3D)
Zawiera domeny białek ze strukturalnej bazy danych Entrez.
Wskazówki wyszukiwania struktur:
EST
Nukleotydowa baza danych zawierająca tylko rekordy Expressed Sequence Tag, czyli tzw. etykiety ekspresyjne (sekwencji pochodzących z aktywnych genów).
Baza danych etykiet ekspresyjnych (dbEST) http://www.ncbi.nlm.nih.gov/pubmed/8401577?dopt=Abstract jest elementem GenBanku, który zawiera sekwencje i inne informacje o sekwencjach "single-pass" cDNA lub "Expressed Sequence Tags", z licznych organizmów.
Krótka relacja z historii ludzkiech EST w GenBank jest dostępna http://www.ncbi.nlm.nih.gov/pubmed/7667885?dopt=Abstract.
"Genome Directory" opublikowany w Nature (t. 377, publikacja 6547S, 28 Wrzesień 1995).
Geny
Geny i związane z nimi informacje dla wielu organizmów oraz człowieka.
Entrez Gene jest bazą danych genów, z genomów RefSeq http://www.ncbi.nlm.nih.gov/RefSeq/ , oraz określonych przez sekwencje zlokalizowane również/lub w Map Viewer NCBI.
Nowości w Entrez Gene http://www.ncbi.nlm.nih.gov/projects/Gene/GeneNews.html
Proste wyszukiwania:
Znajdz geny przez:
Genom
Genomy ponad 1,200 organizmów mogą być znalezione w tej baize danych, pokazująza zarówno kompletne sekwencje oraz te których sekwencjonowanie trawa.
Genomowa baza danych dostarcza widoków na różnorodne genomy, komplety chromosomów, mapy sekwencji z sekwencjami przylegajacymi i zintegrowane mapy genetyczne i fizyczne. Baza danych jest zorganizowana w 6 głównych grup organizmów: Archaea, Bacteria, Eukaryotae, Viruses, Viroids, and Plasmids
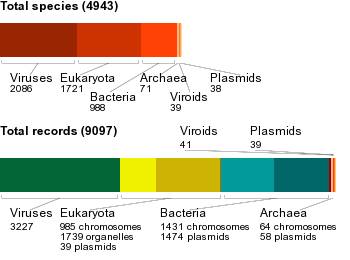
Projekt Genom
Kolekcja pełnych i niekompletne (w toku) wyników sekwencjonowań na dużą skalę, montaż, adnotacje oraz tworzenie projektów dla organizmów komórkowych.
dbGaP
Skojarzone dane genotypowe i fenotypowe.
GENSAT
Atlas genów ulegających ekspresji centralnego układu nerwowego myszy.
Witamy w GENSAT
Projekt GENSAT ma na celu zmapowanie ekspresji genów w centralnym układzie nerwowym myszy, używając zarówno hybrydyzacji in situ jak i technik transgenicznych. Kliknij tutaj, aby uzyskać więcej informacji na temat metod stosowanych w GENSAT. http://www.ncbi.nlm.nih.gov/projects/gensat/static/methods_brief.shtml
Znajdź wskazówki:
*. Szukaj kryteriów włączając nazwy genów, symbole genów, pseudonimy i synonimy genów, wiek myszy i protokoły obrazowania.
* Wiek myszy ograniczony jest do E10.5 (zarodkowych dni 10,5), E15.5 (zarodkowych dni 15,5), P7 (pourodzeniowy dzień 7) i dorosłych (dla dorosłych).
* Łącz frazy wraz z AND, OR i NOT
Przykładowe wyszukiwania:
- drd* : szukaj wszystkich genów, których symbole zaczynają sie od "DRD"
- dopamine AND e10.5[age] : szukaj wszystkich genów, których nazwy zawierają słowo ‘dopamine’, posiadających zdjęcia dostępne myszy zebranych podczas zarodkowych dni 10.5.
Zbiory danych GEO
Geny ulegające ekspresji i molekularna obfitość zgrupowanych danych z NCBI Gene Expression Omnibus, ekspresja genów i macierz hybrydyzacji.
Profile GEO
Indywidualna ekspresja genów i zasoby molekularne profili zmontowane z zasobów GEO
GSS
A Nucleotide database subset that contains only Genome Survey Sequence records. Podzestaw nukleotydowej bazy danych, która zawiera tylko rekordy Genome Survey Sequence.
Sekwencje genomu Survey Database (GSS) to podział GenBanku, który jest podobny do poddziału EST, z wyjątkiem tego, że większość sekwencji to oryginalne sekwencje genomowe a nie cDNA (mRNA). Należy zauważyć, że dwie klasy (exon trapped products and gene trapped products) mogą być uzyskana pośrednio poprzez cDNA. Należy zachować ostrożność podczas analizy sekwencji z obu tych klas, kiedy może mieć miejsce splicing i sekwencja reprezentowana w rekordzie może zostać przerwana kiedy porównujemy ja do genomowej sekwencji. Dział GSS zawiera (ale nie wyłącznie) następujące rodzaje danych:
Chociaż sekwencje bazy danych GSS są włączone do Działu GSS GenBanku, adnotacje w dbGSS są bardziej wszechstronne i zawierają szczegółowe informacje na temat autorów, warunkach doświadczalnych i genetycznych map lokalizacji.
Geny homologiczne
Zawiera homologi wśród odnotowanych genów kilku całkowicie zsekwencjonowanych genomów eukariotycznych.
Czasopisma
Zawiera wszystkie czasopisma zawarte w PubMed i zapewnia dostęp do rejestrów dla tego czasopisma w bazie danych.
MeSH
Kontrolowane słownictwo NLM używane do indeksowania artykułów w PubMed.
Strona Internetowa NCBI
Wyszukiwarki calej strony NCBI
Katalog NLM
Katalog książek, czasopism i audiowizualizacji w kolekcji NLM.
Online Mendelian Inheritance in Animals (OMIA):
Baza danych genów, dziedzicznych zaburzeń i cech gatunków zwierząt (innych niż ludzkie i mysie) autoryzowane przez Profesoar z Uniwersytetu w Sydney, Franka Nicholasa.
Online Mendelian Inheritance in Man (OMIM):
A katalog ludzkich genów i zaburzeń genetycznych autorem i redaktorem których jest Dr Victor A. McKusick i współpracownicy.
PopSet
Zestaw danych dotyczących badań populacji, które zostały pobrane do analizy ewolucyjnego pokrewieństwa w populacji.
Sonda
Publiczny rejestr reagentów kwasu nukleinowego zaprojektowanych do stosowania w szerokim zakresie badań biomedycznych aplikacji, wraz z informacją o dystrybutorach reagentów, skuteczności sond i podobieństwach sekwencji.
Baza danych sekwencji białkowych
Zbiór sekwencji białka z różnych źródeł, w tym Swiss-Prot, PIR, PRF, WPB i translacji z regionów kodujących pochodzących z GenBank i RefSeq.
PubChem BioAssay
Bioaktywność substancji chemicznych.
PubChem Compound
Małe cząsteczki struktur chemicznych.
PubChem Substance
Substancje chemiczne badane pod kątem bioaktywnoci.
PubMed
Dostęp do ponad 15 milionów cytatów z MEDLINE i dodatkowych czasopism nauk naturalnych.
Centrala PubMed
Cyfrowe archiwum pełnotekstowych, literatura czasopism nauk przyrodniczych, która jest w pełni wyszukiwalna i połączona linkami z bazą danych PubMed.
SNP
Repozytorium polimorfizmu pojedynczych substytucji nkleotydowych i krótkich delecji i inercji.
Struktura
The Molecular Modeling Database zawiera trójwymiarowe struktury molekularne, w tym białka i polinukleotydy.
Taksonomia
Zawiera opisy wszystkich organizmów, które są reprezentowane w genetycznej bazy danych z co najmniej jednego nukleotydu lub sekwencji białkowych.
UniGene
System automatycznego podziału sekwencji GenBanku w niezbędny zestaw genów zorientowanych w grupy zawierające sekwencje, które stanowią unikalny (nie powtarzający się) genu, jak również związanych z nim informacji, takich jak rodzaj tkanki i lokalizacja na mapie.
Każda pozycja w UniGen to zestaw transkryptów które pojawiają się z tych samych lokus transkrypcyjnych (genów lub pseudogenów), razem z informacjami o podobieństwach białek, ekspresji genu, klonach cDNA i lokalizacji w genomie.
UniSTS
Raporty informacje o markerach lub STSach, sekwencjach primerów, rozmiarze produktu i informacji o mapowaniu.
NCBI przyjmuje genomowe dane do swoich baz danych z projektów sekwencjonowania z całego świata i służy jako fundament do badań bioinformatycznych
GenBank:
Zbiór wszystkich publicznie dostępnych nukleotydowych i aminokwasowych sekwencji.
Wyszukiwarka tekstowa i podobieństw:
Przeglądarka entrez:
GenBank(nukleotydy i białka), PubMed(MEDLINE), struktury 3D, genomy oraz PopSet baz danych. Nukleotydowy rekord może znajdować się w trzech sektorach: CoreNucleotide, dbEST, dbGSS. Wyszukiwanie Entrez może przeszukiwać te trzy bazy danych razem lub każdą z osobna.
BLAST Wyszukiwanie podobieństw sekwencji
Pytanie dotyczące sekwencji nukleotydowej lub w białku w stosunku do określonej bazy danych zostaje odszukane za pomocą algorytmów BLAST. Zapisy nukleotydowe GenBank, które znajdują się w oddzielnych bazach danych muszą zostać przeszukane samodzielnie. Obejmują one dbEST i sbGSS plus wiele bez danych CoreNucleotide, w tym: nr, htgs, WGS oraz env_nt. Zobacz BLASTinfo(http://www.ncbi.nlm.nih.gov/blast/producttable.shtml) aby dowiedzieć się więcej informacji na temat licznych baz danych BLAST.
Wyszukiwarka dbEST
Baza danych o ekspresji sekwencji (Database of Expressed Sequence Tags)
Wyszukiwarka dbSTS
Baza danych o o sekwencjach (Database of Sequence Tagged Sites)
Wyszukiwarka dbGSS
Baza danych przeglądu genomowych sekwecji (Database of Genome Survey Sequences)
Informacje o dostępie do GenBanku
Klient sieci/serwer aplikacji (http://www.ncbi.nlm.nih.gov/Web/Search/client.html)
BLAST sieci
FTP (http://www.ncbi.nlm.nih.gov/Genbank/GenBankFtp.html)
Pełne udostępnienie i dzienne aktualizacje GenBank.
EST bazy danych:
Zbiór sekwencji podległych ekspresji lub krótkie jednoniciowe sekwencje czytane z mRNA (cDNA).
dbEST- co to jest?
Jest to dział GenBanku, zawiera dane o sekwencjach i inne informacje na temat jednoniciowego cDNA lub "ekspresyjne fragmenty sekwencji" z liczbą organizmów. Krótkie rozliczenie historii ludzkiego EST jest dostępne (http://www.ncbi.nlm.nih.gov/pubmed/7667885?dopt=Abstract). Ponadto, można zajrzeć do specjalnego "Genom Directory" w Nature (t.377, issue 6547S, 29 września 1995 rok)
Inne sposoby uzyskania dostępu do dbEST
(http://www.ncbi.nlm.nih.gov/dbEST/dbEST_access.html)
Jak dodać dane:
(http://www.ncbi.nlm.nih.gov/dbEST/how_to_submit.html)
Informacje na temat obecnego wydania:
Liczba EST (http://www.ncbi.nlm.nih.gov/dbEST/dbEST_summary.html) - podsumowanie dbEST przez organizmy
Obraz (I.M.A.G.E) konsorcjum klonów
"Ślad danych" na sekwencje pochodzące z wizerunku (IMAGE) klonów jest dostępna z Centrum Waszyngtońskiego Uniwersytetu Sekwencjonowania Genomu. Ślady są pobierane przy użyciu identyfikatora i oznakowane "EST nazwa" w dbEST rekordu (np.yb01a01.s1, odpowiadające w GenBank numerowi dostępu T48601). Zauważ że ten identyfikator może być również odnaleziony w definicji Rekordu GenBanku (http://www.ncbi.nlm.nih.gov/entrez/viewer.fcgi?tool=portal&db=nucest&term=T48601&query_key=3&dopt=gb&dispmax=20&page=1&qty=1&WebEnv=16Wq-uuvO_tlvhremiIv_b9QoirZVgjraoTVWbRHRcWcO1KhHBsLp_gN1rumy7pdYrZVLbXZadI8a4_FRKXWzRLkR%4025625EFF9341FDF0_0038SID&WebEnvRq=1)
Naukowe klony DNA z IMAGE wizerunku konsorcjum bibliotek są już dostępne od kilku dystrybutorów. Klony można zamowić za pomocą identyfikatora, oznakowane są "Clone ID" w dbEST rekordu ( np. 69846, odpowiadający numerowi dostępu GenBank T48601). Tak jak wcześniej, ten identyfikator jest również dostępny w definicji Rekordu GenBank.
GSS bazy danych:
Baza danych z badań o sekwencjach genomowych lub krótkich, jednoniciowych genomowych sekwencji.
GSS Co to jest?
Dział GSS w GenBank, jest podobny do działu EST, z wyjątkiem, że tu większość sekwencji jest pochodzenia genomowego, a nie cDNA (mRNA). Należy zauważyć ze dwie klasy (ekzon uzyskanych produktów i geny uzyskanych produktów) mogą wywodzić się pośrednio z cDNA. Należy zachować ostrożnośc podczas analizy sekwencji z obu tych klas, tak jak przy splicingu, oraz sekwencje reprezentowane w rekordzie, mogą być poprzerywane, kiedy będą porównywane z genomową sekwencją. Dział GSS zawiera (ale nie ogranicza się do tego) następujące typy danych:
- losowe "jednoniciowe" ("single pass read") sekwencje genomowe
- zakończenia kosmidów/BAC/YAC sekwecji
- sekwencje egzonowe (exon trapped genomic sequences)
- Alu PCR sekwencje
- oznaczone transpozonowe sekwencje (transpozon-tagged seguences)
Sekcja 1.3.3 GenBanku 96.0 dostarcza dodatkowych informacji o dziale GSS.
Chociaz dbGSS sekwencji jest w dziale GSS GenBanku, przypisy w dbGSS są bardziej uzupełniające oraz zawierają szczegółowe informacje o współautorach, warunkach eksperymentu oraz lokalizacji na genetycznej mapie.
Inne sposoby dostępu do dbGSS - (http://www.ncbi.nlm.nih.gov/dbGSS/dbGSS_access.html)
Jak przekazywać dane - (http://www.ncbi.nlm.nih.gov/dbGSS/how_to_submit.html)
dbGSS podsumowanie przez organizm
Informacje o obecnym wydaniu (http://www.ncbi.nlm.nih.gov/dbGSS/dbGSS_summary.html)
Komentarze i pytania: info@ncbi.nlm.nih.gov
HomoloGene:
Narzędzie "homologia genów", porównuje ono sekwencje nukleotydów w organizmach w celu identyfikacji potencjalnych ortologów.
HomoloGene jest systemem automatycznego wykrywania homologów genów wśród poszczególnych kompletnie zsekwencjonowanych eukariotycznych genomów.
HomoloGene release 62 Statystyka
Początkowa liczba genów z pełnych genomów, liczba genów umieszczona w grupie homologów oraz liczba grup dla każdego gatunku.
Mamy niedawno przyjętą nową procedurę wyszukiwania, która korzysta z sekwencji aminokwasów (blastp) w celu znalezienia bardziej odległych związków, ale odnosi sie ona nadal do obliczeń sekwencji DNA niektórych statystyk. Dobranie strategii jest kierowanie przez drzewo taksonomiczne, tak by organizmy bardziej związane są porównywane pierwsze. Ponadto, HomoloGen obejmują obecnie paralogi oprócz ortologów.
źródła dodatkowych informacji
Wpisy HomoloGen są adotpowane z homologią oraz informacjami fenotypowymi pochodzących z następujących źródeł: OMIM, MGI, ZFIN, SGD, COG, FlyBase.
HTG- bazy danych:
Zbiór wysokiej-przepustowości sekwencji genomu od dużych ośrodków sekwencjonowania genomu, zawiera niedokończone i gotowe sekwecje.
Dział HTG został stworzony aby pomieścić rosnącą potrzebę, aby dane o niedokońca zsekwencjonowanych genomam były dostępne społeczności naukowej. Uzykano to dzięki skoordynowanemu wysiłkowi pomiedzy Miedzynarodową Nukleotydową Bazą Danych DDBJ, EMBL oraz GenBank.Dział HTG zawiera niedokończone sekwencje DNA generowane przez centra sekwencjonowania o wysokiej przepustowości. Sekwencje danych tego działu są dostępne dla wyszukiwarki homologii BLAST przeciw "htgs" bazie danych albo "month" bazie danych, która zawiera wszystkie nowe zgłoszenia z wcześniejszego miesiąca. Dział HTG GenBanku został opisany w artykule "Genome Research" Quellette i Boguski.
Lokalizacja rekordów HTG
Niedokończonym sekwencjom HTG większym niż 2kb jest przypisywany numer dostępu oraz są one umieszczane w dziale HTG. Typowy rekord HTG może składać się z w pierwszej kolejności z danych sekwencji generowanych z kosmidu, BAC, YAC albo klonu P1, które razem stanowią więcej niż 2 kb oraz zawierają jedną lub więcej przerw. Pojedynczy numer dostępu jest przypisany do tej kolekcji sekwencji, i każdy rekord zawiera wyraźne wskazanie etapu (etap 1 i 2) oraz wyraźne ostrzeżenie, że są to niedokończone sekwencje i mogą zawierać błedy. Numer dostępu nie zmienia się gdy sekwencja jest aktualizowana; tylko najnowsza wersja rekordu HTG pozostaje w GenBanku. Zapisy pozostają w fazie 2, aż sekwencja osiągnie poziom gotowa. W związku z tym że istnieją rekordy z brakiem przerw, ale w fazie 2, ponieważ ich ocena jakości jest zbyt niska by osiągnać fazę 3.Gdy HTG będą gotowe (etap 3) zachowują ten sam numer dostępu, ale przenoszone są do odpowiednich działów pierwotnego GenBanku. Przykład złożenia (jeden numer dostępu), który przeszedł przez etap 1,2,3: http://www.ncbi.nlm.nih.gov/HTGS/examples.html. Nową fazą dla sekwencji jest faza 0. Faza zero będzie użyta by sprawdzić czy inne centrum w tym momencie nie zaczeło sekwencjonować tego klonu. Jeśli tak, klon może nigdy nie zostać skończony. Jeśli nie, będzie on podlegał sekwencjonowaniu przez fazę 1,2,3.
Status - faza 0
Lokalizacja - dział HTG
Definicja - od jednego do kilku "czytań" pojedynczego klonu
Status - faza 1
Lokalizacja - HTG dział
Definicja - niekończone, może być nieuporząkowany, niezorientowane fragmenty, z przerwami.
Status - faza 2
Lokalizacja - dział HTG
Definicja - niedokończone, uporządkowany, zorientowane fragmenty, bez lub z przerwami.
Status - faza 3
Lokalizacja - dział podstawowy
Definicja - dokończone, bez przerw (z lub bez odpowiednimi adnotacjami)
Jest pewna swobodna (zmienność) w budowaniu definicji faz. Na przykład, chociaż większość propozycji reprezentuje zbiór nieuporządkowany lub uporządkowany sekwencji wywodzących się z pojedynczego kosmidu, BAC albo klonu PAC, są przypadki gdzie każda indywidualna sekwencja była dodana do fazy 1, później uaktualniona jako faza 3, a następnie zmontowana do fazy 3. Jest to bardziej dogodne dla NCBI by przyjmować 1 propozycję przynależności do grupy nieuporządkowanej albo uporządkowanej sekwencji niż przyjmować 200 propozycji reprezentujących każdy nieuporządkowany lub uporządkowany fragment długiej sekwencji.
Wiadomośc ze końce BAC powinny być dodane jako rekord GSS (http://www.ncbi.nlm.nih.gov/dbGSS/index.html)
Dodawanie rekordów do HTG
Sekwencje są przygotowywane do dodania przez użycie narzędzi oprogramowania NCBI: Sequin, tbl2asn albo fa2htgs lub przez własne narzędzia oprogramowania. Każde centrum posługuje się katalogiem FTP, gdzie nowe albo uaktualniane sekwencje się znajdują. Dane pojawiające się w katalogu FTP są poddawane katalogowej obróbce na podstawie codziennej, oraz obróbka rozpoczyna się natychmiastowo (aby uzyskać więcej informacji: http://www.ncbi.nlm.nih.gov/HTGS/processing.html). Centra sekwencjonowania dodające sekwencje HTG powinny skontaktować się z NCBI (info@ncbi.nlm.nih.gov) aby uzyskać dostęp do konta FTP. Aby uzyskać instrukcje do przygotowania HTG proszę obejrzeć (http://www.ncbi.nlm.nih.gov/HTGS/subinfo.html)
HTG FAQ
Wszelkie dodatkowe źródła informacji o procesie dodawania HTG (http://www.ncbi.nlm.nih.gov/HTGS/faq.html), mogą one przyśpieszyć proces dodawania przez podkreślenie specyficznych przykładów. Jeśli potrzebujesz więcej informacji, proszę się skontaktować: info@ncbi.nlm.nih.gov w celu uzyskania dodatkowych informacji.
Baza danych SNPs
Centralny zbiór dla obu pojedynczych, podstawowych zmian nukleotydowych, oraz krótkich delecji, oraz polimorfizmu insercji.
RefSeq:
Baza danych niezmiennie odniesiona do standardów sekwencji, wliczając genomowe DNA, mRNA oraz białka dla znanych genów. Wiele współpracy, zarówno w NCBI jak i zewnętrznych grupach, wsparcie naszych wysiłków zbierania danych.
NCBI referencyjnych sekwencji.
Referencyjne sekwencje (RefSeq), ta kolekcja ma na celu zapewnienie kompleksowego, zintegrowanego, dobrze usytuowanego w sekwencji włączenia sekwencji genomowego DNA, transkryptów i protein. RefSeq jest podstawą do zastosowań medycznych, funkcjonalnych i różnorodnych badań; zapewnia stabilne odniesienia dla genomu, identyfikacja i charakteryzację genów, mutacji i analizy polimorfizmu (zwłaszcza RefSeqGene rekordy), badania ekspresji i analizy porównawcze.
Zakres:
NCBI wprowadza RefSeqs dla różnorodnych organizmów, włączając eukariota, bakterie i wirusy. Dodatkowe rekordy są dodawane do kolekcji jako dane dostępne publicznie.
Ogłoszenia:
Dostęp do danych i ich dostępność:
RefSeq jest dostępny za pośrednictwem BLAST, Entrez oraz strony FTP NCBI. Informacje są również dostępne w Entrez Genomes i Entrez Gene, oraz dla niektórych genomów dodawane informacje są dostępne w Map Viewer. Specjalne właściwości zostały zdefiniowane w celu ułatwienia przeszukiwania opartego na Entrez. Zobacz również:
Cechy wyróżniające:
Główne cechy RefSeq zawierają:
- brak redundancji
- wyraźnie powiązane nukleotydowe i białkowe sekwencje
- aktualizacje odzwierciedlają aktualny stan wiedzy o sekwencji danych i biologii
- walidacji danych i format spojności
- odrębne numery dostępu (wszystkie zawierają podkreślenia "_")
- pracownicy NCBI analizują, wykonują rewizję rekordów wskazanych
Referencje:
Proszę odwiedzić http://www.ncbi.nlm.nih.gov/About/tools/index.html%26hl%3Dpl%26lr%3D%26sa%3DG&usg=ALkJrhj9hpYJNUwuIp9N0NMgSiJ-XAmy4g
aby odnaleźć pełną listę artykułów opisujących lub użytych w RefSeq
Bazy STS:
Baza danych sekwencji zidentyfikowanych stron, albo krótkich sekwencji które unikalne w genomie.
dbSTS jest zasobem, który zawiera sekwencje i mapowane dane na krótkich sekwencjach genomu albo Sequence Tagged Sites
STS sekwencje są włączone do GenBanku.
Sekwencje STS mogą być bezpośrednio składane do zgłoszeń GenBanku.
Baza danych STS oferuje alternatywną drogę składania sekwencji STS do GenBanku. Został on specjalnie zaprojektowany dla składania dużych parti sekwencji STS.
Szukaj w STS
Testowe wyszukiwanie zostało przerwane 19 marca 2001.
Raporty informacji UniSTS na temat markerów z GenBanku i innych źródeł.
Dostęp do działów STS w GenBanku może być pobrany używając Entrez.
Dział STS sekwencji nukleotydowych w FASTA formacie jest dostępny przez anonimowy FTP.
UniSTS:
Ujednolicony, niezbędny pokaz (widok) sekwencji na zindentyfikowanych stronach.
UniGene:
Kolekcja ESTs i pełnej długości mRNA sekwencji zorganizowanych w klastry (clusters), każdy reprezentuje unikalną wiedzę albo domniemany ludzki gen z adnotacją do map i informacji ekspresji oraz odsyłacze do innych źródeł.
UniGene: Zorganizowany widok transkryptomów. Każdy UniGene jest umiejscowiony w transkrypcie sekwencji, który pojawia się w tym samym locus transkrypcji, razem z informacją na temat podobieństwa białek, ekspresji genów, cDNA oraz genomowych lokalizacji.
Rzodkiewnik
Map Viewer pokazuje genom dostępny wraz z wpisami w bazie danych Entrez Genome Project.
Aspergillus
Dostarcza informacji o rodzaju Aspergillus (i pokrewnych) z NCBI i ze środowiska naukowego zajmującego się rodzajem Aspergillus.
Bakterie
Graficzne przedstawienie ponad 550 genomów bakterii, które mogą być przeglądane w całości lub badane bardziej szczegółowo, z linkami do danych dotyczących sekwencji.
Pszczoła
Ta strona oferuje brame do zasobów miódu pszczeliego w i poza NCBI.
Chrząszcz
Ta strona oferuje informacje o genomie Tribolium castaneum (trojszyk gryzący) z NCBI środowiska naukowego badającego chrząszcze .
Bizon
Ta strona zawiera informacje o bawołach oraz informacje powiązane z NCBI i środowisk naukowych.
Kot
Ta strona oferuje informacje o genomie Felis catus (kociej) zasobów NCBI i poza nią.
Kurczak
To stanowi źródło informacji na temat Gallus gallus (kurczak) i zasobów związanych pochodzących z NCBI i środowisk naukowych.
Szympans
Ten przewodnik po genomie dostarcza informacji na temat Pan troglodytes z NCBI i spoza.
Krowa
The Bos taurus homepage provides information on bovine and bovine-related resources from NCBI and the research community. The Bos taurus homepage dostarcza informacji na temat bydła bydła i zasobów związanych z NCBI i naukowym.
Dictyostelium
Ta strona będzie bramą jednego kliknięcia łączącą z zasobami związanymi z Dictyostelium na NCBI i ścieżki do dodatkowych zasobów.
Pies
Ta strona oferuje informacje na temat genomu psa z zasobów NCBI i spoza
Żaba
Ta strona dostarczy informacji o Xenopus laevis i Xenopus tropicalis na NCBI i zasobów zgromadzonych przez środowiska naukowe badające Xenopus.
Muszka owocowa
Ta strona zgromadzi informacje o Drosophila zasobów związanych z NCBI i spoza.
Genomy grzybów
Portal do informacji i zasobów na temat grzybów i projekty sekwencjonowania genomów grzybów z NCBI i środowisk naukowych.
Koń
Portal do informacji i zasobów na temat Equus caballus z NCBI i środowisk naukowych.
Człowieka
Serwuje zintegrowane, pojedyncze, informacje o genomie dla biomedycznych naukowców z całego świata, tak aby mogli one korzystać z tych danych w ich wysiłkach badawczych.
Malaria
Zawiera dane i informacje dotyczące genetyki i genomiki malarii.
Moskity
Graficzna reprezentacja całego genomu Anopheles gambiae z linkami do sekwencji danych.
Myszy
Gromadzenie informacji na temat różnorodnych myszy z wielu ośrodków w tym sekwencje, mapowanie, informacje o klonach, jak również wskazówki o odmianach i mutantach.
Nicienie
Dostęp do genomu C. elegans z zasobów NCBI i spoza.
Opos
Ta strona zawiera informacje o oposie i informacje powiazane pochodzące z NCBI i spoza.
Dziobak
Ta strona zawiera informacje o dziobaku i informacje powiązane pochodzące z NCBI i spoza.
Świnia
Dostęp do genomu świni i informacji związanych pochodzących z NCBI i środowisk naukowych.
Genomy Roślinne
Graficzna reprezentacja chromosomów z różnych genomów roślin, które można przeglądać w całości lub bardziej szczegółowo, w detalach z linkami do sekwencji danych.
Królik
Źródło informacji o genomie królika i zasobów związanych pochodzących z NCBI i spoza
Szczur
Źródło informacji na temat szczurów i zasoby związane pochodzące z NCBI i spoza.
Retrowirusy
Zbiór zasobów stworzony specjalnie w celu wspierania wysiłków badawczych retrovirus.
Rhesus maccacus
Ta strona zawiera informacje o małpie rwzus i zasobów powiązanych pochodzących z NCBI i od naukowców spoza.
Jeżowiec
Ta strona główna gromadzi informacje na temat zasobów genomu Strongylocentrotus purpuratus z NCBI i spoza.
Owce
Ta strona oferuje główną bramą do genomu Ovis aries z NCBI i spoza.
Wirusowe Genomy
Graficzne przedstawienie kompletnych wirusowych genomów i linki do sekwencji.
Osa
Ta strona oferuje informacje o osach z NCBI I spoza.
Drożdże
Ta strona skupia informacje związane z Saccharomyces z NCBI i ze środowisk naukowych badających drożdże.
Zeberka prążkogardła
Ta strona zawiera informacje na temat zeberki i zasobów powiązanych pochodzących z NCBI i ze środowisk naukowych.
Danio pręgowany
Ten serwis gromadzi informacje na temat różnorodnych zasobów związanych z Danio z wielu ośrodków, w tym sekwencja, mapowanie, informacje o klonachi, jak również wskazówki o odmianach i mutantach.
Różnorodne dane do pobrania oraz złożone narzędzia mogą zostać znalezione na stronie NCBI, włączając:
Entrez:
Zapewnia zintegrowany dostęp do sekwencji nukleotydów i danych o białkach z ponad 100000 organizmów wraz z trójwymiarowymi strukturami białkowymi, mapowaniem informacji o genomie i PubMed Medline.
LinkOut:
Rejestr usług do tworzenia powiązań z poszczególnych artykułów, czasopism lub danych biologicznych w entrez do zasobów zewnętrznych witryn sieci Web.
My NCBI:
Wyszukuje i zapisuje wyniki oraz funkcję automatycznych aktualizacji oraz przesyła na e-mail wyniki wyszukiwań z zapisanych wyszukiwań.
Citation Matcher:
Pozwala znaleźć PubMed ID albo MEDLINE UID w każdym artykule w bazie danych PubMed, udostępniając jego informacje bibliograficzne.
Wyszukiwanie podobieństw sekwencji
Homepage BLAST
Dostęp do programu BLAST (Basic Local Alignment Search Tool), przeglądu, dokumentacji pomocniczej oraz FAQ.
Blink
Wyświetla wyniki BLAST, które były zrobione dla każdej sekwencji białka w Entrez białkowej bazie danych.
Stand-alone BLAST
Pobieranie BLAST jest dostępne dla każdego. Aby uzyskać więcej informacji na temat jak BLAST pracuje, jego output, oraz jak output i sam program może być manipulowany lub dostosowany, należy obejrzeć BLAST sekcję w NCBI Handbook.
Taksonomia
Przeglądarka taksonomiczna
Narzędzie do przeglądania taksonomicznej bazy danych.
Taksonomia BLAST
Grupy BLAST trafione przez źródła organizmy do ich klasyfikacji w Taksonomicznej Bazie danych NCBI.
TaxPlot
Zapewnia porównanie genomów w trzy sposoby, w oparciu na sekwencjach białkowych.
Kolejność składania
Sekwencja
Narzędzie przekazywania danych, które zawiera ORF Finder, edytor/pokazanie uliniowienia oraz link do PowerBLAST. Zobacz GenBans do złożenia informacji ogólnych.
BankIt
Internetowe narzędzie do składania jednego lub prostych wniosków o sekwencji.
BLAST (Basic Local Alignment Search Tool)
Narzędzie dla porównania sekwencji genów i białek z innymi z publicznych baz danych, jest w kilka rodzajów, w tym PSI-BLAST, PHI-BLAST, a BLAST 2 sekwencji.
CDD Conserved Domain Database
Zestaw alignmentów sekwencji i profilów reprezentujących domeny białek konserwatywnych w ewolucji molekularnej. CD Search Sernice może być wykorzystywany do poszukiwań CDD.
Białka często zawierają kilka modułów lub domen, z których każda posiada odrębne ewolucyjnie pochodzenia i funkcję. Baza danych zachowanych domen (domen konserwatywnych) NCBI jest kolekcją wielokrotnych uliniowień (porównań) konserwatywnych domen i całych białek. Usługa CD-Search może być używana do rozpoznawania konserwatywnych domen obecnych w sekwencji danym białeku.
e-PCR
Może być używany do porównywania zadanej sekwencji do zmapowanych miejsc znaczonych sekwencyjnie w celu znalezienia możliwych map lokalizacji zadanej sekwencji
PCR elektroniczny (e-PCR) jest to procedura obliczeniowa, która służy do określenia sekwencji STS (miejsca znaczone sekwencyjnie – markery służące do zagęszczania map genetycznych oraz poszukiwania sprzężeń z cechami użytkowymi), w sekwencjach DNA. E-PCR szuka potencjalnych STSs w sekwencjach DNA przez szukanie sekwencji, które blisko pasują do primerów reakcji PCR i prawidłowy porządek, orientację i odstępy, które mogłyby stanowić primery reakcji PCR używane do wygenerowania znanych STSów.
Co nowego?
Ulepszona czułość wyszukiwania
Nowa wersja e-PCR wdraża strategię ‘nieostrego’ dopasowania. Aby zmniejszyć prawdopodobieństwo, że prawdziwy STS zostaną pominięte z powodu niedopasowań (nietrafień), wielokrotne daleko położone wyrazy mogą być używane zamiast jednego dokładnego słowa. Każde te słowo ma grupy znaczących pozycji oddzielonych przez pozycje "wildcard", które nie są wymagane, aby je dopasować. Ponadto, możliwe jest również, aby zezwolić na luki w alignmentach primera.
Odwrotne wyszukiwanie
Główną motywacją do wykonania odwrotnego wyszukiwania (zwanego Reverse e-PCR), było zrobienie z tego narzędzia zdolnego do wyszukiwania sekwencji genomu ludzkiego i innych dużych genomów. Nowa wersja e-PCR dostarcza trybu wyszukiwanie używając wybranej sekwencji niekoniecznie z bazy danych.
Geny w ENTREZ
Znajdź informacje o analizie sekwencji dla poszczególnego genu i organizmu.
Gene Expression Omnibus (GEO)
GEO dostarcza kilku narzędzi do pomocy w wizualizacji i eksploracji curated GEO danych.
Gene Expression Omnibus: obfity zbiór sekwencji ulegających ekspresji wspierający zgodność z danymi MIAME i obsługiwane, dostępne online zasoby genów ulegających ekspresji do wyszukiwania.
Wyszukiwarka ORF
Graficzne narzędzie do analizy, które znajduje wszystkie otwarte ramki odczytu w danej sekwencji lub w sekwencji z bazy danych.
Narzędzie identyfikuje wszystkie otwarte ramki odczytu przy użyciu standardowych lub alternatywnych kodów genetycznych. Sekwencji wyprowadzonych aminokwasów zasadowych mogą być zapisane w różnych formatach i wyszukuje z bazy danych za pomocą sekwencji serwera WWW BLAST. Wyszukiwacz ORF powinien być pomocne w przygotowaniu kompletnych i dokładnych propozycji sekwencji To jest również pakowane z the Sequin sequence submission software.
Algorytm wyszukiwania otwartej spektrometrii mas (Open Mass Spectrometry Search Algorithm)
OMSSA pozwala na identyfikację widm peptydów MS/MS poprzez przeszukiwanie bibliotek proteinowych sekwencji.
Znajdź widma MS/MS z jonowych pułapek spektrometrów.
Znajdź widma ETD MS/MS
[OMSSA] jest efektywną wyszukiwarką do identyfikacji widm MS/MS peptydów przez przeszukiwanie bibliotek znanych sekwencji proteinowych.. Wyniki OMSSA są trafne z prawdopodobieństwem obliczonym używając classical hypothesis testing, ta sama metoda statystyczna jest użyta w BLAST. OMSSA jest domeną darmową i upublicznioną.
Aby skorzystać z naszej usługi wyszukiwania, wybierz jedną z powyższych linków. Możesz także pobrać http://pubchem.ncbi.nlm.nih.gov/omssa/download.htm OMSSA i używać go jako wiersz polecenia wykonywalnego na własnym komputerze.
OMSSA jest opisany i zatwierdzony w publikacji http://www.ncbi.nlm.nih.gov/pubmed/15473683?dopt=Abstract . Aktualna statystyka wyszukiwań jest opisana na posterze. Źródło kodów OMSSA jest w publicznej domenie (proszę zajrzeć do FAQ aby zobaczyć szczegóły ściągania i budowy).
NCBI utrzymuje listę emaili dotyczących ogłoszeń i generalnej dyskusji o OMSSA. Dopisz się do listy jeśli chcesz pisać. Archiwum listy emaili jest ciągle dostępne online.
Ściżka archiwalna
Dostarczone do zasobów surowych sekwencji zasadnicze sekwencje generowanei przez różne projekty genomowe.
VecScreen
Narzędzie do identyfikowanie segmentów kwasów nukleinowych które mogą być wektorami, linkerami lub adapter origin przed użyciem Tools dla Sequence Analysis lub propozycji.
O VecScreen
VecScreen jest system szybkiej identyfikacji segmentów z sekwencji kwasu nukleinowego, które mogą być wektorowego pochodzenia. NCBI rozwiniął VecScreen w celu rozwiązania problemu wektorowych skażeń sekwencji w publicznych baz danych. Ta strona sieci Web ma pomóc naukowcom zidentyfikować i usunąć wszystkie segmenty pochodzące od wektora przed analizą sekwencji.
Nierozpoznanie obcych segmentów może:
* prowadzić do błędnych wniosków na temat biologicznego znaczenia sekwencji
* sequence spowodować stratę czasu i wysiłku na analizie sekwencji zanieczyszczonej
* opóźnić opublikowanie sekwencji w publicznej bazie danych
* zanieczyścić publiczne bazy danych zanieczyszczoną sekwencją
Naukowcy są zachęcani do pokazywania zanieczyszczających sekwencji wektora przy użyciu formy: Accession, GI, lub FASTA.
Narzędzia do celów porównawczych i analizy trójwymiarowych struktur makromolekularnych
Wyszukiwarka CD
Wyszukiwarka konserwatywnych domen (CD-search) może być użyta do indentyfikacji konserwatywnych domen w sekwencji białek.
Białka często składają się z kliku modułów albo domen, każde z różnego ewolucyjnie regionów i funkcji. Jest to baza danych różnorodnych sekwencji uliniowienia dla starych domen i pełnej długości białek. Może być on użyty do identyfikacji konserwatywnych domen w białkowej sekwencji.
Cn3D
Ukazywanie trójwymiarowych struktur i uliniowienia sekwencji dla struktur baz danych NCBI.
Cn3D jest pomocnikiem dla wyszukiwarki internetowej, który pozwala na wyświetlenie widoku 3D struktur z NCBI Entrez usług pobierania. Działa w systemie Windows, Macintosh i Unix. Cn3D jednocześnie wyświetla strukturę, sekwencję i dostosowanie, a teraz ma dużo adnotacji i dostosowanie funkcji edycyjnych.
Poniżej jest stosunkowo prosta próbka tego, co możemy zrobić Cn3D. Istnieje wiele więcej przykładów w Tutorial, wraz z instrukcjami, aby pomóc nowemu użytkownikowi zacząć.
Ten przykład jest kuratorem CD z domeny WD40, która jest wielofunkcjonalnym 7-krotną beta helisą. Cn3D wyświetla reprezentacyjne struktury białek, rodziny dopasowania oraz panele adnotacji z informacjami o funkcjach adnotacji rodziny białek. Wyróżnienie w obu strukturach i sekwencjach są zachowywane pozostałości w strukturze charakterystyczne dla tej dziedziny.
Nowe funkcje Cn3D 4.1:
- ulepszone algorytmy dopasowania sekwencji
- automatyczne dostosowanie importu
- wiedza taksonomiczna
Nowe funcke Cn3D 4.0:
- od nowa napisana w C++, aby poprawić wydajność
- ulepszone renderowanie OpenGL prędkości
- nowy interfejs użytkownika z wykorzystaniem wxWindows
- zakończeny system dostosowania, wraz z algorytmem na budowę nowych dostosowań
- rozległe struktury i dostosowania adnotowanych cech
- szczegółowy styl kontroli
- wysokiej rozdzielczości zdjęcia eksportu
- wbudowany system pomocy
CDART
Wyświetla funkcjonalne domeny, które tworzą białka i listę białek z podobieństwem architektury domen.
Wyszukiwarka VAST
Wyszukiwarka podobieństw struktura-struktura. Porównuje trójwymiarowe współrzędne nowo ustalonych struktur białka z tymi w MMDB/PDB bazy danych.
Wyszukiwarka VAST oblicza strukturę sąsiadów, ktore można interaktywnie wyszukać, przeglądać superpozycje i dopasowania dzięki modelowi molekularnemu. Z tej strony, można albo startować nowym wyszukaniem albo odzyskać wyniki wyszukiwania VAST z poprzenich prac.
Aby rozpocząc nowe wyszukiwanie, proszę umieścić swoje dane w pliku tekstowym używając formatu PDB. Wpisać nazwę w poniższej lini, wybrać opcje wyszukiwania oraz kliknąć "Submit". "Medium redundancy Subset of PDB" jest około 1/10 rozmiarem "All of PDB" pozwala to na szybsze wyszukiwanie.
Aby uzyskać rezultaty poprzedniej pracy, należy wpisać "request identifier" numer który otrzymaliśmy podczas poprzedniego wyszukiwania, w poniższej lini i kliknąć "pokaż"
Threading
Algorytm do rozpoznawania podobieństw białek.
Pracujemy nad metodami potwierdzającymi homologię przez porównywanie przypuszczalnych struktur sekwencji. Jeśli strukturalne podobieństwo między próbą a celem jest wystarczająco duże, ich związek może być wykryty nawet w braku statystycznie istotnego podobieństwa sekwencji.Badane tematu obejmują:
- Połączenia punktacji opartej na kontakcie systemów i matrycy punktacji specyficznych pozycji (PSSMs)
- Znaczenia bazy danych Threading z wynikami wyszukiwania
- Opracowanie przyjaznych dla użytkownika narzędzi do przeglądania wyników Threading
- Strukturalne szablony oparte na modelach zmian strukturalnych
- Zakres stukturalnych podobieństw wykrytych przez Threading.
Dostęp do różnych map genetycznych i fizycznych
Przegląd map
Dostarcza zintegrowanych z chromosomem map dla 40 organizmów w tym kręgowców, bezkręgowców, pierwotniaków, roślin i grzybów. Wyświetla jedną lub więcej map, które zostały dostosowane do siebie w oparciu o podzielony marker i nazwy genów oraz dla map sekwencyjnych w oparciu o skoordynowany system wspólnych sekwencji.
Mapa człowieka
Zapoznaj się z ludzkim genomem z ‘Przeglądem map’ Homo sapiens serwisu NCBI, który pokazuje właściwości ensibijajowskiego zestawu danych dotyczących ludzkiego genomu tak jak cytogenetyczne, genetyczne, fizyczne i radiacyjne hybrydowe mapy.
Model markera
Pozwala użytkownikowi na skonstruowanie sekwencji mRNA z danych genomowych, wybór egzonów zidentyfikowanych przez zestawienie cząsteczek mRNA i EST, montowanie modeli, testowanie otwartych ramek odczytu i zapisywanie wlasnych rezultatów. Model Marker jest dostępny z ‘przeglądu map’ wszędzie gdziekolwiek jest link ‘mm’.
Dokument pomocniczy Model Marker
Model Marker pozwala ci na pokazanie dowodów które były użyte do budowy modelu genu na zgromadzonych sekwencjach genomowych i utworzyć własną wersję modelu przez wybór egzonów. Aby zobaczyć przykład śledź link ‘mm’ obok jakiegokolwiek genu przypisane do ludzkiej mapy sekwencji genów: http://www.ncbi.nlm.nih.gov/mapview/maps.cgi?taxid=9606&chr=1&maps=ideogr,loc&verbose=on
Model Maker jest dostępny z map sekwencji, które były analizowane przez NCBI I pokazane w ‘przeglądzie map’ (Map Viewer) http://www.ncbi.nlm.nih.gov/mapview/ . Tylko niektóre organizmy reprezentowane Map Viewer maja takie sekwencyjne mapy a wiec linki Model Maker. Dokument pomocniczy zawiera linki do plików z danymi i poradami dotyczącymi wyszukiwania gatunków. Dokumenty te opisują mapy dostępne dla każdego organizmu.
Mapa genowa OMIM
Cytogenetyczne lokalizacje genów, które zostały zgłoszone w literaturze i ustalone w oparciu o różne metody mapowania
Przedstawia lokalizację genów chorób i innych genów ulegających ekspresji opisanych w OMIM. Zobacz listę tych genów: http://www.ncbi.nlm.nih.gov/Omim/getmorbid.cgi . Aby znaleźć bardziej wyrafinowane mapy genów i segmentów DNA kliknij Location aby przywołać NCBI Entrez Map Viewer.
· Wprowadź symbol genów, lokalizację chromosomów, zaburzenia do wyszukiwania, np. "CYP1", "5", "1pter", "Xq" lub "choroba".
· Musisz wykapitalikować X i Y, aby szukać tych chromosomów.
Mapa chorób OMIM
Alfabetyczny spis chorób i odpowiadająca im lokalizacja na mapie cytogenetycznej, z linkami do wpisów w OMIM
OMIM Morbid Map prezentuje cytogenetyczne mapy lokalizacji genów chorób opisanych w OMIM. Zobacz: http://www.ncbi.nlm.nih.gov/Omim/getmap.cgi . Aby znaleźć bardziej wyrafinowane mapy genów i segmentów DNA użyj NCBI Entrez Map Viewer http://www.ncbi.nlm.nih.gov/mapview/map_search.cgi?taxid=9606 i Genome Database.
NCBI współpracuje z Narodowym Instytutem Raka (NCI) w szeregu projektów badawczych, zaliczając:
Rak chromosomów:
Trzy bazy danych, NCI/NCBI, SKY/M-FISH i CGH, w NCI Mitelman bazy danych o chromosomowych abberacjach w raku, oraz NCI Powtarzające się abberracje w raku, są teraz zintegrowane w systemie NCBI Entrez jako Chromosomy Raka.
Wyszukiwanie cytogenetyki, klinicznych i/lub polecanych informacji. Kwarendy są gotowe do użycia tego samego wejścia jak dla innych baz danych Entrez jak PubMed lub Nukleotydowej. Wyszukiwanie odbywa się przy użyciu trzech metod: Entrez Query Box, Proste wyszukiwanie albo Zaawansowane.
Obejrzyj publikacje na temat tej bazy danych: http://www.ncbi.nlm.nih.gov/portal/query.fcgi?db=pubmed&cmd=Retrieve&dopt=Abstract&list_uids=15934046
GEO
Ekspresja Genu Omnibus jest wysokiej przepustowości ekspresją genu/ składowiskowo liczebnych molekularnych danych, jak również źródłem dla wyszukiwania, pytania danych o ekspresji genów.
Anatomia Projektu na temat Raka Genomu (CGAP)
Interdyscyplinarny program do identyfikacji ekspresji genów w różnych stadiach raka.
The Anatomy Cancer Genome Project (CGAP) jest to interdyscyplinarny program ustanowiony i zarządzany przez National Cancer Institute (NCI) w celu wygenerowania informacji i narzędzi technologicznych niezbędnych do rozszyfrowania cząsteczkowej anatomii komórek nowotworowych.
Od powstania CGAP w 1996 r., NCBI był kluczowym uczestnikiem bioinformatyki, umożliwiał śledzenie danych, archiwizacji danych oraz udostępniał narzędzia analityczne do opracowanie i wdrożenia danych. Rola NCBI's jest stale ewoluuje w związku z potrzebami zmieniających się i rozszerzających źródeł danych, jak również na potrzeby środowisk naukowych.
W poniższych informacjach NCBI podkreśla wkład do CGAP.
NCBI ściśle współpracuje z NCI jak CGAP przygotowuje duża liczbę znaczników ekspresji sekwencji (EST), z których wszystkie są zdeponowane w dbEST, oraz następnie włączane do UniGene i HomoloGene. Oparte na genie, ręczne adnotacje mogą być również właczane do Feelback for RefSeq oraz Entrez Gene.
NCBI stworzył pierwszą publiczną stronę CGAP i zaprojektowal wszystkie analityczne narzędzia dla CGAP Tumor Gene Index podczas pierwszych czterech lat trwania projektu. Ponadto NCBI oferuje narzędzia analityczne do wnioskowania informacji o ekspresji genów z szerokiej bazy bibliotek EST. Profile ekspresji genów są powiązane z wieloma zespołami Unigene; ponad to istnieją narzędzia Digital Differential, porównujące profile komputerowej ekspresji genów miedzy wybranymi bibliotekami cDNA. Na stronie CGAP istnieje wiele podobnych narzędzi w ramach tkanek.
Seria analiz ekspresji Genu (SAGE) jest bardziej ekonomiczną metodą produkcji danych o ekspresji genów (porównanych z danymi EST). CGAP wspiera produkcję bibliotek SAGE i ich sekwencjonowanie podczas gdy NCBI archiwuje biblioteki SAGE w Gene Ekspression Omnibus (GEO). Dane są wyszukiwane za pomocą entrez GEO.
NcBI zaprojektowało i kontynuuje utrzymanie wewnętrznych baz danych, które śledzą próbki CGAP, biblioteki oraz klony użyte w generacji EST i danych SAGE.
Bogactwo danych produkowanych przez CGAP doprowadziło NCBI oraz pracowników NCI do rozwijania ad hoc systemu klasyfikacji hierarchicznie powiązanych słów kluczowych. OBa NCBI oraz NCI były odpowiedzialne za klasyfikację wszystkich nowych bibliotek EST i SAGE ludzi i myszy.
NCBI blisko współpracuje z komponentem CGAP nazwanym Projekt Rakowych abberacji chromosomu (CCAP). Wykorzystanie fluorescencyjnej hybrydyzacji in situ, CCAP generuje klony, które są rozmieszczone w przestrzeni 1-2 Mb całej długości ludzkiego genomu. Raz zmapowany, gotowe klony sekwencji DNA BAC są udostępniane dla osób wyszukujących. Udział NCBI polega na określeniu kandydata klonów BAC by być zmapowanym FISHem, zarchiwizować wyniki oraz zlokalizować te klony w projekcie sekwencjonowania kontigów. Dane te mogą być pokazane w NCBI Map Viewer, są one związane z NCBI Clone Registry oraz stronami UniSTS oraz mogą dodatkowo być wyświetlane w NCI CGAP Chromosome oraz NCBI Human BAC Resource.
CCAP charakteryzuje abberacje chromosomowe w wybranych typach nowotworów za pomocą spektral karyotyping (SKY) oraz porównawczych hybrydyzacji genomu (CGH). Ułatwienia identyfikacji aberracji chromosomalnych SKY oraz CGH mogą być użyte do wygenerowania mapy liczby kopii DNA w genomach guza. Baza danych SKY/CGH była zaprojektowana do przechowywania danych i publicznego dostępu.
Dr. Mitelman, Mertens i Johansson systematycznie sumowali powtarzające neoplasia abberacje chromosomowe związane z Mitelman Database of Chromosome Aberrations in Cancer. Praca ta, która pierwotnie pojawiła się w kwietniu 1997 Special Issue of Nature Genetics, zatytuowana: "A breakpoint map of recurrent chromosomal rearrangements in human neoplasia", jest stale aktualizowany i podsumowanie późniejszych danych jest pokazane w NCBI Map Viewer. O wszystkie powtarzające się aberracje można interaktywnie zapytać na NCI CGAP stronie w Recurrent Chromosome Abberrations in Cancer.
The Cancer Chromosome database intergruje SKY/M-FISH i CGH database z Mitelman Database of Chromosome Aberrations in Cancer oraz the Recurrent Chromosome Aberrations in Cancer database. Te trzy bazy mogą być przeszukiwane korzystając z Entrez wyszukiwania oraz systemu wyszukiwania dla aberracji chromosomów, danych klinicznych oraz cytowanych opini. Wspólne diagnozy, anatomiczne strony, chromosomowe breakpoint, skrzyżowania, numeryczne i strukturalne nieprawidłowości oraz zespoły zdobytych i straconych wśród wybranych przypadków mogą być porównywnane przez korzystanie z raportu "podobieństwa". Ponieważ model stosowany do CGH jest podzbiorem danych kariotypu, możliwe jest teraz zbadanie podobieństw pomiędzy wynikami CGH i kariotypami bezpośrednio.
Różne środki mogą być pobierane za pośrednictwem naszej witryny FTP.
Bazy danych:
GenBank
Można pobrać najczęściej odwiedzane w pełnym udostępnieniu i/lub dziennie skumulowane, nieskumulowane pliki w formacie, ASN.1 format albo FASTA format.
RefSeq:
Baza danych niezmiennie odniesiona do standardów sekwencji, wliczając genomowe DNA, mRNA oraz białka dla znanych genów. Wiele współpracy, zarówno w NCBI jak i zewnętrznych grupach, wsparcie naszych wysiłków zbierania danych.
Entrez Gene:
Zapewnia oparty na genie widok kluczowych genomów oraz dostarcza kluczowych powiązań w mapach, sekwencjach, expresji, strukturze, funkcji, cytowaniu oraz homologii danych. Jest ona następcą LocusLink.
dbSNP:
Baza danych pojedyńczego nukleotydowego polimorfizmu, drobnych insercji/delecji, polimorficznych powtarzalnych sekwencji oraz odmian mikrosatelitów.
Human Genome Data:
Ten katalog zawiera jeden folder dla każdego chromosomu, który zawiera genomowe powiązania budowy z dokończonych i niedokończonych danych o sekwencjach. Są one dostępne w różnych formatach, włączając ASN.1 format, FASTA format, GenBank flat file i GenBank summary format.
Inne genomy:
Dane dla wielu genomów, włączając widok map danych, które mogą zostać ściągnięte (download)
Rezpozytorium baz danych:
UniGene, GeneMap, dbEST, dbGSS, dbSTS, OMIM, oraz liczbę zewnętrznie utrzymywanych specjalistycznych baz danych.
Taksonomia:
Umożliwia pobieranie danych taksonomii dla różnych organizmów.
Oprogramowanie:
BLAST:
Umożliwia pobranie oprogramowania dla porównania sekwencji.
Cn3D:
Oprogramowanie do pokazywania struktur w trzech wymiarach.
Oprogramowanie klient/serwer:
Sequin:
Złożone oprogramowanie programu dla jednego lub wielu propozycji, długich sekwencji, całych genomów, uliniowienia, studiów populacji/filogenezy/mutacji.
Klient sieci BLAST:
Klient BLAST (blastcl3), których ma dostęp do wyszukiwarki NCBI BLAST. Blastcl3 może wyszukać wszystkich sekwencji w pliku FASTA i produkować od jednego do wielu uliniowień w tekscie albo w formacie HTML. Może również przeszukiwać wiele baz danych.
Oprogramowanie narzędzi NCBI:
Zestaw oprogramowania i wymiany danych specyficznie użytych w NCBI do produkcji przenośnego, modułowego oprogramowania dla biologii molekularnej.
Rozrastanie się zasobów GenBanku - statystyka
Na dzień dzisiejszy w GenBanku jest zdeponowanych ponad 80mln sekwencji
W tabeli i na wykresie przedstawione dane z lat 1982-2005 – liczba sekwencji i liczba par zasad DNA
Statystyka GEO (Gene Expression Omnibus)
Najnowsze statystyki bazy danych GEO, czyli bazy danych zawierającej informacje dotyczące ekspresji genów, wspomagającej zgodność z MIME http://www.ncbi.nlm.nih.gov/geo/info/MIAME.html
Wiele czasopism i agencji finansujących wymaga danych o mikromacierzach zgodnych z MIME (minimum informacji które musza być dostarczone aby opisać eksperyment mikromacierzy). GEO zawiera procedury zgodne z MIME.
Sześć najważniejszych elementów zasilających MIAME:
- Surowe dane dla każdego hybrydyzacji (np. CEL lub pliki GPR)
- Poddane ostatecznej obróbce (normalizowane) dane dotyczące zestawu hybrydyzacji w eksperymencie (badaniu) (np. matryca ekspresji genów wykorzystana do wyciągnięcia wniosków z badania)
- Zasadnicze adnotacje zawierające parametry doświadczenia i ich wartości (np. związek i jego dawka w określonej reakcji)
- Projekt eksperymentu zawierający wzory wzajemnych relacji między danymi (np. który plik danych odnieść do której próbki, jakie rodzaje hybrydyzacji są techniczne, a które są biologicznymi replikami [naśladują naturalne procesy?])
- Odpowiednie (wystarczające) adnotacje na matrycy (np. geny identyfikatory, współrzędne genomu, sekwencje sond oligonukleotydowych lub odniesienia do katalogów komercyjnych matryc)
- Podstawowe warunki laboratoryjne i dane i protokołów (np. jaka standaryzacja metody została użyta w celu uzyskania ostatecznych przetworzonych danych)
W GEO posiada się dostęp do opublikowanych danych dotyczących genow ulegajacych ekspresji, które są pogrupowane w platformy, próby i serie. Potrzebnych danych można też szukać poprzez nazwiska autorów.
Wszystkie kontakty
Wszystkie platformy:
Wszystkie próby
Wszystkie serie
Postęp w sekwencjonowaniu ludzkiego genomu
Fragmenty (frakcje) genomu ludzkiego – ukończone i projektowane sekwencje
Wirus grypy – zasoby
Postępy w senwenjonowaaniu genomu wirusa grypy w GenBanku
Zawiera dane z NIAID National Institute of Allergy and Infectious Diseases jak I z GenBanku
Oraz narzedzia do analizy sekwencji wirusa grypy i adnotacjami. Są tu też linki do innych zasobów, które zawierają sekwencje wirusa grypy, publikacje i najważniejsze informacje o tym wirusie.
Na grafach przedstawione są postępy w sekwencjonowaniu
Statystyka dotycząca PudMed
Przedstawione tu są najnowsze statystyki wyszukiwania
Statystyka dotycząca RefSeq
Statystyka taksonomiczna
Przedstawienie statystyk dla poszczególnych węzłów taksonomicznych
Są tu do wyboru: Archea, Bakterie, Eukariota, Grzyby, Wielokomórkowce, Wyższe rośliny zielone, Wirusy a wśród nich: wyższe taksony, rodzaje, gatunki, niższe taksony.
Można wykluczyć: niesklasyfikowane, niewykształcone, niesprecyzowane
Używa się formatu: YYY/MM/DD (miesiąc i rok są opcjonalne)
Wyniki mogą być wyświetlane z dnia na dzień (również tydzień, miesiąc, rok) w wybranym zasięgu – ale tylko wtedy, gdy zawiera cały szereg tygodni lub miesięcy i nie ma więcej niż 20dni, 10tygodni, 23miesiące lub 20lat.
Ścieżka archiwalnych statystyk
Statystyki śledzenia zbiorów
Graficzny widok archiwów statystyk