Przez ostatnich kilka dekad rozwój biologii molekularnej i genomiki doprowadził do szybkiego wzrostu informacji biologicznej stworzoną przez naukowców. Duży napływ informacji wymusił potrzebę utworzenia komputerowych baz danych wraz z wyspecjalizowanymi narzędziami umożliwiających przeglądanie i analizę tych danych.
W ramce: W czerwcu 2000 roku zostały zakończone prace nad poznaniem ludzkiego genomu. 15 lutego 2001 roku pojawił się w Nature pierwszy artykuł na ten temat.
Co to jest biologiczna baza danych?
Baza danych jest złożona z różnych elementów. Najważniejszymi z nich jest rekord podzielony na kilka pól, w których są przechowywane informacje poszczególnych kategorii. Na przykład w książce adresowej każdy rekord to zbiór informacji na temat jednej osoby. Składa się on z kilku pól przechowujących takie informacje, jak: nazwę, opis cząsteczki, nazwę organizmu, z którego została ona wyizolowana i dane literaturowe na temat danej sekwencji W każdym polu zapisywane są dane oddzielonej kategorii. Aby naukowcy mogli skorzystać z informacji przechowywanych w bazie danych muszą być spełnione dwa dodatkowe wymogi:
- łatwy dostęp do informacji;
- segregacja informacji, tak aby osoba zainteresowana uzyskała konkretną odpowiedź na pytanie.
W NCBI wiele baz danych są ze sobą powiązane poprzez unikalny system wyszukiwania i pobierania, zwany Entrez.
Entrez pozwala użytkownikowi nie tylko na dostęp do informacji z pojedynczej bazy danych, ale z wielu. Na przykład, białkowa baza danych jest połączona z taksonomiczną. To pozwala na wyszukanie gatunku/organizmu, z którego pochodzi interesujące nas białko.
Dane w GenBank są udostępniane na wiele różnych sposobów, każda dostosowana do szczególnych zastosowań, takich jak data złożenia lub sekwencji wyszukiwania.
Bioinformatyka to dyscyplina zajmująca się stosowaniem narzędzi matematycznych i informatycznych do rozwiązywania problemów z nauk biologicznych. Z bioinformatyką powiązane są: genomika, proteomika, metabolomika i transkryptomika. Ostatecznym celem tej dziedziny jest umożliwienie odkrycia nowych biologicznych spostrzeżeń, jak również kreowanie globalnego punktu widzenia, będącego ujednoliceniem zasad w biologii. Na początku „rewolucji genomowej”, zadaniem bioinformatyki było stworzenie i utrzymanie bazy danych do przechowywania informacji genetycznych, dotyczących np.: nukleotydów i sekwencji aminokwasowych. Rozwój tego typu bazy danych umożliwia naukowcom korzystanie zarówno z istniejących już danych, jak również przedstawianie nowych lub zmienionych danych.
Jakkolwiek, wszystkie te informacje muszą być łączone, tworząc pełniejszy obraz normalnej działalności komórkowej, dzięki czemu naukowcy mogą analizować, w jaki sposób zachodzą zmiany w kolejnych stadiach danej choroby. W związku z powyższym zakres bioinformatyki ewoluował w taki sposób, że obecnie największy nacisk kładzie się na analizę i interpretację różnych typów danych, dotyczących nukleotydów, sekwencji aminokwasowych domen białkowych i struktur białkowych. Rzeczywisty proces analizowania i interpretowania danych jest określany jako computational biology (biologia obliczeniowa). Bioinformatyka i computational biology obejmują:
- opracowanie i wdrożenie narzędzi, które umożliwiają wykorzystanie i zarządzanie różnymi rodzajami informacji;
- rozwój nowych algorytmów i statystyk do oceny relacji między członami dużych zestawów danych, takich jak: metody służące do zlokalizowania sekwencji genu, przewidywania struktury białka i jego funkcji oraz wyszukiwania sekwencji wspólnych dla danej rodziny białek.
Uzasadnienie dla stosowania metod obliczeniowych, w celu ułatwienia zrozumienia różnych procesów biologicznych, obejmuje:
- szerszą perspektywę w przeprowadzanych doświadczeniach;
- zdolność do kapitalizowania wschodzących technologii database-mining – proces, w którym są generowane hipotezy dotyczące funkcji lub struktury geny, bądź białka poprze określenie podobnych sekwencji u lepiej scharakteryzowanych organizmów.
Nowe spojrzenia na chorobę na poziomie molekularnym może pochodzić z badań funkcji homologów chorób genetycznych przeprowadzonych na organizmach modelowych. W tym przypadku homologia odnosi się do dwóch genów wspólnej historii ewolucyjnej. Naukowcy używają również terminów homologia lub homologiczny bez względu na ewolucyjny związek. Równie ekscytująca jest możliwość odkrywania wzorców i ewolucyjnych związków między różnymi formami życia. Za pomocą sekwencji nukleotydów i białek powinno być możliwe znalezienie wspólnych przodków . Jak dotąd, doświadczenie uczy nas, że blisko spokrewnione organizmy mają podobne sekwencje a daleko spokrewnione różnią się większą liczbą sekwencji. Naukowcy są w stanie zrekonstruować ewolucyjne relacje pomiędzy dwoma gatunkami, oszacować czas rozbieżności między dwoma organizmami.
W ramce|: Filogenetyka jest dziedziną biologii, która zajmuje się identyfikacją i zrozumieniem relacji pomiędzy różnymi rodzajami życia na ziemi.
Proces ewolucji doprowadził do produkcji sekwencji DNA, które kodują białka o specyficznych funkcjach. W przypadku braku struktury białka, które zostało określone przy pomocy krystalografii rentgenowskiej lub rezonansu magnetycznego (NMR), mogą starać się przewidzieć trójwymiarową strukturę używając białkowego lub molekularnego modelowania. Ta metoda wykorzystuje doświadczalnie ustalone szablony struktur białkowych do przewidywania struktur innych białek, które mają podobną sekwencję aminokwasów.
Cztery kroki modelowania.
Mapy genomu służą jako szablony do orientowania informacji o sekwencji. Kilka lat temu naukowiec chcący zlokalizować gen lub sekwencję nukleotydów, był zmuszony do ręcznego mapowania genomu w interesującym go regionie, co jest czasochłonnym oraz żmudnym procesem. Obecnie, dzięki nowym technologiom i napływowi sekwencji danych, dostępne są wysokiej jakości mapy całego genomu, z których może korzystać cała społeczność naukowa w swoich badaniach.
Skomputeryzowane mapy, sprawiają że poszukiwania są szybsze, tańsze oraz bardziej praktyczne dla każdego z naukowców. W skrócie, naukowcy najpierw używają mapy genetycznej, aby przypisać genowi miejsce na stosunkowo małym obszarze w chromosomie. Następnie używają oni map fizycznych aby badają najbliższy region, aby określić dokładną lokację genu. W świetle tego postępu, kłopot dla naukowca został przesunięty z mapowania genomu lub regionu zainteresowań genetycznych, do przeglądania dużej liczby witryn WWW oraz baz danych.
Map Viewer: narzędzie do wizualizacji całych genomów lub pojedynczych chromosomów.
NCBI map Viewer jest narzędziem, które pozwala użytkownikowi wyświetlić pełen genom organizmu, zintegrowane mapy każdego z chromosomów (jeśli dostępne), i/lub sekwencję dla regionu zainteresowań. Korzystając z Map Viewer naukowiec ma do wyboru opcję „Pokaż cały genom” lub „Chromosom lub Map View”. Pokaż genom wyświetla schemat dla wszystkich chromosomów organizmu, podczas gdy Map View pokazuje jedną lub więcej szczegółowych map dla pojedynczych chromosomów. Jeśli istnieje więcej niż jedna mapa dla chromosomu, Map Viewer umożliwia wyświetlenie tych map jednocześnie.
Korzystając z Map Viewer naukowcy mogą znaleźć odpowiedzi na takie pytania jak:
Szybko powstająca dziedzina bioinformatyki obiecuje doprowadzić do postępu w zrozumieniu podstawowych biologicznych procesów, a także postęp w diagnostyce, leczeniu i zapobieganiu wielu chorobom genetycznym. Bioinformatyka zmieniła dyscypliny biologii z czysto opartych na badaniach laboratoryjnych nauk do nauk informatycznych również. Coraz częściej, badania biologiczne zaczynają się od przeszukania dużej liczby baz danych oraz stron internetowych w celu sformułowania specyficznej hipotezy lub zaprojektowania eksperymentów na wielką skalę. Konsekwencje tych zmian, zarówno w nauce jak i medycynie, są zdumiewające.
Mapowanie genomu: Genetyczny przewodnik po „autostradzie” ludzkiego genomu.
Wyobraź sobie, że jesteś w samochodzie i jedziesz odwiedzić starego przyjaciela, który właśnie przeniósł się do Los Angeles. W radiu leci Twój ulubiony kawałek a Ty nie wiesz gdzie masz jechać. Zatrzymujesz się, sprawdzasz mapę i uświadamiasz sobie, że znajdują się na niej tylko międzystanowe autostrady, brak zaś pojedynczych, małych ulic. Czy kiedykolwiek znajdziesz dom Twojego przyjaciela?
Ten scenariusz jest podobny do sytuacji przed jaką stoją naukowcy szukając konkretnego genu w ogromnym ludzkim genomie. Mają oni dostęp do genetycznej i fizycznej mapy genomu, które dostarczają informacji o kolejności poszczególnych elementów wzdłuż chromosomu. Jednakże mapy genetyczne są jak międzystanowe autostrady, pozwalają bowiem na oszacowanie odległości między dwiema pozycjami. Można powiedzieć, że mapy genetyczne SA naukowym przewodnikiem po genomie, tak jak mapa drogowa wskazująca kierowcy kierunek z miasta do miasta. Mapy fizyczne pozwalają oszacować rzeczywistą odległość. Aby kontynuować naszą analogię mapy fizyczne można byłoby przyrównać do mapy ulicy, gdzie odległość między dwoma placówkami może być dokładnie określona. Mapy fizyczne zatem ułatwiają naukowcom lokalizację genu.
Rodzaje obiektów znalezionych w mapie genetycznej.
Podobnie jak w mapach międzypaństwowych miasta i miasteczka służą jako punkty orientacyjne, tak w mapach genetycznych punktami tymi są markery genetyczne. Termin markera jest wykorzystywany do opisywania jakichkolwiek obserwowanych zmian, mutacji w pojedynczym locus. Może być użyty jako jeden punkt orientacyjny na mapie jeśli w większości przypadków ten wycinek DNA jest dziedziczony z pokolenia na pokolenie, według standardowych zasad. Markerem może być gen kodujący zauważalne cechy fizyczne, takie jak: kolor oczu lub niezauważalne, takie jak choroba. DNA- based może służyć również jako marker. Te typy znaczników znajdują się w niekodowanych regionach genów i są używane do wykrywania unikalnych regionów w chromosomie. Markery DNA są szczególnie przydatne do generowania map genetycznych, gdy nie zachodzą sporadyczne, przewidywalne mutacje, które występują podczas mejozy – formowanie komórek jajowych i spermy- co prowadzi do wysokiego stopnia zmienności w zawartości DNA przekazywanego z jednego pokolenia na drugie.
Powszechnie stosowane markery DNA.
RFLP – polimorfiz długości fragmentów restrykcyjnych, był jedną z pierwszych opracowanych narzędzi w rozpoznawaniu DNA. Tą techniką wykrywa się różnice pomiędzy rozmiarami fragmentów DNA pociętych enzymami restrykcyjnymi. Enzymy te tną DNA w miejscach zawierających odpowiednią dla nich sekwencję DNA.
VNTR – zmienna liczba tandemowych powtórzeń. Technika ta opiera się na analizie tandemowo powtarzających się sekwencjach par zasad o długości kilkunastu do kilkudziesięciu nukleotydów.
Polimorfizm sekwencji mikrosatelitarnych – definiowane są przez zminę liczby powtrzórzeń bardzo małej liczby par zasad ułożonych kolejno na chromosomie. Często powtórzenia te a nukleotydów lub zasad: cytozyny i adenozyny. Liczba powtórzeń dla danego mikrosatelitarnego DNA mogą się różnić między poszczególnymi osobnikami, stąd termin polimorfim – istnienie kilku form w populacji.
SNP – polimorfizm pojedynczego nukleotydu, polega na zmianie pojedynczego nukleotydu pomiędzy osobnikami danego gatunk, nie zmieniające długości całkowitej sekwencji DNA w danym regionie.
Od analizy połączeń do mapowania genetycznego.
Pierwsi genetycy uznawali, że geny zlokalizowane są na chromosomach i wierzyli, że poszczególne chromosomy były dziedziczone jako jednostka nienaruszalna. Wysunęli oni hipotezę, że jeśli dwa geny zostały zlokalizowane na tym samym chromosomie to są one fizycznie ze sobą powiązane i dziedziczone razem. Teraz już wiemy, że nie zawsze tak się dzieje. Badania z roku 1910 wykazały, że bardzo niewiele par genów wykazuje pełne powiązanie. Pary genów były dziedziczone albo samodzielnie albo wykazywały częściowe powiązanie.
Podczas procesu mejozy, w którym gamety (plemniki i komórki jajowe), produkują dwie kopie każdego z pary chromosomów. Ramiona chromosomów mogą ulec przerwaniu i następuje wówczas wymiana ich fragmentów, proces ten nazywamy rekombinacją lub crossing – over. Jeżeli zachodzi rekombinacja to każdy chromosom znajdujący się w gametach składa się z „mieszanki” chromosomów homologicznych. Zatem rekombinacja ma bezpośredni wpływ na dziedziczenie genów zaangażowanych. Ponieważ nie można fizycznie zobaczyć procesu crossing – over, trudno ustalić ile razy zjawisko to miało miejsce. Ale z wykorzystaniem zjawiska co-segregation of alleles of nearby markers naukowcy mogą odwrócić mechanizm mejozy i zidentyfikować markery leżące blisko siebie. Następnie przy użyciu techniki statystycznej zwanej analizą genetyczną, naukowcy mogą wnioskować prawdopodobieństwo z jakim zachodzi crossing – over oraz markery zaangażowane w ten proces. Mogą również oni wywnioskować na tej podstawie czy rekombinacja zachodzi między każdą parą markerów.
Jeśli rekombinacja występuje jako zdarzenie losowe, to dwa markery, które są blisko siebie powinny występować rzadziej niż dwa markery, które są bardziej oddalone od siebie.
Prawdopodobieństwo rekombinacji między dwoma markerami, które może wynosić od 0 do 0,6, zwiększa monotonicznie odległość między dwoma markerami. Dlatego też prawdopodobieństwo rekombinacji może być użyte jako zastępstwo markerów genetycznych wzdłuż chromosomu. Jeśli natomiast określimy częstotliwość rekombinacji dla różnych par markerów, wówczas możemy zbudować mapę ich względnej pozycji na chromosomie.
Ale niestety przewidywanie rekombinacji nie jest takie proste. Fragmenty crossing – over są losowe, nie są równomiernie rozłożone na chromosomie. Niektóre regiony chromosomów, zwane „gorącymi” miejscami są bardziej podatne na crossing – over niż inne regiony chromosomu. Oznacza to, że genetyczna mapa odległości nie zawsze wskazuje fizyczne odległości między markerami. Pomimo utrudnień parametry te są wystarczające do generowania map genetycznych, które mogą służyć jako cenne ramy dla sekwencjonowania genomu.
Mapy genetyczne.
U ludzi, dane do obliczenia częstości rekombinacji poprzez badania genetyczne pokoleń członków rodziny – rodowód analizy. Badania należy rozpocząć od uzyskania próbki krwi od grupy osób powiązanych ze sobą. W przypadku rzadkich chorób, naukowcy muszą znaleźli kilka rodzin wielodzietnych, u których wielu członków było chorych i uzyskać od nich próbki. W przypadku chorób bardziej powszechnych, gdy sposób dziedziczenia choroby jest niejasny, naukowcy określają liczbę członków rodziny dotkniętej tą chorobą i pobierają próbki od 4 do 30 bliskich krewnych. DNA jest następnie zbierany ze wszystkich próbek i rozpatrywany pod kątem obecności lub współpracy dziedziczenia dwóch markerów. Jeden marker jest zwykle „zainteresowany” genem, który jest powiązany z fizycznie rozpoznawalną cechą. Drugi jest zwykle wykrywalny jednym z różnych możliwych układów wspomnianych wcześniej, tak jak mikrosatelity. Następnie wykonuje się analizę komputerową w celu ustalenie czy dwa markery są ze sobą powiązane i jak daleko są od siebie oddalone. Teraz wartości te mogą być umieszczone na mapie, wykonują dziedzictwo DNA u osób dotkniętych chorobą, ale brakuje tu zdrowej osoby, chociaż na podstawie molekularnej choroba nie może być jeszcze określona, ani gen odpowiedzialny za jej wywołanie.
Budowanie map fizycznych.
Mapy genetyczne są również używane do generowania bardziej szczegółowej mapy ludzkiego genomu. Te szczegółowe mapy zwane są mapami fizycznymi, określają sekwencje DNA między markerami genetycznymi i są niezbędne do szybkiej identyfikacji genów.
Typy map fizycznych i ich pomiar.
Mapy fizyczne można podzielić na trzy rodzaje:
chromosomalne lub cytogenetyczne, poradiacyjne (RH) i sekwencyjne. Różne rodzaje map różnią się od siebie stopniem rozdzielczości, czyli zdolności do mierzenia oddzielnych elementów, które są blisko siebie. Im wyższa rozdzielczość tym lepsze zdjęcie.
Najmniejszą rozdzielczością charakteryzują się mapy cytogenetyczne, które pokazują pasmowy wzorzec zabarwienia chromosomów. Tak jak mapy genetyczne, mapy chromosomowe mogą być użyte, aby zlokalizować cechy markerów genetycznych w organizmie. Ponieważ mapy chromosomalne są oparte na szacunku odległości fizycznej, są one uważane za mapy fizyczne. Jednak liczba par w paśmie można tylko oszacować.
RH mapy są bardziej szczegółowe. RH mapy są w stanie dostarczyć więcej dokładniejszych informacji dotyczących odległości między markerami.
Fizyczną mapą, która jest najbardziej szczegółową jest mapa sekwencyjna. Mapy sekwencyjne pokazują markery genetyczne, jak również sekwencje między mierzonymi parami markerów.
Jak wybierać mapy fizyczne i jak ich używać?
Mapowanie RH.
Za pomocą mapowania RH możemy ocenić odległości między markerami genetycznymi. Jednak, zamiast opierać się na naturalnej rekombinacji z wykorzystaniem dwóch markerów, naukowcy wykorzystują przerwy wywołane przez promieniowanie, aby ustalić odległości między dwoma markerami. W mapowaniu RH naukowiec poddaje DNA procesowi radiacji, kontrolując przy tym średnie odległości między przerwami na chromosomie. Zmieniając stopień napromieniowania DNA naukowiec może indukować przerwy między dwoma markerami, które znajdują się blisko siebie. Zdolność oddzielania blisko spokrewnionych markerów, pozwala naukowcom tworzyć bardziej szczegółowe mapy. Mapy RH pozwalają na zlokalizowanie prawie każdego markera genetycznego, jak również innych fragmentów genomu w określonym miejscu mapy oraz mapy RH są bardzo pożyteczne do porządkowania markerów w regionach, gdzie polimorficzne markery genetyczne są ograniczone. Naukowcy używają również map RH jako pomostu między powiązaniem mapy a kolejnością map. W ten sposób można łatwiej określić lokalizację genów zaangażowanych w powstawanie chorób, jakich jak zanik mięśni kręgosłupa i hypereklepxia, powszechnie znane jako „przepłoszenie reakcji”.
Kolejność mapowania.
Miejsca znaczone sekwencyjnie (STS) są innym rodzajem technik mapowania fizycznego. W STS znajdują się krótkie sekwencje DNA, które okazują się być wyjątkowe. Aby zakwalifikować STS dokładna lokalizacja i kolejność musi być znana, a ta sekwencja musi występować tylko raz w chromosomie lub genomie jako całość, jeśli zestaw fragmentów DNA obejmuje cały genom.
Specyficzne znaczniki ekspresji (ESTs) - reprezentują one znaną, unikatową sekwencję klonu cDNA, amplifikowana dzięki starannie dobranym sekwencjom starterów. Posługiwanie się starterami EST daje pewność identyfikowania sekwencji kodującej jednego genu. Aby marker EST mógł spełniać to zadanie wobec matrycy stanowiącej genomowy DNA, miejsca przyłączenia starterów powinny znajdować się w obrębie eksonu.
Polimorfizm długości prostych sekwencji (SSLPs) – zestawy powtórzeń sekwencji, które charakteryzują się różną długością. Polimorficzne SSLPs, które zostały już zmapowane są szczególnie cenne, gdyż zapewniają połączenie między mapami genetycznymi i fizycznymi.
Przypadkowe sekwencje genomu są otrzymywane przy losowym sekwencjonowaniu sklonowanych fragmentów genomu lub poprzez badanie sekwencji DNA już składowane w bazie danych.
Aby stworzyć mapę STSs wycina się fragmenty DNA z chromosomu przy użyciu enzymów restrykcyjnych, enzymy te tną DNA na fragmenty w określonych miejscach. Uzyskane dane z mapy są następnie uzyskiwane poprzez zauważalne fragmenty STSs. Naukowcy skopiowali fragmenty DNA za pomocą procesu zwanego klonowanie molekularne. Klonowanie polega na wykorzystaniu specjalnej technologii, zwanej rekombinacja DNA, aby skopiować fragmenty DNA wewnątrz obcego gospodarza.
Po pierwsze fragmenty te są połączone z przewożnikiem (wektorem). Po wprowadzeniu do odpowiedniego gospodarza, fragmenty DNA mogą być następnie powielone wraz z DNA komórki gospodarza, zapewniając nieograniczony materiał do badań eksperymentalnych. Zestawy sklonowanych fragmentów DNA tworzą tzw. biblioteki.
Następnie klony te układany są w taki sposób jak znajdują się na chromosomie. Gdy ułożenie klonów jest znane, informacje na ich temat umieszcza się w komputerze, które mogą być później wykorzystywane do dalszych badań. Dane te są następnie wykorzystywane jako materiał do utworzenia sekwencji DNA i STSs służących do tworzenia map fizycznych.
Potrzeba integracji map fizycznych i genetycznych.
Podobnie jak w przypadku najbardziej złożonych technik, technika STS wykazuje liczne ograniczenia. Fragmenty DNA mogą zostać utracone lub przez pomyłkę naniesiony na mapę w złej pozycji. Te błędy mogą występować z wielu powodów. Fragmenty DNA mogą się popsuć w wyniku czego STS wykryje je w innym miejscu. Fragmenty DNA mogą również zostać usunięte w procesie replikacji, w wyniku czego nie występują STS, które powinny się tam znajdować. Fragmenty DNA mogą również zostać zanieczyszczone przy pobieraniu materiału genetycznego, po raz kolejny prowadząc do niewłaściwie zlokalizowanego STS. Aby pomóc przezwyciężyć te problemy, jak również doprowadzić do lepszej dokładności map, badacze zaczęli porównywać i integrować STS – fizyczne z mapami genetycznymi, RH oraz cytogenetycznymi. Powołując się na różne mapy genomu zwiększa się użyteczność danej mapy.
NCBI i mapy integracyjne.
Porównywanie wielu dostępnych map genetycznych i fizycznych może być czasochłonne zwłaszcza, gdy próbuje się wskazać lokalizację nowego genomu Bez użycia komputera i specjalnych programów mających na celu dostosowanie różnych map, dopasowanie sekwencji do regionu na chromosomie, któremu odpowiada położenie w genie byłoby bardzo trudne. To tak jak próbować porównać 20 różnych międzystanowych map ulic, aby dostać się z domu w Ukiah, California do domu w Beaver Dam, Wisconsin. Można porównać te mapy do siebie i stworzyć własną trasę podróży, ale prawdopodobnie będzie to długo trwało. Czy nie byłoby łatwiej i szybciej, gdybyśmy mieli automobil club, który stworzyłby tą mapę dla ciebie? To jest właśnie zadanie NCBI's Human Genome Map Viewer.
NCBI Map Viewer: narzędzie służące do porównywania map genetycznych i fizycznych.
NCBI Map Viewer umożliwia graficzne wyświetlenie dostępnych danych dotyczących sekwencji ludzkiego genomu, jak również sekwencji cytogenetycznej, genetycznej i RH. Map Viewer może jednocześnie wyświetlać maksymalnie siedem map, wybranych spośród dużego zestawu map i pozwala użytkownikowi na dostęp do szczegółowych informacji o wybranym regionie mapy. Map Viewer używa wspólnego systemu numeracji w celu dostosowania kolejności na mapie i wspólnych markerów, jak również nazw genów w celu dostosowania ich do innych map. Możesz użyć NCBI Map Viewer do poszukiwania genów w wielu genomach, wybierając opcje ze strony głównej Map Viewer.
Modelowanie molekularne - metoda poznawania struktury i funkcji białek.
Białka są podstawowymi składnikami wszystkich żywych komórek. Charakteryzują się one ogromną różnorodnością chemiczną i strukturalna, dzięki czemu posiadają zróżnicowany zakres funkcji biologicznych. Białka walczą z zakażeniami, kontrolują chemię naszego organizmu, w ogóle, dzięki nim całe nasze ciało funkcjonuje sprawnie. Krytyczna funkcja białka to jest jego zdolność do dopasowania kształt do przeprowadzenia danej funkcji. Ale czasami białka mogą zmienić swój kształt lub utracić jakąś część. Wiele chorób, takich jak choroba Alzheimera i "szalonych krów", wynikają ze zmiany konformacji białek.
Identyfikacja białka kształtu lub struktury, jest kluczem do zrozumienia jego biologicznej funkcji i roli w zakresie zdrowia i choroby. Struktura białka daje możliwość rozwoju nowych czynników i urządzeń mających na celu leczenia chorób. Jednak poznanie struktury białka nie jest łatwe. Naukowcy często pracują w laboratorium miesiącami, czasem latami, by doświadczalnie określić strukturę białka. Dlatego naukowcy zaczęli używać komputerów w celu przewidywania struktury białek na podstawie jego sekwencji.
Do produkcji białek, służą struktury komórkowe zwane rybosomami, które łączą razem długie łańcuchy składające się z podjednostek. Zestaw 20 różnych podjednostek, zwanych aminokwasami, może być ustawiony w każdej kolejności by zbudować polipeptyd, który może składać się z tysiąca aminokwasów. Łańcuchy te mogą tworzyć pętle ze sobą, lub fałdy, w różny sposób, ale tylko jeden z tych sposobów pozwala białku funkcjonować prawidłowo. Krytyczną funkcją białka jest jego zdolność do tworzenia konformacji, która stanowi o jego strukturalnych cechach, takich jak powierzchnia rowków, grzebienie oraz kieszenie, które pozwalają na spełnienie ich roli w komórce. Konformacja białka jest zwykle opisywana na podstawie poziomów struktury. Tradycyjnie, białka mają cztery różne poziomy struktury, gdzie każdy poziom struktury zależy od tego pod nim. W niektórych białkach, funkcjonalna różnorodność może wzrosnąć przez dodanie nowych grup chemicznych po zakończonej syntezie.
Łańcuch sekwencji aminokwasów formujący polipeptyd nazywany jest strukturą pierwszorzędową.Struktura drugorzędowa jest generowana przez składanie pierwszorzędowej sekwencji oraz odnosi się do toru jaki kręgosłup polipeptydu w białku przyjmie w przestrzeni. Niektóre typy struktur drugorzędowych są szczególnie powszechne. Dwie dobrze opisane struktury drugorzędowe to alfa-helisa i beta-kartka. W pierwszym przypadku, niektóre typy wiązań pomiędzy grupami zlokalizowane w tym samym łańcuchu polipeptydowym powodują że kręgosłup skręca się w helisę, najczęściej jest to alfa-helisa. Beta-kartka jest formowana, gdy wiązania jednego łańcucha polipeptydowego biegną w przeciwnym kierunku niż w innym łańcuchu. Beta-kartka może być również formowana między dwoma częściami pojedynczego łańcucha polipeptydowego, który jest ułożony tak aby, sąsiadujące regiony występowały w odwrotnym kierunku.
Białka można podzielić na dwie ogólne kategorie na podstawie ich struktury trzeciorzędowej.
Trzeciorzędową strukturę opisuje organizacja w trzech wymiarach wszystkich atomów w polipeptydzie. Jeśli białko składa się tylko z jednego łańcucha polipeptydowego, ten poziom opisuje kompletna strukturę.
Białka wielorzędowe lub białka, które składają się z więcej niż jednego łańcucha polipeptydowego, wymagają wyższego poziomu organizacji. W strukturach czwartorzędowych konformacja jest determinowana przez wielorzędowe białka. W tym przypadku indywidualny łańcuch polipeptydowy, który tworzy wielorzędowe białka jest często określany jako podjednostka białka. Cztery poziomy struktury białka są hierarchiczne, co znaczny, że każdy poziom w procesie budowy zależy od poziomu niższego.
Białka można podzielić na dwie ogólne kategorie na podstawie ich struktury trzeciorzędowych.
Podstawowa sekwencja łańcucha aminokwasowego ma zasadnicze znaczenie w określaniu ostatecznej struktury. W niektórych przypadkach, sekwencja aminokwasów jest jedynym czynnikiem, podczas gdy w innych, dodatkowe interakcje mogą być wymagane przed tym zanim białko może osiągnąć swoją ostateczną konformację, Na przykład, niektóre białka wymagają obecności kofaktora, lub drugiej cząsteczki, która jest częścią aktywnego białka, przed osiągnięciem ostatecznej konformacji. Wielorzędowe białka często wymagają jednej lub więcej podjednostek, by być prezentowanym dla innych podjednostek aby przyjąć prawidłową wyższą strukturę. W każdym razie, jak podano wcześniej, cały proces jest wspólny, tak jak, utworzenie jednego regionu drugorzędowej struktury, determinuje utworzenie innego regionu.
Białka allosteryczne
Białka te mogą zmieniać swój kształt i funkcję zależnie od warunków środowiska w którym się znajdą. W pewnych warunkach, białka mogą mieć trwałą alternatywną konformację, lub kształt, który nadaje im inną funkcję biologiczną. Białka, które posiadają tą cechę nazywają się allosterycznymi. Interakcja między allosterycznym białkiem a specyficznym kofaktorem, lub z innym białkiem, mogą powodować zmianę białka w kształcie. W dodatku, każda zmiana w konformacji spowodowanej interakcji w jednym miejscu mogą prowadzić do zmian w strukturze, a więc w funkcji w innym miejscu. Należy jednak pamiętać, że tego typu zmiany dotyczą tylko kształtu białka, a nie podstawowej sekwencji aminokwasów. Allosteryczne białka odgrywają ważną rolę w metabolicznej i genetycznej regulacji.
Tradycyjnie struktury białka są ustalane przy użyciu jednej z dwóch technik: krystalografii rentgenowskiej i jądrowego rezonansu magnetycznego (NMR).
Krystalografia promieniowania rentgena
Podczas wykonywania tej techniki, molekuły podczas badania muszą najpierw być poddane krystalizacji, kryształy muszą być pojedyncze oraz doskonałej jakości – jest to czasochłonne i trudne zadanie.
Kryształy są to trwałe formy substancji, w których składniki cząsteczek są obecne w uporządkowanej matrycy nazywanej kratownicą. Podstawowym składnikiem budulcowym kryształu jest jednostka komórki. Każda jednostka komórki zawiera dokładnie jeden unikalny zestaw kryształu z części składowych, najmniejszy możliwy zestaw jest w pełni reprezentatywny dla kryształu. Kryształy kompleksu molekuł, jak w białkach, wytwarzają złożone struktury dyfrakcji rentgenowskiej, lub rozproszenia promieniowania rentgenowskiego. Gdy kryształ jest umieszczany w wiązce promieniowania rentgenowskiego, wszystkie jednostki komórek obecnych w tych samych miejscach do wiązki, dlatego wiele molekuł jest w tej samej orientacji w odniesieniu do przechodzącego promieniowania rentgenowskiego. Wiązka wchodzi w kryształ i kilka mniejszych wiązek pojawia się: każdy z nich w innym kierunku, każdy z różną intensywnością. Jeśli detektor promieniowania rentgenowskiego, tak jak fragment filmu, umieszczany jest po przeciwnej stronie kryształu niż źródło promieniowania, każde rozszczepienie promieniowania, nazywane, refleksją, powstanie jako miejsce na filmie. Jednakże, ponieważ tylko kilka refleksji można wykryć w jednej orientacji kryształu, ważnym elementem wszelkich dyfrakcji promieniowania rentgenowskiego jest urządzenie do dokładnego ustalania i zmiany orientacji kryształu. Zestaw rozproszonych wiązek wchodzących zawiera informacje na temat podstawowej struktury krystalicznej.
Jeśli moglibyśmy używać światła zamiast promieniowania rentgenowskiego, moglibyśmy stworzyć system soczewek do rekombinacji wiązek wychodzących z kryształu, a tym samym doprowadzić do skoncentrowania się rozszerzonego wizerunku jednostki komórki i molekuł w niej. Jednak molekuły nie rozszczepiają światła widzialnego, i promieniowania rentgenowskiego, w odróżnieniu od światła, nie mogą być skoncentrowane w soczewkach. Jednak, prawa naukowe, którym są poddawane soczewki są zrozumiałe, i jest to możliwe do obliczenia molekularnego obrazu z komputera. W efekcie komputer naśladuje działanie obiektywu.
Główną wadą związaną z tą techniką jest to iż krystalizacja białek jest trudnym zadaniem. Kryształy są formowane przez powolną precypitacje białek w warunkach, które utrzymują je w natywnej konformacji lub strukturze. Te dokładne warunki mogą być tylko odkryte przez powtarzające się próby, które pociągają za sobą pewne urozmaicone eksperymentalne warunki, po jednym. W niektórych przypadkach, zadania krystalizacji białek są niemożliwe.
Spektroskopia jądrowego rezonansu magnetycznego (NMR)
Badanie NMR jest przeprowadzane dla makromolekuł podczas spadania i wibrowania z termicznym ruchem tych molekuł.
Podstawowe zjawiska NMR zostały odkryte w 1945 roku. W tej technice, próba jest zanurzana w polu magnetycznych i bombardowana falami radiowymi. Fale te powodują iż jądro w molekule zostaje poddane rezonansowi lub spinowi. Pozytywnie naładowane spinowe jądro, tworzy poruszający się ładunek który nazywany jest momentem magnetycznym. Termiczny ruch molekuły – ruch cząsteczki związany z temperaturą materiału – ponadto tworzy moment obrotowy, lub siły skręcania, które powodują, że moment magnetyczny „trzęsie się” jak dziecko w górę. Kiedy fale radiowe trafią w jądro w spinie, one pochylają się nawet mocniej, czasem nawet przewracają się. Czasem jądro w rezonansie emituje unikatowy sygnał, który jest następnie zabierany przez specjalny odbiornik radiowy i tłumaczony za pomocą dekodera. Dekoder ten jest nazywany transformatą algorytmu Fouriera, złożonym równaniem, które przekłada język w jądrach w coś co naukowiec jest w stanie zrozumieć. Przez pomiar częstotliwości, w którym różne jądra zostają przewrócone, naukowcy determinują molekularną strukturę, tak samo jak wiele innych właściwości molekuł.
W ciągu ostatnich 10 lat, NMR okazał się potężną alternatywą dla krystalografii rentgenowskiej dla określania struktury molekuł. NMR ma przewagę nad technikami krystalografii, w tych eksperymentach w które rozwiązywane są przeciwnie do kryształu kraty. Jednakże, zasady, które czynią NMR możliwe, są czasochłonne i ograniczają się do małych i średnich cząsteczek.
Naukowcy przez dziesięciolecia dążyli do opracowania procedur przewidywania struktury białek, które nie są tak czasochłonne, i które nie są hamowane przez ograniczenia wielkości i rozpuszczalności. Aby to uzyskać, naukowcy zwrócili się do „komputerów” o pomoc w przewidywaniu struktury białek na podstawie sekwencji genu, a to pojęcie nazywane jest modelowaniem homologicznym. Kompletne genomy różnych organizmów, w tym ludzi, zostały zsekwencjonowane (zdekodowane) i pozwalają naukowcom do podejścia do swojego celu w sposób logiczny i zorganizowany.
Zanim pójdziemy dalej, jest ważne aby określić kilka wspólnych terminów używanych w tej dziedzinie.
Niektóre podstawowe teorie
Obraz struktury białka generowany komputerowo pokazuje względne lokalizacje większości, jeżeli nie wszystkich, z tysięcy atomów w białku. Dodatkowo obraz pokazuje fizyczne, chemiczne oraz elektryczne właściwości białek oraz dostarcza wskazówek na temat ich roli w organizmie.
Teoretycznie białka, które mają podobną sekwencję zazwyczaj mają również tą samą podstawową strukturę. Dlatego też, eksperymentalnie określona struktura jednego przedstawiciela rodziny białek, nazywanego obiekt (target), dostarcza naukowcom modelu, na którym opiera się struktura innych białek tej rodziny. Idąc krok dalej, wybierając obiekt każdej superrodziny naukowcy mogą badać wszechświat zagięć białka w sposób systematyczny i podkreślać zestaw sekwencji związanych z każdym motywem zagięć. Wiele z tych sekwencji może nie wykazywać podobieństwa do siebie, ale ich identyfikacja i przypisanie do konkretnego zagięcia ma zasadnicze znaczenie dla przewidywania przyszłych struktur białkowych używając modelowania homologicznego.
Podstawą naukową dla tych teorii jest silnie zachowanie trójwymiarowego kształtu przez białka o dużych odległościach ewolucyjnych, zarówno w obrębie jednego gatunki, między gatunkami oraz pomimo zmienności sekwencji – wykazano to wiele razy. Chociaż większość naukowców wybrała struktury wysoko-priorytetowe do swoich celów, ta teoria przewiduje że możliwość wyboru jednego z białek w rodzinie jako obiektu, a nie stara się osiągnąć eksperymentalne wyniki przy użyciu białka, które są szczególnie trudne w pracy z użyciem krystalografii lub technik NMR.
Kroki prowadzące do maksymalizacji wyników
Szczególne zadania muszą być prowadzone by maksymalizować wyniki podczas określania struktury białek przy użyciu modelowania homologicznego.
Po pierwsze, sekwencje muszą być zorganizowane w postaci rodziny, najlepiej w postaci bazy danych, i musi zostać wybrany obiekt. Białko rodziny może być zidentyfikowane i zorganizowane przez porównanie sekwencji białka pochodzącej z zsekwencjonowanego genomu. Obiekty mogą być wybrane dla rodzin, które nie wykazują widocznych homologi w sekwencji do białek ze znaną trójwymiarową strukturą.
Następnie naukowcy muszą uzyskać oczyszczone białka do analiz wybranego obiektu, a następnie eksperymentalnie określić strukturę obiektu, albo przez krystalografię oraz/albo przez NMR. Struktura obiektu określona doświadczalnie może być następnie analizowana w celu oceny ich podobieństwa do innych znanych struktur białka oraz do określenia możliwości ewolucyjnych związków, które nie są rozpoznawalne przez same sekwencje białek. Struktura obiektu będzie również służyć jako szczegółowy model do determinacji struktury innych białek w obrębie tej rodziny. W korzystnych przypadkach, tylko znając strukturę pojedynczych białek może to również przynieść znaczny wgląd w jego ewentualne funkcje.
PDB była pierwszą bioinformatyczną bazą danych kiedykolwiek zbudowaną i zaprojektowaną do przechowywania złożonych trójwymiarowych danych. PDB był pierwotnie opracowany i trzymany w Brookhaven National Laboratories, ale jest teraz zarządzany i utrzymywany przez Research Collaboratory for Structural Bioinformatics (RCSB). PDB to zbiór wszystkich dostępnych publicznie trójwymiarowych struktur białek, kwasów nukleinowych, węglowodanów i wiele innych kompleksów doświadczalnie wyznaczanych przez krystalografię rentgenowską i NMR.
PDB jest wspierane przez fundusze z National Science Foundation, Department of Energy, i dwie jednostki Naukowego Instytutu Zdrowia: Krajowy Instytut Ogólnych Nauk Medycznych oraz Bibliotekę Narodową Medycyny.
Baza danych modelowania molekularnego
NCBI baza danych modelowania molekularnego (MMDB), integralna część systemu wyszukiwania informacji Entrez, jest kompilacją wszystkich PDB trójwymiarowych struktur biomolekuł. Różnica między dwoma bazami danych jest taka że rekordy MMDB są organizowane i weryfikowane w informacje zapisane w bazie danych w sposób, który umożliwia porównanie między chemią a trójwymiarową strukturą makromolekuł. Integrując informacje o chemii, sekwencji i strukturze, MMDB ma służyć jako źródło informacji dla konstrukcji struktury białek opartych na homologii modelowania.
Rekordy MMDB mają dodane wartościowe informacje w porównaniu do oryginalnych wpisów PDB, w tym wyraźne wykres chemiczne, uzyskane przez struktury drugorzędowe, informacje z domen struktur, dopasowanie cytowanej literatury i podstawowe cząsteczki powiązane z taksonomią do każdego biologicznie przypisanego białka albo łańcucha kwasu nukleinowego.
NCBI opracował również program do uwidaczniania trójwymiarowej struktury, nazwany Cn3D, dla łatwej interaktywnej wizualizacji struktur molekularnych z Entrez. Cn3D służy jako narzędzie do wizualizacji sekwencji i alignmentu sekwencji. Co wyróżnia Cn3D, spośród innego oprogramowania to jego zdolność do skorelowania informacji o strukturze i sekwencji. Na przykład, używając Cn3D, naukomanie może szybko zlokalizować pozostałości w strukturze kryształu, które odpowiadają znanym chorobom opierającym się na mutacjach albo zachowanym aktywnym pozostałościom z rodziny sekwencji homologów, albo sekwencji, które łączy wspólny przodek. Cn3D wyświetla strukturę – strukturę alignmentów wraz z odpowiednimi strukturami – opartymi na sekwencji alignmentów aby podkreślić te regiony, w ramach grupy powiązanych białek, która są najbardziej zachowane w strukturze i sekwencji. Cn3D posiada również niestandardowe funkcje etykietowania opcji, wysokiej jakości grafiki, a różnorodność eksportu plików, które stanowią łącznie Cn3D potężnym narzędziem dla adnotacji literaturowych.
PDBeast: Taksonomia w MMDB
Taksonomia jest naukową dyscypliną, która zmierza do katalogowania i odtworzenia ewolucyjnej historii życia na Ziemi. NCBI Structure Group, we współpracy z taksonomistami NCBI, podjęła się dopisania adnotacji taksonomicznych dla danych przechowywanych w MMDB. Zostało wdrożono częściowo automatyczne podejście w którym ekspert sprawdza, koryguje i potwierdza zadania automatyczne.
Program PDBeast zostało opracowane przez NCBI do tego celu. Wyciąga z tekstu opis „Źródło organizmu” w oryginalnym zapisie PDB lub określonych przez użytkownika informacji i szuka dopasowania w bazie danych taksonomi NCBI w celu rejestrowania zadań taksonomicznych.
Rola taksonomii:
COGs: filogenetyczna klasyfikacja białek
Baza danych Clusters of Orthologous Groups białek (COGs) stanowi próbę filogenetycznej klasyfikacji białek – system uwzględnia ewolucyjne powiązania między organizmami – od kompletnych genomów. Każdy COG zawiera białka, które są uznawane za ontologiczne. Ortologi są genami u różnych gatunków pochodzących od wspólnego przodka i prowadzone przez ewolucję. COG mogą być używane do wykrywania podobieństw i różnic między gatunkami do identyfikacji rodziny białek oraz przewidywania nowych funkcji białek i wskazują na potencjalne celowość użycia leków w chorobach różnych gatunków. Baza danych występuje wraz z programem COGnitor, który wyznacza nowe białka, zazwyczaj z nowo zsekwencjonowanych genomów, do istniejących wcześniej COGs. Strona internetowa zawiera dodatkowe strukturalne i funkcjonalne informacje, które są obecnie związane z każdym COG. Ta strona zawiera informacje zawierające: systematyczną klasyfikację użytkowników COG w ramach różnych systemów klasyfikacji, wskazania, które COG (jeśli jakieś) zostały scharakteryzowane genetycznie i biochemicznie; informacje na temat architektury białek oraz trójwymiarowej struktury domen jeśli są znane lub przewidywane; zwięzłe podsumowanie wspólnych cech strukturalnych i funkcjonalnych COG, jak również specyfikę pojedynczego członka; oraz kluczowe odniesienia.
Wykrywanie nowych podobieństw sekwencji: BLAST przeciwko MMDB
Artykuł w czasopiśmie opisujący oryginalny algorytm używany w BLAST stał się jednym z najczęściej cytowanych dokumentów dziesięciolecia, z ponad 10 000 cytowań.
Porównanie, czy to funkcji strukturalnych czy sekwencji białek, leży w samym sercu biologii. Wprowadzenie do BLAST, czy Podstawowego Narzędzia Wyszukiwania Local Alignment, w 1990 sprawiło, że szybkie przeszukiwanie wielkich baz danych w celu znalezienia homologii, albo podobieństw sekwencji oraz ich statystyczna ocena stała się łatwiejsza. BLAS działa poprzez porównywanie nieznanej sekwencji użytkownika do znanych sekwencji bazy danych aby określić najbardziej pożądane dopasowania. Podobne sekwencje zostały wyszukane krytycznie przez BLAST w kilku odkryciach genów. Setki sekwencji głównych ośrodków oraz instytucji badawczych z całego kraju używają tego oprogramowania do przesłania ich wyszukiwanej sekwencji z lokalnego komputera na serwer BLAST na NCBI za pośrednictwem Internetu. W ciągu kilku sekund, serwer BLAST porównuje sekwencję użytkownika z milionem znanych sekwencji i określa najbliższe dopasowania.
Jednakże, nie wszystkie znaczące homologie są łatwo wykrywane. Niektóre z najbardziej interesujących są podobieństwami subtelnymi, które nie zawsze prowadzą do istotności statystycznej, podczas standardowego wyszukiwania BLAST. Dlatego NCBI rozszerzył, statystyczną metodologię używaną w oryginalnym BLAST do problemu z wykrywaniem słabych, ale znaczących podobieństw sekwencji. PSI-BLAST lub Position-Specyfic Iterated BLAST, przeszukuje sekwencje baz danych przez profil skonstruowany z wykorzystaniem BLAST alignments, od którego następnie konstruuje tzw. Position-Specyfic score matrix (matrycę z specyficznymi pozycjami, za które przyznaje punkty). Do analizy białek, nowy Pattern Hit Initiated BLAST, albo PHI-PLAST, służy do uzupełniania profilu opartego na wyszukiwaniu, które zostało wcześniej prowadzone z użyciem PSI-BLAST. PHI-BLAST zawiera dalsze hipotezy co do biologicznych funkcji wyszukiwanej sekwencji oraz ogranicza analizę do zestawu sekwencji białka, który zawiera znane specyficzne wzory i motywy.
BLAST jest teraz w kilku odmianach w uzupełnieniu o te funkcje opisane powyżej. Specjalistyczny BLAST dostępne są także dla ludzi, mikroorganizmów i innych genomów, jak również dla wektora zakażeń, immunoglobulin, oraz niepewnych sekwencji ludzkiego porozumienia.
Wyszukiwanie strukturalnego podobieństwa za pomocą VAST
Jak wspomniano, program podobieństwa sekwencji stanowi dopasowanie dwóch zestawów sekwencji. Program podobieństwa struktury stanowi superpozycję dla trójwymiarowej struktury. Serwis wyszukiwania podobieństw struktur jest oparty na założeniu, że pewne działania mogą być obliczane między dwoma strukturami w celu oceny ich podobieństwa, bardzo podobnie jak wykonuje to BLAST.
VAST, albo Vector Alignment Serach Tool, jest komputerowym algorytmem opracowanym przez NCBI w celu stosowania go, do identyfikacji podobnych trójwymiarowych struktur białek. VAST jest zdolny do wykrycia strukturalnych podobieństw miedzy białkami zapisanymi w MMDB, nawet jeśli nie zostanie wykryte żadne podobieństwo w sekwencji.
Wykrycie strukturalnych podobieństwo w przypadku braku oczywistych podobieństw sekwencji jest potężnym narzędziem w badaniach nad odległymi ewolucyjnie homologami i białkami.
Wyszukiwarka VAST, to serwis NCBI do wyszukiwania podobieństw strukturalnych, któ®y porównuje współrzędne trójwymiarowe nowo ustalonych struktur białek, do tych w MMDB albo bazie danych PDB. Wyszukiwarka VAST tworzy listę strukturalnych sąsiadów, albo powiązanych struktur, które użytkownik może następnie wybrać interaktywnie. Wyszukiwarka VAST będzie pobierać prawie wszystkie struktury z identyczną trójwymiarową fałdą, chociaż czasami może ominąć kilka struktur albo raportować o szansie podobieństwa.
Baza danych konserwatywnych domen
Baza danych konserwatywnych domen (CDD) to zbiór alignmentów sekwencji oraz profili reprezentujących domeny białek zachowane w ewolucji molekularnej. Obejmuje domeny z SMART oraz Pfam, dwóch popularnych opartych na Web narzędziach dla badań sekwencji domen, jak również wniesione przez naukowców NCBI. Wyszukiwarka CD, inny serwis NCBI, może być użyte do identyfikacji zachowanych domen w wyszukiwanej sekwencji białka. Wyszukiwarka CD wykorzystuje RPS-BLAST aby porównać wyszukiwaną sekwencję z specyficznymi matrycami, które były przygotowane z konserwatywnych alignment domen prezentowanych w CDD. Alignmenty są także mapowane do znanych trójwymiarowych struktur i mogą być wyświetlane przy użyciu Cn3D (patrz wyżej).
Narzędzie do uzyskiwania architektury konserwatywnej domeny
Narzędzie NCBI do uzyskiwania architektury konserwatywnej domeny (CDART) wyświetla funkcjonalne domeny, które tworzą białka i wymienia inne białka o podobnej architekturze domen. CDART określa architekturę domeny wyszukiwanej sekwencji białka przez porównanie go z CDD, bazą danych konserwatywnych domen alignmentów, używając RPS-BLAST.
CDART następnie porównuje białkowe architektury domen do innych białek NCBI z dazy danych sekwencji niezbędnych. Podobne sekwencje zidentyfikowane jako te białka, która dzielą jedną lub więcej podobnych domen. CDART wyświetla te sekwencje przy użyciu graficznego podsumowania, które przedstawia rodzaje i miejsca domen określone w każdej sekwencji. Linki do poszczególnych sekwencji, jak również do dalszych informacji na temat ich struktury domen są również udostępniane. Ponieważ domeny białek mogą zostać uznane za podstawowe jednostki funkcji molekularnych oraz białek związanych z strukturą domeny mogą odgrywać podobną rolę w procesach komórkowych, CDART służy jako przydatne narzędzie do analizy porównawczej sekwencji.
RPS-BLAST jest „odwróceniem” wersji PSI-BLAST, które jest opisane powyżej. Oba RPS-BLAST i PSI-BLAST wykorzystują podobne metody do wprowadzenia konserwatywnych cech rodziny białek. Jednakże, RPS-BLAST porównuje wyszukiwaną sekwencję z bazą danych profili przygotowanych z gotowych alignmentów, podczas gdy PSI-BLAST buduje alignment począwszy od pojedynczej sekwencji białkowej. Programy różnią się także w celu: RPS-BLAST jest używany do identyfikacji konserwatywnych domen w wyszukiwanej sekwencji, natomiast PSI-BLAST jest używany do identyfikacji innych członków rodziny białek, do których wyszukiwana sekwencja należy.
Chociaż informacje uzyskane z badań modelowania są przede wszystkim o funkcjach molekularnych, danych strukturalnych białek, dodatkowo dostarczają bogactwo informacji o mechanizmach związanych z funkcjami oraz historii ewolucji powiązań pomiędzy makromolekułami. Celem NCBI w dodawaniu danych o strukturze na ich stronę internetową jest sprawić aby te informacje były łatwo dostępne dla społeczności biomedycznej na całym świecie oraz aby ułatwić porównywanie analizy obejmującej trójwymiarową strukturę.
Czy nie byłoby wspaniałe, jeśli wiedzielibyśmy dokładnie, jakie działania można podjąć, aby zażegnać, a nawet uniemożliwić, początek choroby? Czy nie byłoby ulgą wiedzieć, że nie jesteś uczulony na leki przepisane przez lekarza.Czy nie byłoby cudownie wiedzieć, że leczenia jakim jesteś poddawany mają szansę powodzenia, ponieważ zostały zaprojektowane właśnie dla Ciebie? Naukowcy uważają że to może być wkrótce możliwe.
Jakie są SNPs?
DNA (kw. dezoksyrybonukleinowy) składa się z czterech rodzajów nukleotydów. Nukleotyd z kolei tworzą 3 składniki: - element cukrowy – w wypadku DNA jest to pentoza; - zasada azotowa – pierścieniowy związek organiczny, który w szkielecie węglowym posiada atomy azotu; w przypadku DNA mamy 4 zasady azotowe:1) dwupierścieniowe puryny: adenina (A) i guanina (G) 2) jednopierścieniowe pirymidyny: cytozyna (C) i tymina (T)
Przykładem z SNP jest zmiana w DNA- AAGGTTA na ATGGTTA, gdzie "A" zastępuje "T". Przeciętnie SNPs występuje w populacji ludzkiej więcej niż 1 procent. Ponieważ tylko około 3 do 5 procent to sekwencje DNA znajdujące się w regionie kodującym białeka, natomiast większość SNPs znajdują się poza "regionem kodowania". SNPs występujące w regionie kodowania sekwencji są przedmiotem szczególnego zainteresowania naukowców, ponieważ są one bardziej narażone na zmiany biologiczne funkcji białka.
Znalezienie pojedynczego nukleotydu ludzkiego genomu wydaje się być trudną perspektywą, ale w ciągu ostatnich 20 lat, naukowcy opracowali szereg technik biomedycznych, które umożliwiają nie tylko to. Każda technika wykorzystuje różne metody porównywania wybranych regionów sekwencji DNA uzyskanych od wielu osób, które mają wspólne cechy. W każdym badaniu, wynik pokazuje fizyczne różnice w DNA próbek SNP tylko w przypadku, gdy zostanie wykryta w jednym, a nie w innych.
Wiele wspólnych chorób u ludzi nie jest spowodowanych przez zmianę genetyczną w obrębie jednego genu, ale powstają pod wpływem wielu skomplikowanych interakcji między genami, jak również czynnikami środowiskowiskowymi i stylem życia. Choć zarówno czynniki środowiskowe i styl życia, są obecnie trudne do zmierzenia i oceny ich ogólnego wpływu na proces choroby. Dlatego też zwraca się uwagę głównie na osoby z uwarunkowaniami genetycznymi, i poznanie choroby w oparciu o geny i czynniki dziedziczne.
Genetyczne czynniki mogą również wpływać na podatność lub odporność na choroby i ustalenie stopnia lub progresji choroby. Ponieważ jeszcze nie wszystkie z tych czynników są poznane, naukowcy odkryli, że trudno rozwijać badania dla większości chorób i zaburzeń. Definiowanie i rozumienie roli czynników genetycznych w chorobie umożliwi naukowcom lepiej ocenić rolę braku czynników genetycznych, takich jak zachowanie, sposób odżywiania, stylu życia i aktywności fizycznej, na choroby.
Ponieważ czynniki genetyczne mają również wpływ na organizm w odpowiedzi na leczenie, polimorfizm DNA tj. SNPs będzie pomocny naukowcom w zrozumieniu, dlaczego osoby różnią się pod względem ich zdolności do przyjmowania niektórych leków, jak również w celu ustalenia, dlaczego u danej osoby może wystąpić niekorzystny efekt uboczny po zażyciu danego leku. W związku z niedawnym odkryciem SNPs obiecuje się zrewolucjonizować nie tylko proces wykrywania choroby, ale również profilaktykę i udoskonalanie leków
Każda osoba jako materiał genetyczny zawiera unikalny wzór SNP, który składa się z wielu różnych odmian genetycznych. Naukowcy stwierdzili, że większość SNPs nie jest odpowiedzialnych za choroby. Zamiast tego służą jako markery biologiczne identyfikujace chorobę na mapie ludzkiego genomu, ponieważ zwykle są one zlokalizowane w pobliżu genów uznanych za związane z niektórymi chorobami. Czasami SNP może w rzeczywistości prowadzić do choroby.
Naukowcy wykonali badania genetyczne, w celu wykrycia genów powodujących choroby, zostały one zidentyfikowane; naukowcy zbierają próbki krwi od grupy osób dotkniętych chorobą i wykonują analizę DNA dla wzorców SNP. Następnie naukowcy porównują te wzorce z uzyskanymi poprzez analizę DNA z grupy osób niedotkniętych przez choroby. Tego typu porównania, zw. "stowarzyszenie studiów", umożliwiają wykrycie różnic między wzorcami SNP dwóch grup, wskazując tym samym, który jest wzorem najprawdopodobniej związany z chorobą Ostatecznie profile SNP, które są charakterystyczne dla różnych chorób zostaną ustanowione. Następnie, co będzie tylko kwestią czasu, zanim lekarze będą mogli szybko stwierdzić podatność na chorobę tylko poprzez analizę ich DNA próbek dla poszczególnych wzorców SNP.
Jak wspomniano wcześniej, SNPs jest również związane z oddziaływaniem leków na różne organizmy. Obecnie nie istnieje prosty sposób by określić, w jaki sposób pacjent będzie musiał przyjmować leki, gdyż mogą one różnie wpływać na różnych pacjentów. Na jednych mogą zadziałać leki, na drugich nie. Gorzej kiedy, u niektórych pacjentów mogą wystąpić niekorzystne reakcje immunologiczne dla danego leku. Obecnie firmy farmaceutyczne są ograniczone. W rezultacie, istnieje wiele leków, z których może korzystać niewielka liczba pacjentów.
W przyszłości, najbardziej odpowiednie dla poszczególnych leków może być ustalenie z góry profilu SNP, poprzez analizę pacjenta.
Ponieważ SNPs często występują w całym genomie i zwykle są stosunkowo stabilne genetycznie mogą one służyć jako doskonałe markery biologiczne. Markery biologiczne są tosegmenty DNA pozwalające na zidentyfikowanie fizycznej lokalizacji, która może być łatwo śledzona i wykorzystywana do budowy mapy chromosomów pokazujacej miejsca znanych genów, lub innych markerów. Mapy te umożliwiają naukowcom badania i dentyfikowanie cech wynikających z interakcji z więcej niż jednym genem. NCBI odgrywa istotną rolę w ułatwianiu identyfikacji i katalogowania SNPs poprzez jego utworzenie i utrzymanie publicznych danychSNP (dbSNP). To potężne narzędzie genetyczne może być dostępne dla biomedycznych społeczności na całym świecie i ma na celu stymulowanie wielu dziedzin badań biologicznych, w tym identyfikacji genetycznej składników choroby.
W celu ułatwienia badań naukowych, NCBI's dbSNP jest wliczone w system, który zapewnia zintegrowany dostęp do wielu narzędzi, oprogramowania i baz danych, które mogą pomoc w analizie SNP. Zapewnienie dostępu do strony internetowej dla "one-stop-shopping SNP" ułatwia badania naukowe w różnych dziedzinach, począwszy od genetyki populacji i biologii ewolucyjnej, po szeroką skalę chorób i leków. Do długoterminowych inwestycji należą nowe i ekscytujące takich badania mogące zrewolucjonizować nowoczesną medycynę.
Badacze ciężko pracują nad zsekwencjonowaniem genomów różnych organizmów, w tym myszy i ludzi. Głównym celem sekwencjonowania genomów jest identyfikacja pełnego zestawu genów oraz uzyskanie informacji gdzie, kiedy i jak dochodzi do ekspresji konkretnego genu. Kiedy zaczynamy rozumieć, gdzie i w jaki sposób gen jest włączany w normalnych warunkach, możemy następnie badać co się dzieje w zmianie stanu, takich jak choroby. Aby osiągnąć ten ostatni cel, jednak naukowcy muszą zidentyfikować i przeprowadzać badania białek kodowanych przez dany gen.
Jak można sobie wyobrazić, znalezienie genu kodującego dane białko lub białka, nie jest łatwe. Początkowo naukowcy rozpoczynają badania poprzez zdefiniowanie problemu i opracowanie strategii. Często rozpoczyna się od wyszukiwania informacji w literaturze naukowej. Pozwala to na zaoszczędzenie czasu i wykonywania badań które częściowo były już przeprowadzenie oraz możliwości skorzystania z wyników tych badań.
Obecnie dzięki szybkiemu rozwojowi nowych technologii oraz łatwemu dostępowi do danych badawczych z całego świata, cały proces badawczy jest ułatwiony i skrócony. ESTs ma za zadanie zapewnić naukowcom szybką i niedrogą drogę odkrywania nowych genów, w celu uzyskania danych na temat ekspresji genów i regulacji, a także do budowy mapy genomu.
ESTs to małe kawałki sekwencji DNA (zwykle od 200 do 500 nukleotydów długości), które są generowane przez sekwencjonowanie jeden lub oba końce genu. Chodzi o sekwencję DNA, które stanowią znajduję się w niektórych komórkach, tkankach lub narządach pochodzących z różnych organizmów i wykorzystywaniu tych "tagów", w celu odnalezienia interesujących nas genów z części chromosomów DNA przez dopasowanie par zasad.
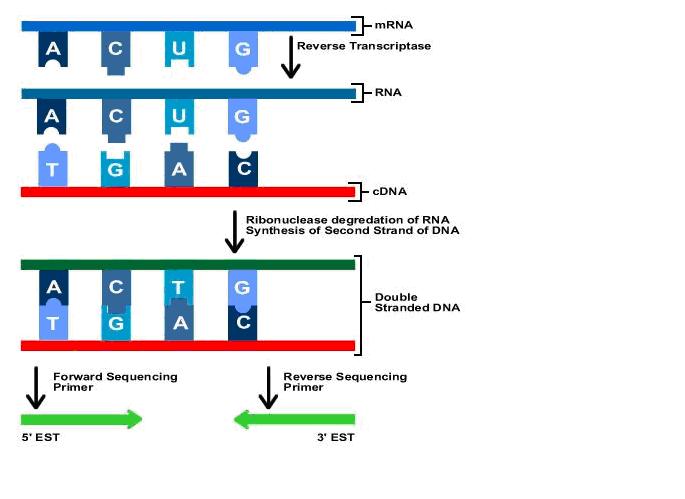
Używanie mRNA w celu wygenerowania cDNA
Identyfikacja genu jest bardzo trudna u ludzi, ponieważ większość naszego genomu składa się z intronów oddzielających sekwencje kodujące. Każdy gen (DNA) musi być zamieniony, lub przepisywany, na matrycowy RNA (mRNA), RNA, które służy jako szablon do syntezy białek. mRNA następnie prowadzi syntezę białek poprzez proces zwany transkrypcją. Co ciekawe, mRNAs w komórce nie zawierają sekwencji niekodujących między regionami kodującymi. Dlatego izolowanie mRNA jest kluczem do znalezienia interesujących nas genów ludzkiego genomu. Problem stanowi niestabilność mRNA, dlatego też naukowcy przekształcili go w komplementarny cDNA, który jest bardziej stabilny i co najważniejsze również nie posiada intronów. Pozwala to następnie na uzyskanie znaczników ESTs, które w szybki sposób umożliwią identyfikacje konkretnych genów.
Wykorzystanie ESTs umożliwia naukowcom jak najszybszą identyfikacje choroby, pozwala badać mutacje i inne zmiany jakie zachodzą w genomie, mogące mieć ujemny wpływ na pacjenta. Korzystając z tej metody, naukowcy zbadali już pojedyncze geny zaangażowane w chorobą Alzheimera, raka okrężnicy, i wiele innych chorób. Łatwo zrozumieć, dlaczego ESTs poszerza horyzonty w dziedzinie badań genetycznych.
Naukowcy utworzyli więc na NCBI nowy katalog ESTs do przechowywania i zamieszczania wyników nowych badań związanych z lokalizacją konkretnych genów oraz czasui miejsca ich uaktywniania, oraz lepszego dostępu do tych danych.
Od cDNA do ESTs
Kiedyś cDNA reprezentowało ekspresje genu, który został wyizolowany, naukowcy mieli następnie sekwencje z kilku tysięcy nukleotydów z jednego końca molekuły aby stworzyć dwa różne rodzaje ESTs. Sekwencjonowanie tylko początku fragmentu cDNA produkuje tzw. 5’EST. 5’EST uzyskane są z części transkryptu, który zwykle koduje białko. Regiony te są zwykle zachowane u wszystkich gatunków i nie zmieniają się za bardzo w obrębie rodziny genów. Sekwencjonowanie końcowego regionu cDNA produkuje tzw. 3’EST. Ponieważ te ESTs są generowane od 3’ końca transkryptu, mogą one nie zostać „przetłumaczone” lub nie regionami nie podlegającymi translacji (UTRs), a zatem na ogół wykazują mniejszą w różnych gatunkach konserwacje, niż sekwencje kodujące.
UTR jest tą częścią genu, która nie ulega translacji w białku.

ESTs są generowane przez sekwencjonowanie cDNA, które jest samo syntetyzowane z mRNA z komórki. mRNA nie zawiera sekwencji pomiędzy genami, bez intronów, które są prezentowane w wielu interesujących częściach genomu.
ESTs jako genomowi obiekty
Podobnie jak osoba prowadząca samochód może potrzebować mapy aby odnaleźć swój cel, naukowcy poszukują dla genów map genomu aby pomogły im poruszać się wśród miliardów nukleotydów, które składają się na ludzki genom. Na mapie, aby nabrała sensu poszukiwań, niezbędne jest aby posiadała wiarygodne obiekty lub „markery”. Obecnie, najpotężniejszą techniką mapowania, oraz jedyną która jest używana do generowania wielu map genomów, jest Sequence Tagged Site (STS) mapping. STS jest krótka sekwencją DNA, która jest łatwo rozpoznawalna i występuje tylko raz w genomie (albo chromosomie). 3’ESTs służy jako wspólne źródło STSs, ponieważ ich prawdopodobieństwo jest unikalne dla danego gatunku i dostarcza dodatkowych funkcji wskazując bezpośrednio do genów podległych ekspresji.
ESTs jako zasoby odkrywania genu.
ESTs są to potężne narzędzia w poszukiwaniu znanych genów, ponieważ w znacznym stopniu redukują czas potrzebny do zlokalizowania genu.
Ponieważ ESTs reprezentują kopię tylko interesującej części genomu, co wyraża się tym że, okazały się one ponownie i ponownie jako potężne narzędzia w poszukiwaniu genów zaangażowanych w dziedziczne choroby. ESTs mają także kilka praktycznych korzyści w swojej sekwencji mogą być generowane szybko i niedrogo, tylko jedno sekwencjonowanie jest potrzebne na każde wygenerowane cDNA, oraz nie muszą być one sprawdzane w przypadku błędów sekwencjonowania ponieważ błędy nie uniemożliwiają identyfikacji genów, z którego EST pochodzi.
Używając ESTs, naukowcy wyizolowali szybko kilka z genów odpowiedzialnych za chorobę Alzheimer’a oraz raka okrężnicy.
Aby znaleźć choroby genetyczne używając tej metody, naukowcy najpierw używali biologicznych, możliwych do obserwacji wskazówek do identyfikacji ESTs, które mogą współdziałać z kandydatami na choroby genetyczne. Naukowcy następnie zbadali DNA chorych pacjentów pod względem mutacji w jednym lub kilku kandydatów (genów) dla potwierdzenia tożsamości genetycznej. Korzystając z tej metody, naukowcy wyizolowali już geny zaangażowane w chorobę Alzheimer’a, raka okrężnicy i wiele innych chorób. Łatwo zrozumieć dlaczego ESTs nakreśla nową drogę do nowych horyzontów w dziedzinie badań genetycznych.
Ze względu na ich użyteczność, prędkość z jaką mogą być generowane i niskie koszty związane z technologią, wielu indywidualnych naukowców jak również wielkie centra zajmujące się sekwencjonowaniem generują setki tysięcy ESTs dla publicznego użytku. Kiedy EsT zostało wygenerowane, naukowcy składali ich cele w GenBank’u, w NIH bazie danych sekwencji obsługiwanej przez NCBI. Z szybkiego składania tak wielu ESTs, stało się trudne zidentyfikowanie sekwencji, która została już złożona w bazie danych. Było coraz bardziej oczywiste dla badaczy NCBI, że jeśli ESTs mają być łatwo dostępne i użyteczne jako narzędzie odkrywania genów, muszą oni zorganizować przeszukiwana bazę danych, która również ułatwia dostęp do innych danych genomu. Dlatego, w 1992, naukowcy NCBI opracowali nową bazę danych zaprojektowaną, aby służyć jako punkt kolekcji ESTs. Kiedy EST składało się na GenBank były one pokazywane z adnotacją, że zostały przeniesione do nowej bazy danych, nazywanej dbEST.
dbEST: Opisowy Katalog ESTs
Naukowcy stworzyli na NCBI dbEST do organizowania, przechowywania i dostępu do wielkich masowych danych EST, które zostały już nagromadzone i które rosną z dnia na dzień. Korzystając z dbEST, naukowiec ma dostęp nie tylko do danych ludzkich ESTs, ale także do informacji o EST z ponad 300 innych organizmów. Tam gdzie to możliwe, naukowcy NCBI dodali adnotacje do rekordu EST z wszelkimi znanymi informacjami. Na przykład, jeśli dopasowanie EST do sekwencji DNA, która koduje znany gen ze znaną funkcją, to nazwa genu i funkcja są umieszczone na rekordzie EST. Adnotacje rekordów EST pozwalają naukowcom publicznym używać dbEST jako drogi do odkrycia genu. Poprzez użycie narzędzia przeszukiwania bazy danych, takiego jak BLAST w NCBI, każda z zainteresowanych partii może prowadzić wyszukiwanie podobieństw sekwencji przed dbEST.
UniGene: niezbędny zestaw Grup genów zorientowanych
Ponieważ gen ulega ekspresji jako wiele mRNA, wiele razy, ostatecznie EST pochodzące z niego może być zbędne. Oznacza to, że może istnieć wiele identycznych lub podobnych kopii tego samego EST. Taka zbędność i zachodzenie na siebie oznacza, że niektóre wyszukiwanie dbEST dla pojedynczych EST, może pobrać długą listę celów, wiele z nich może reprezentować ten sam gen. Przeszukiwanie tych wszystkich identycznych EST może być bardzo czasochłonne. Aby rozwiązać ten problem, naukowcy NCBI opracowali UniGene database. UniGene to automatyczne partycje sekwencji GenBanku w niezbędnym zbiorze grup genów zorientowanych.
Choć jest powszechnie uznane, że generacja ESTs stanowi skuteczną strategię do identyfikacji genów, jest ważne aby potwierdzić, pomimo zalet, jest kilka ograniczeń związanych z podejściem EST. Jednym z nich jest bardzo trudna izolacja mRNA z niektórych rodzajów tkanek i komórek. Powoduje to niedostatek danych dotyczących niektórych genów, które mogą być tylko znalezione w tych rodzajach tkanek i komórek.
Po drugie jest to, ze ważne sekwencje regulatorowe genów mogą się znajdować w intronach. Ponieważ EST są małymi segmentami cDNA, wygenerowanymi z mRNA, w których introny zostały usunięte, mogą zostać utracone ważne informacje przez skupianie się tylko na sekwencji cDNA. Pomimo tych ograniczeń, EST nadal są nieocenione w charakteryzowaniu ludzkiego genomu, tak jak i innych organizmów. Umożliwiają one mapowanie wielu genów do miejsc na chromosomie oraz stanowią także pomoc w odkrywaniu wielu nowych genów.
Prawie wsztstkie komórki organizmu zawierają pełny zestaw chromosomów i identycznych genów. "Ekspresjia genów" to termin oznaczający transkrypcję informacji zawartych w DNA, repozytorium informacji genetycznej, w messenger RNA (mRNA), które następnie są tłumaczone na białka, które wykonują większość krytycznych funkcji komóreke Naukowcy badają rodzaju i ilości mRNA wytwarzanych przez komórki, aby dowiedzieć się, które geny ulegają ekspresji, co z kolei zapewnia wgląd na to, w jaki sposób reaguje komórka w miarę dojrzewania i potrzeb. Ekspresja genów jest bardzo złożonym i ściśle regulowanym procesem, który pozwala komórce reagować dynamicznie w odpowiedzi zarówno na środowisko naturalne i bodźce wewnętrzne.
W związku z projektem poznania ludzkiego genomu, doszło do wybuchu ilości dostępnych informacji na temat sekwencji DNA ludzkiego genomu. W związku z tym, naukowcy zidentyfikowali dużą liczbę nowych genów w tych nieznanych wcześniej sekwencji. Wyzwanie stojące obecnie przed naukowcami to znalezienie sposobu organizowania i katalogowania tej ogromnej ilości informacji w formie użytkowej. Dopiero po odkryciu funkcji nowych genów projekt poznania ludzkiego genomu będzie w pełni zrealizowany. Identyfikacja i klasyfikacja sekwencji DNA oraz informacje dotyczące funkcji tych nowych genów: pozwala na powstawaniu DNA microarray technologii. Technologia ta działa wykorzystując zdolność danej cząsteczki mRNA którą przypasowywuje do szablonu DNA, z którego pochodzi. Wykorzystując tablicę zawierającą wiele próbek DNA, naukowcy mogą określić, w jednym eksperymencie, setki czy tysiące genów w komórce poprzez pomiar ilości mRNA związanego z każdym miejscem na tablicy. Przy pomocy komputera, mRNA wiąże się do określonych plam na microarray, które można dokładnie zmierzyć, tworząc profil ekspresji genów w komórce.
Microarrays są wynikiem ogromnego postępu zarówno datlego iż mogą one zawierać bardzo dużą liczbę genów jak również ze względu na ich niewielkie rozmiary. Microarrays są zatem przydatne, gdy chce się wykonać badanie wielu genów szybko lub gdy próbka badana jest niewielka. Ponieważ microarray może być użyty do zbadania ekspresji setek czy tysięcy genów jednocześnie, umożliwia to naukowcom badać ekspresje wielu genów. Ta technologia jest wciąż we wczesnym stadium, dlatego wiele wstępnych badań wykorzystujących microarrays to proste badania profili ekspresji genów w różnych typach komórek. Nevertheless Jednakże, badania te stanowią ważny i konieczny krok w zrozumieniu i katalogowaniu ludzkiego genomu.
Im więcej informacji gromadzą naukowcy tym większe możliwości wykorzystania microarrays, stawianie nowych, coraz bardziej złożonych pytań i wykonywanie bardziej skomplikowanych eksperymentów. Naukowcy będą mogli wnioskować prawdopodobne nowe funkcji genów w oparciu o podobieństwa wzorców znanych genów. Ponieważ produkt jednego genu zwykle sprzężony jest z wieloma innymi, zrozumienie tych genów może być łatwiejsze dzięki analizom i dokładnej znajomości tych wzajemnych zależności. Wykorzystanie microarrays może również przyspieszyć identyfikację genów zaangażowanych w rozwój różnych chorób poprzez umożliwienie naukowcom zbadanie znacznie większej liczby genów. Technologia ta będzie również pomocą w integracji badań ekspresji genów i funkcji na poziomie komórkowym, ujawniając, jak wiele produktów genów wspólnie wpływają na fizyczne i chemiczne reakcje w miarę zmieniających się potrzeb komórkowych.
DNA microarrays są to małe, solidne wsparcia, w których sekwencja z tysiąca różnych genów jest unieruchomiona lub przyczepiona, na stałych lokacjach. Ich wsparciem są zwykle szklane mikroskopowe slajdy, wielkości dwóch palców, ale mogą być to także silikonowe chip’y lub nylonowe membrany. DNA jest odbijany, robi odciski, lub aktualnie syntetyzowany bezpośrednio na wsparcie.
American Heritage Dictionary definiuje „array” jako miejsce uporządkowane. Ważne jest, aby sekwencja genu w mikromacierzy, była przywiązana do swojego wsparcia w sposób uporządkowany, lub na stałe, ponieważ naukowiec używa lokalizacji w każdym miejscu macierzy do identyfikacji poszczególnych sekwencji genów. Miejsca-plamy w mikromacierzy mogą stanowić DNA, cDNA albo oligonukleotydy.
Oligonukleotydy, lub „oligo” – jak jest powszechnie nazywane, jest to krótki fragment jednoniciowego DNA, które typowo składa się z 5-50 nukleotydów.
Można by zapytać, w jaki sposób naukowiec uzyskuje informacje na temat rozwoju choroby z tak małej wielkości szkła albo silikonowego chip’u zawierającego tysiące indywidualnych sekwencji genów? Cały proces jest oparty na hybrydyzacji, technice, która wykorzystuje znaczniki fluorescencyjne będące cząsteczkami kwasu nukleinowego, jako "mobilną sondę" do identyfikacji cząsteczek uzupełniających, sekwencje, które są w stanie się parować na zasadzie komplementarności. Każdy fragment DNA składa się z czterech różnych nukleotydów, adeniny (A), tyminy (T), guaniny (G) i cytozyny (C). Adenina tworzy zawsze parę z tymina, natomiast guanina z cytozyną. Dlatego też uzupełnieniem sekwencji GTCCTA będzie CAGGAT. Gdy dwie komplementarne sekwencje się odnajdą, tak jak immobilizowane DNA i mobilna próba DNA, cDNA albo mRNA, to zostaną one zamknięte lub hybrydyzowane.
Teraz, dwa typy komórek: typ 1, komórka zdrowa, typ 2, komórka chora. Obie zawierają identyczny zestaw czterech genów A, B, C i D. Naukowcy są zainteresowani określeniem profilu ekspresji tych czterech genów w tych dwóch typach komórek. Aby to osiągnąć, naukowcy izolują mRNA z każdego typu komórki i używają tego mRNA jako próby do uzyskania cDNA z przyłączonym znacznikiem fluorescencyjnym. Różne znaczniki (czerwony albo zielony) są używane, więc próby będą zróżnicowane w kolejnych krokach. Obie oznakowane próby są następnie mieszane i inkubowane z mikromacierzą zawierającą immobilizowane geny A, B, C i D. Oznakowanie wiązania cząsteczek do miejsca na tablicy, współdziała ekspresji genów w każdej z komórek.
Po tym kroku hybrydyzacja jest kompletna, a pracowanik umieszcza mikromacierz w „Odczytywaczu” albo „Skanerze”, który zawiera różne lasery, specjalny mikroskop i kamerę. Znaczniki fluorescencyjne zostają pobudzone przez laser, a mikroskop i kamera współpracują aby stworzyć cyfrowy obraz macierzy. Dane te są następnie zapisywane na komputerze, a specjalny program oblicza stosunek intensywności fluorescencji czerwonego do zielonego, albo odejmuje dane tła dla każdego miejsca na mikromacierzy przez analizę cyfrową obrazu. Jeśli zostaną obliczone wskaźniki, program tworzy następnie tabelę, która zawiera poziom intensywności czerwono do zielonej fluorescencji dla każdego miejsca na macierzy. Na przykład, korzystając z opisanej powyżej sytuacji, komputer może stwierdzić, że oba typy komórek ujawniają ekspresję genu A, na tym samym poziomie, że w komórce pierwszej gen B ulega większej ekspresji, że w komórce 2 większej ekspresji ulega gen C, że komórka 2, wyraża więcej genów C oraz, że nie wyraża ona genu D. Należy jednak pamiętać, że jest to prosty przykład użyty do zobrazowania kluczowych punktów w projektowaniu eksperymentów. Niektóre mikromacierze mogą zawierać nawet do 30 000 docelowych miejsc. Dlatego dane uzyskane z jednej mikromacierzy mogą rosnąć szybko.

Na schemacie:
Zielony reprezentuje kontrolne DNA, gdzie DNA albo cDNA pochodzące z normalnej tkanki poddane zostało hybrydyzacji z docelowym DNA.
Czerwony reprezentuje próbki DNA, gdzie DNA lub cDNA pochodzi z chorej tkanki i zostało poddane hybrydyzacji z docelowym DNA.
Żółty reprezentuje kombinację kontroli i DNA docelowego, gdzie oba hybrydyzowały jednakowo z docelowym DNA.
Czarny stanowi obszary, gdzie ani w kontroli ani w docelowym DNA nie zaszła hybrydyzacja z docelowym DNA.
Każde miejsce na macierzy jest związane z poszczególnym genem. Każdy kolor w macierzy reprezentuje zdrową (kontrola) lub niezdrową (próba) tkankę. W zależności od rodzaju użytej matrycy, miejsce i intensywność koloru powie nam coś gdzie jest gen, jakich mutacji albo czy jest on obecny w kontroli/ albo w próbce DNA. Zapewni on również oszacowanie poziomu ekspresji genów w próbce i kontroli.
Istnieją trzy podstawowe rodzaje prób, które mogą zostać użyte do zbudowania mikromacierzy DNA, dwa są genomu a inne są „transkryptami”, które są miarą poziomu mRNA. Co czyni je różnymi od siebie, to rodzaj immobilizowanego DNA użytego do wytworzenia macierzy oraz, ostatecznie, rodzaj informacji, który dociera z chip’u. Docelowe DNA determinuje także typ kontroli i próby DNA, który jest użyty w hybrydyzacji.
1. Zmiany w poziomie ekspresji genów
Ustalenie poziomu, albo objętości, w którym pewne geny ulegają ekspresji nazywane jest analizą ekspresji mikromacierzy, a macierze użyte w tym rodzaju analizy nazywane są chip’y ekspresji. Immobilizowane DNA jest cDNA dostarczanym z mRNA znanych genów, i po raz kolejny, przynajmniej w niektórych przypadkach, kontrola i próba hybrydyzujące do chip’a są cDNA dostarczone z mRNA z normalnych i chorych tkanek Jeśli gen ulega wzmożonej ekspresji w niektórych chorych tkankach, niż próba cDNA, po porównaniu z kontrolą, to hybrydyzacja zajdzie w miejscu reprezentującym ten gen. Odwrotnie, na miejscu fluorescencji czerwonej z większą intensywnością niż fluorescencja zielona. Kiedy naukowcy charakteryzują ekspresję różnych genów zaangażowanych w wiele chorób, cDNA pochodzące z tkanek chorych może ulegać hybrydyzacji aby ustalić, czy poziom ekspresji genu z indywidualnych prób dopasować do poziomu ekspresji znanych chorób. W takim przypadku, leczenie dla tej choroby może zostać rozpoczęte.
Jak naukowcy używają ekspresji chip’ów do detekcji próby ekspresji – czy poszczególne geny ulegają ekspresji w większym lub mniejszym stopniu w pewnym okolicznościach – ekspresja chip’ów może być również użyta do sprawdzenia zmian w ekspresji genów w danym okresie czasu, tak samo jak w czasie cyklu komórkowego. Cykl komórkowy jest molekularną siecią, która określa, w normalnej komórce, czy komórka powinna przechodzić przez jego cykle życia. Istnieje wiele różnych genów uczestniczących w regulacji etapów życia komórki. Ponadto wbudowany w tą sieć mechanizm zaprojektowany do ochrony ciała, gdy ten system zawodzi albo upada przez mutacje w jednym z „genów kontroli”, tak samo jak w przypadku wzrostu komórek nowotworowych. Ekspresja mikromacieży „eksperymentu” może być zaprojektowane, gdy cykl komórki jest generowany w wielu macieżach i odwołuje się do czasu „zero”. Analiza zebranych danych może doprowadzić do dalszego wyjaśnienia szczegółów cyklu komórkowego oraz jego „zegar”, dostarczając wiele niezbędnych danych w punkcie, w którym mutacje genów prowadzą do wzrostu nowotworowego tka samo jak źródła terapeutycznego.
W ten sam sposób, ekspresja chip’ów może zostać wykorzystana do tworzenia nowych leków. Na przykład, jeśli pewien gen ulega nadmiernej ekspresji w poszczególnych formach raka, naukowcy mogą używać ekspresji chip’ów, aby zobaczyć czy nowy lek zredukuje tą nadmierną ekspresję i zmusi raka do zmniejszenia. Ekspresja chip’ów może być również użyta do diagnozy choroby tak jak, na przykład w identyfikacji nowych genów zaangażowanych wywołujących choroby, tak jak choroby układu immunologicznego, nerwowego i płuc/układu oddechowego.
2.Genetyczne zyski i straty
Naprawa genów DNA jest pierwszą linią obrony organizmu przeciwko mutacjom oraz, tak jak, odgrywa główną role w rozwoju raka. Mutacje w obrębie tych genów często pojawiają się jako utracone lub uszkodzone chromosomy. Stwierdzono, że niektóre zyski i straty chromosomów związane są z progresją nowotworu oraz, że próby tych zmian są istotne ze względu na kliniczne rokowania. Używając różnych metod laboratoryjnych, naukowcy mogą mierzyć zyski i straty w liczbie kopii regionów chromosomalnych w komórkach guza. Następnie, używając matematycznych modeli do analizy tych danych, mogą przewidzieć które regiony chromosomów najprawdopodobniej odpowiadają za inicjację guza i rozwój choroby. Wyniki takiej analizy mogą być przedstawione jako hierarchiczne diagramy odgałęzień drzewa, określonego jako „drzewo modeli progresji nowotworów”
3.Mutacje w DNA
Kiedy naukowcy używają mikromacierzy do wykrywania mutacji albo polimorfizmu w sekwencji genu, docelowy albo immobilizowany DNA jest zazwyczaj pojedynczym genem. W tym przypadku jednak, docelowa sekwencja wprowadzona w danym miejscu macierzy będzie się różniła od innych miejsc w tej samej mikromacierzy, czasami przez jeden lub kilka specyficznych nukleotydów. Jeden rodzaj sekwencji powszechnie stosowany w tym typie analizy nazywany jest polimorfizmem pojedynczego nukleotydu lub SNP, małe zmiany genetyczne lub zmiany, które mogą wystąpić w sekwencji DNA poszczególnej osoby. Kolejna różnica w analizie mutacji mikromacierzy, w porównaniu do ekspresji albo CGH mikromacierzy, jest taka że eksperyment wymaga tylko genomowego DNA dostarczonego z normalnej próby do użycia w miksturze do hybrydyzacji.
Kiedy naukowcy stwierdzili ze wzór SNP jest związany z daną chorobą, mogą oni użyć technologii mikromacierzy SNP do indywidualnych testów na tę chorobę wzoru ekspresji do określenia czy on lub ona jest podatny na (na ryzyko rozwoju) tą chorobę. Gdy genomowe DNA od osoby indywidualnej podlega hybrydyzacji do macierzy zawierającej różne SNP, próba DNA będzie hybrydyzować z większą częstotliwością tylko do specyficznych SNP związanych z tą osobą. Te miejsca w mikromacierzy będą później fluoryzować z większą intensywnością, wskazując, z że dana osoba podlegająca testowi może posiadać lub jest w grupie ryzyka choroby.
Aplikacje mikromacierzy – w skrócie
Typ - aplikacja:
CGH – Klasyfikacja guza, ocena ryzyka i przewidywania prognozy
Sposób analizy – opracowywanie leków, odpowiedź na leki i rozwój terapii
Mutacja/ polimorfizm analizy – opracowywanie leków, rozwój terapii i śledzenie progresji choroby
Dlaczego konieczne jest ujednolicenie systemu, który by zarządzał i stanowił punkt wydawania danych mikromacierzy? Rozważmy ilości danych, które potencjalne mogą być generowane za pomocą jednego chip’u mikromacierzy. Zakłada się, że chip zawiera 30 00 punktów docelowego DNA. Naukowcy interpretują dane uzyskane przez to, że chip musi znać biologiczną tożsamość każdego celu – który gen jest gdzie; biologiczne właściwości kontroli i prób DNA; eksperymentalne warunki i procedury używane do tworzenia eksperymentu; i w końcu wyniki. Choć eksperymenty takie jak te, będą niewątpliwie szły naprzód nasze obecne zrozumienie ekspresji genu i regulacji, wiele nowych wyzwań jest w zakresie monitorowania i analizy danych.
Technologia mikromacierzy jest jedną z najnowszych i najważniejszych doświadczalnych odkryć w biologii molekularnej. Dziś, biegłość w tworzeniu danych szybko przezwycięża zdolność do przechowywania i analizowania go. Wiele z tych informacji jest rozsiana po całym Internecie albo nie jest jeszcze publicznie dostępna. Ponieważ większość laboratoriów chce nabyć tą technologię, problem będzie się tylko rozwijał. Ta lawina informacji wymaga standaryzacji przechowywania, udostępnienia i opublikowania technik.
W celu wsparcia publicznego wykorzystania i upowszechnienia danych ekspresji genów, NCBI założył Gene Expression Omnibus – GEO. GEO reprezentuje starania NCBI do budowy repozytorium danych ekspresji oraz źródło online dla przechowywania i wyszukiwania danych ekspresji genu z każdego organizmu lub sztucznego źródła. Miele danych ekspresji genów, takie jak te omówione w tym rozdziale, są akceptowane i zarchiwizowane jako publiczne dane.
Podniesiony Język Mikromacierzy, opracowany przez MAML, grupy roboczej MGED, Microarray Gene Expression Database, jest pierwszą próbą wprowadzenia standardowych podstaw do składania i analizy ogromnych ilości danych ekspresji mikromacierzy uzyskanych przez różne laboratoria z całego świata. Celem tej grupy, która zawiera naukowców NCBI, jest ułatwienie przyjęcia standardów dla eksperymentów DNA-macierzy i reprezentacji danych, jak również wprowadzenie standardowych kontroli i danych metod normalizacji. Podstawowym celem jest ułatwienie tworzenia danych repozytorium ekspresji genów, porównywalności danych ekspresji genów z różnych źródeł, interpelacyjności różnych baz danych ekspresji genów i oprogramowania analizy danych.
MAML proponuje ramy do opisu informacji o DNA-macierzy i systemu danych przekazywania tych informacji, zawierających szczegóły na temat:
Projektowania eksperymentu: zestaw eksperymentów hybrydyzacji przedstawiony jako całość
Zaprojektowanie macierzy: każda używana macierz i każde używane miejsce w macierzy
Próby: używane próby, przygotowanie ekstraktu i oznakowanie
Hybrydyzacja: procedury i parametry
Pomiary: obrazów, ilościowe i specyfikacji
Sterowanie: rodzaje, wartości i specyfikacje
MAML jest niezależny od poszczególnych eksperymentalnych platform i prowadzi do ram dla opisania wykonanego eksperymentu wszystkich typów macierzy DNA, wliczając „plamowe” i te do syntezy, zarówno oligo i cDNA macierze. Co więcej, MAML dostarcza format do reprezentacji danych mikromacierzy w elastyczny sposób, który pozwala danym analizowanym uzyskanym z nie tylko istniejących mikromacierzy ale również wielu możliwych wariantów, wliczając macierze białkowe. Choć dane w GEO nie są aktualnie wprowadzane w formacie MAML , jest to celem NCBI by dane dostarczone w wielu formatach, wliczając MAML, niedługo były zastąpione nowszą wersją nazywaną MAGEML (MicroArray Gene Expression Markup Language).
Korzyści wynikające z GEO i MAML
- przez przechowywanie dużej ilości danych na temat profili ekspresji genów pochodzących z wielu eksperymentów używanie różnych kryteriów i warunków, GEO będzie pomocne w badaniu genomiki funkcjonalnej – rozwój i zastosowanie metod doświadczalnych globalnej oceny funkcji genów
- GEO ułatwi wzajemne walidacje danych uzyskane przez różne techniki i technologie oraz będzie pomocne w zestawieniu norm i standardów dla dalszych badań ekspresji genów
- dzięki informacjom zamieszczonym i publicznie dostępnym w GEO, dziedziny bioinformatyki i genomiki funkcjonalne będą promowane i rozwijane
- takie dane eksperymentalne powinny być swobodnie dostępne dla wszystkich, co zgodne jest z mandatem legislacyjnym i misją NCBI: aby rozwijać nowe techniki informacji by pomóc w zrozumieniu fundamentalnych molekularnych i genetycznych procesów, które są odpowiedzialne za ochronę zdrowia i choroby
Teraz, kiedy rozumiemy koncepcję technologii macierzy, wyobraźmy sobie: mamy instrument, który lekarz może użyć do szybkiej diagnozy raka albo innych chorób podczas standardowej wizyty. Co jeśli ten sam instrument mógłby również ułatwić leczenie indywidualne, dokładnie dla Ciebie? Indywidualne leki. Diagnostyka molekularna. Integracja diagnostyki i terapii. Są to długoterminowe obietnice technologii mikromacierzy. Może nie dziś czy jutro, ale pewnego dnia. Po raz pierwszy, macierze oferują nadzieję na uzyskanie globalnych poglądów biologicznych procesów – jednocześnie biorąc pod uwagę wszystkie składniki organizmu – przez wprowadzenie systematycznego sposobu na badanie zmienności DNA i RNA.
NCBI, przez kontynuowanie swoich wysiłków do wprowadzenia standardowego formatu dla danych mikromacierzy i wprowadzenia darmowego, dostępu do tych danych, pomoże społeczności naukowej aby urzeczywistnić te obietnice.
Komórki są strukturalne i funkcjonalne jednostki wszystkich żywych organizmów. Niektóre organizmy, takie jak bakterie, są jednokomórkowe, składający się z pojedynczej komórki. Inne organizmy, takie jak człowiek, są wielokomórkowe, lub mają wiele komórek, szacuje, że mają 100000000000000 komórek! Każda komórka jest niesamowity świat w sobie: potrafi przyswajać składniki odżywcze, przetwarzać je w energie, przeprowadzać wyspecjalizowane funkcje i, w razie potrzeby, reprodukować się (odtwarzac). Jeszcze bardziej niesamowite jest to, iż każda z tych komórek gromadzi informacje niezbędne do wykonywania każdej z tych czynności.
Organizacja komórki
Zanim zaczniemy dyskutować o różnych składnikach komórki, ważne jest, aby wiedzieć, skąd pochodzą komórki organizmu. Istnieją dwie ogólne kategorie komórek: prokariotach i eukarioty.
Rysunek 1. Historia życia na Ziemi.
Prokariotyczne organizmy
Wydaje się, że życie na Ziemi powstało około 4 miliardów lat temu. Najprostsze komórki i pierwsze typy komórek mogły ewoluować, były to komórki organizmów prokariotycznych, w których brak jest jądrowej membrany, która otacza jądro komórki. Bakterie są najbardziej poznaną i najbardziej zbadaną formą organizmów prokariotycznych, choć niedawne odkrycie drugiej grupy prokariotów, zwaną archaea, przedstawiły dowody trzeciej komórkowych domeny życia i nowe spojrzenie na pochodzenie życia.
Prokarioty są organizmami jednokomórkowymi, które nie tworzą lub nie różnicują się w formy wielokomórkowe. Niektóre bakterie rosną w filamentach lub masie komórek, ale każda komórka w kolonii jest identyczna i zdolne do niezależnego istnienia. Komórki mogą być obok siebie, ponieważ nie są one oddzielne po podziale komórkowym lub ponieważ pozostają zamknięte we wspólnej otoczce lub złączone śluzem wydzielanym przez komórki. Zazwyczaj jednak nie ma ciągłości lub komunikacji pomiędzy komórkami. Prokarioty są zdolne do zamieszkiwania niemal każdego miejsca na ziemi, od głębokiego oceanu, na krawędziach gorących źródeł, aż do powierzchni naszego ciała. br />
Prokarioty można odróżnić od eukarioty na podstawie organizacji jądrowej, a konkretnie ich braku jądrowej membrany. Prokarioty cechuje także brak któregokolwiek z wewnątrzkomórkowych organelli i struktur, które są charakterystyczne dla komórek eukariotycznych. Większość funkcji organelli, takich jak mitochondria, chloroplasty oraz aparat Golgiego, zostały przejęte przez prokariotyczną membranę plazmy. Prokariotycznych komórek mają trzy regiony architektoniczne: dodatki nazywane flagella i pili - białka dołączone do powierzchni komórki; okrycie komórki składające się z kapsułki, ściany komórkowej, a membrany plazmatycznej, oraz cytoplazmatycznego regionu, który zawiera genom komórkowy (DNA) oraz rybosomy i różnego rodzaju dodatki.
Organizmy eukariotyczne
Eukarioty obejmują grzybów, zwierzęta i rośliny, jak również niektóre jednokomórkowe organizmy. Komórki eukariotyczne są około 10 razy mniejsze od prokariotów i może być aż 1000 razy większej objętości. Główną i niezwykle istotną różnicy między prokariotach i eukarioty jest to, że komórki eukariotyczne zawierają membrany - granice przedziałów, w których zachodzą szczególne metaboliczne działania. Najważniejszą wśród nich jest obecność w jądro, membrany-ograniczającej miejsce, w którym mieści się w DNA komórek eukariotycznych. Jest to jądro, które daje eukariontom – dosłownie, prawdziwe jądro-jego nazwę.
Organizmy eukariotyczne mają także inne wyspecjalizowane struktury, zwane organellami, które są małymi strukturami komórek, które wykonują określone funkcje. Jak sama nazwa wskazuje, można pomyśleć o organelli jak o małym narządzie. Istnieje kilkanaście różnych typów organelli powszechnych w komórkach eukariotycznych. Na ten przykład, będziemy koncentrować naszą uwagę tylko na garść organelli i zbadamy te organelle z punktu widzenia ich roli na poziomie molekularnym w komórce.
Pochodzenie z komórek eukariotycznych był kamieniem milowym w ewolucji życia. Chociaż eukarioty używać tego samego kodu genetycznego i procesów metabolicznych jak prokarioty, ich wyższy poziom złożoności organizacyjnej pozwolił na rozwój prawdziwie organizmów wielokomórkowych. Bez eukariotów, świat byłby bez ssaków, ptaków, ryb, bezkręgowców, grzybów, roślin i skomplikowanych organizmy jednokomórkowych.
Rysunek 2. Eukarionty i prokarioty.
Ten obrazek pokazuje typową ludzką komórkę (eukariotyczną) i typową komórkę bakterii (prokariotyczną). Rysunek po lewej podkreśla wewnętrzne struktury komórek eukariotycznych, w tym jądro (jasnoniebieski), w jąderko (pośredni niebieski), mitochondria (pomarańczowy) oraz rybosomy(ciemnoniebieski). Rysunek po prawej pokazuje, w jaki sposób DNA bakterii mieści się w struktury zwane nukleoidem (bardzo jasny niebieski), jak również inne struktury zwykle obecne w prokariotycznych komórkach, w tym komórek membrany (czarny), ściany komórkowe (pośredni niebieski), kapsuły (pomarańczowy), rybosomy (ciemny niebieski), oraz flagellum (również czarny).
Membrany plazmatyczne –płaszcz ochronny komórki
Zewnętrzna ochrona komórek eukariotycznych nazywa się membraną plazmatyczną. Membrana ta służy do rozdzielenia i ochrony komórki od otaczającego środowiska i jest wykonana głównie z podwójnej warstwy białek i lipidów, tłuszczy. W tej membranie osadzone są różne inne cząsteczki, które działają, jako kanały i pompy, powodując, że inne cząsteczki są wprowadzone do komórki lub z niej wyprowadzone. Forma membrany plazmatycznej jest również w prokariotach, ale w tym organizmie jest zazwyczaj określane, jako membrana komórkowa.
Cytoszkielet—rusztowanie komórki
Cytoszkielet jest ważnym, złożonym i dynamicznym składnikiem komórek. Działa on tak, aby zorganizować i utrzymać kształt komórki; zakotwicza organelle w miejscu; pomaga podczas endocytozy, wykorzystaniu zewnętrznych materiałów przez komórkę i przenosi części komórek w procesach wzrostu i ruchliwość. Istnieje wiele białek związanych z cytoszkieletem, każda kontroluje struktury komórki przez właściwości kierujące, pakujące i dostosowujące włókna.
Cytoplazma – wewnętrzna przestrzeń komórki
Wewnątrz komórek znajduje się duża przestrzeń wypełniona płynem o nazwie cytoplazma, czasami nazywana cytosolem. W prokariotów przestrzeń ta jest stosunkowo wolna od przedziałów. W eukariotów w cytosolu jest "zupa", w której znajdują się wszystkie organelle komórki. Jest to również siedziba cytoszkieletu. Cytozol zawiera rozpuszczone składniki odżywcze, pomaga rozbić odpady produktów i porusza materiałem wokół komórki za pomocą procesu zwanego cytoplazmatycznymi strumieniowej. Jądro często pływa z cytoplazmą zmienia swój kształt, ponieważ się porusza. Cytoplazma również zawiera wiele soli i jest doskonałym przewodnikiem elektrycznym, tworząc idealne środowisko dla mechaniki komórki. Funkcje cytoplazmy i organelli, które mieszkają w niej, mają krytyczne znaczenie dla przetrwania komórki.
Materiał genetyczny
Istnieją dwa różne rodzaje materiału genetycznego: kwas deoksyrybonukleinowy (DNA) i kwas rybonukleinowy (RNA). Większość organizmów jest zbudowana z DNA, ale jest kilka wirusów, które posiadają RNA, jako ich materiał genetyczny. Biologiczna informacja zawartych w organizmie jest zakodowana w sekwencji DNA lub RNA.
Co ciekawe, aż 98 procent ludzkiego DNA nie koduje konkretnego produktu.
Prokariotyczny materiał genetyczny jest organizowany w proste, koliste struktury spoczywające w cytoplazmie. Eukariotyczny materiał genetyczny jest bardziej skomplikowany i jest podzielony na dyskretne jednostki zwane genami. Ludzkiego materiału genetyczny składa się z dwóch odrębnych części: genomu jądrowego i mitochondrialnego. Genom jądrowy jest podzielony na 24 liniowe cząsteczki DNA, każda zawarta w różnym chromosomie. Genom mitochondrialny jest okrągłym DNA oddzielonym od jądrowego DNA. Chociaż mitochondrialnego genomu jest bardzo mało, to koduje on niektóre bardzo ważne białka.
Organelle
Ciało ludzkie zawiera wiele różnych organów, takich jak serce, płuca i nerki, każdy z nich wykonuje różne funkcji. Komórki również zawierają zestaw "mały organów", zwany organellami, które są dostosowywane i / lub wyspecjalizowane do przeprowadzania jednej lub więcej funkcji życiowych. Organelle występują tylko w eukariotów i zawsze otoczony są ochronną membraną. Ważne jest, aby znać kilka podstawowych faktów na temat następujących organelli.
Jądro komórki – Jej Centrum
Jądro jest najbardziej wyrazistą organellą znalezioną w eukariotycznej komórce. Są to domy na komórkowe chromosomy i są one miejscem, gdzie występuje prawie każda replikacja DNA i syntezy RNA. Jądro jest w kształcie sferoida i oddzielone jest od cytoplazmy przez membranę o nazwie okrycie jądrowe. Jądrowe okrycie izoluje i chroni komórki DNA od różnych cząsteczek, które mogłyby przypadkowo uszkodzić jego strukturę lub ingerować w jego przetwarzanie. Podczas przetwarzania, DNA jest przepisywane lub syntetyzowane, w specjalne RNA, zwane mRNA. Ten mRNA jest transportowany z jądra, gdzie jest ona tłumaczona na konkretną cząsteczki białka. W prokariotów DNA odbywa się to w cytoplazmie.
Rybosomy – maszyny produkujące białka
Rybosomy znajdują się zarówno u prokariotów i eukariotów. Rybosomy są dużym kompleksem składającym się z wielu cząsteczek, w tym RNAs i białek, i są odpowiedzialne za przetwarzanie instrukcji genetycznych przeprowadzonych z mRNA. Proces przekształcania kodu genetycznego mRNA do dokładnej sekwencji aminokwasów, które tworzą białka nazywany jest tłumaczeniem - translacją. Syntezy białek jest niezwykle ważna dla wszystkich komórek, a tym samym duża liczba rybosomów - czasem nawet setki tysięcy, można znaleźć w całej komórki.
Rybosomy unoszą się swobodnie w cytoplazmie lub czasami wiążą się z innym organellami zwanymi endoplazmatycznym retikulum. Rybosomy składają się z jednej dużej i jednej małej podjednostki, każda o innej funkcji w trakcie syntezy białek.
Mitochondria i Chloroplasty - Agregaty prądotwórcze
Mitochondria są samo-replikującymi się organellami, które występują w różnej liczbie, kształcie i rozmiarach w cytoplazmie wszystkich komórek eukariotycznych. Jak wspomniano wcześniej, mitochondria zawierają własny genom, który jest oddzielony i odrębny od jądrowego genomu komórki. Mitochondria mają dwie odrębne funkcjonalnie membrany rozdzielonych przestrzenią: zewnętrzna membrana, która otacza całą organelle, wewnętrzna membrana, która jest kształtowana w fałdy lub półki, dając projekt wnętrza. Te wewnętrzne fałdy nazywane są grzebieniami. Liczba i kształt grzebieni w mitochondriach może się różnić, w zależności od tkanki organizmu, w którym znajdują, i służą do zwiększenia powierzchni membrany.
Mitochondria odgrywają kluczową rolę w wytwarzaniu energii w komórkach eukariotycznych, a proces ten obejmuje wiele złożonych ścieżek. Rozbijmy każdy z tych etapów, dzięki czemu można lepiej zrozumieć, jak żywność i składniki odżywcze są przekształcane w energię i wodę. Niektóre z jedzenia jakie jemy są najlepszymi dostarczycielami energi, zawierają one cukry złożone. Te złożone cukry można podzielić na mniej chemicznie skomplikowaną cząsteczkę cukru o nazwie glukozy. Glukoza może następnie zostać wprowadzona do komórki za pomocą specjalnych cząsteczek znalezionych w membranie, nazywanych transporterami glukozy. Wewnątrz komórek glukoza jest rozbijana, aby utworzyć trójfosforanu adenozyny (ATP), formę energii, dzieje się to za pośrednictwem dwóch różnych ścieżek.
Pierwsza droga, glikoliza, nie wymaga tlenu i jest określana, jako beztlenowy metabolizm. Glikoliza zachodzi w cytoplazmie poza mitochondriami. Podczas glikolizy, glukoza jest dzielona na cząsteczki zwane pirogronianem. Każda reakcja jest przeznaczony do produkcji jonów wodoru, które mogą być następnie wykorzystywane do wytwarzania energii, jako ATP. Jednak tylko cztery cząsteczki ATP mogą być wykonane z jednej cząsteczki glukozy w tej drodze. U prokariotów glikoliza jest tylko metodą stosowaną do konwersji energii.
Drugi szlak, zwany cyklem Krebsa lub kwasu cytrynowego występuje wewnątrz mitochondriów i jest zdolne do wytwarzania takiej ilości ATP, która wystarczy do uruchomienia wszystkich funkcji komórek. Po raz kolejny cykl rozpoczyna się od cząsteczki glukozy, które w trakcie procesu glikolizy jest pozbawiana niektórych jej atomów wodoru, następuje przekształcanie glukozy do dwóch cząsteczek kwasu pirogronowego. Dalej, kwas ten zmieniony jest przez usunięcie węgla oraz dwóch tlenów, które uwalniają się w postaci dwutlenku węgla. Kiedy dwutlenek węgla jest usuwany, energia jest wyłączana, a cząsteczka o nazwie NAD + jest przekształcana w wyższe formy energii, NADH. Kolejna cząsteczka, koenzym A (CoA), następnie przywiązuje do pozostałych jednostkę acetylotransferazy, tworząc acetylo-CoA.
Acetylo-CoA wchodzi do cyklu Krebsa przyłączając się do cztero-węglowej cząsteczki nazywanej szczawiooctanem. Gdy przyłączą się dwie cząsteczki, powstaje sześciu-węglowa cząsteczka o nazwie kwas cytrynowy. Kwas cytrynowy jest rozbijany i modyfikowany w sposób stopniowe. W takiej sytuacji, jony wodoru i węgla zostają uwolnione. Cząsteczek węgla jest wykorzystywane do zwiększenia emisji dwutlenku węgla. Jony wodoru są zabierani przez NAD i inną cząsteczkę o nazwie FAD. Ostatecznie proces produkuje cztero-węglowy szczawiooctan ponownie, kończąc się tam, gdzie został on wyłączony. Ogólnie biorąc, cykl Krebsa jest zdolny wygenerować od 24 do 28 cząsteczek ATP z jednej cząsteczki glukozy przekonwertowanej do pirogronianu. Dlatego łatwo jest sprawdzić, ile energii można uzyskać z cząsteczki glukozy, jeśli nasze mitochondria działają prawidłowo i jeśli mamy tlen.
Chloroplasty są podobne do mitochondriów, ale znajdują się tylko w roślinach. Obie organelle jest otoczone podwójną membraną z przestrzenią miedzy nimi; mają własne DNA i są zaangażowane w metabolizm energii i mają wypuklenia lub wiele fałd, które wypełniają ich wewnętrzną przestrzeń. Chloroplasty dokonują konwersji energii świetlnej ze słońca poprzez proces fotosyntezy.
Retikulum Endoplazmatyczne i Aparat Golgiego - makromolekularni menedżerowie
Retikulum endoplazmatyczne (ER) jest siecią transportową dla cząsteczek ukierunkowaną na pewne zmiany i zajmującą szczególne miejsca, w porównaniu do molekuł, które będą unosić się swobodnie w cytoplazmie. ER ma dwie formy: szorstkie ER i gładkie ER. Szorstkie ER jest oznaczone, jako takie, ponieważ ma rybosomy przylegające do jego zewnętrznej powierzchni, mając na uwadze, że gładkie ER ich nie ma. Translacja mRNA dla tych białek, które albo zostają w ER lub zostać wyprowadzone (przeniesiony z komórki) występuje na rybosomach występujących na szorstkim ER. Gładkie ER służy, jako odbiorca tych białek syntezowanych na szorstkim ER. Białka, które mają być wyprowadzone są przekazywane do aparatur Golgiego, który czasami nazywany jest ciało Golgiego lub kompleks Golgiego, gdzie dalej jest przetwarzane, pakowane i transportowane do wielu innych miejsc komórkowych.
Aparat Golgiego został po raz pierwszy opisany w 1898 roku przez włoskiego anatomistę Camillo Golgi.
Lizosomy i Peroksysomy – komórkowy system trawienny
Lizosomy i peroksysomy są często określane jako system do wywozu śmieci komórkowych. Oba są organellami sferycznymi, otoczone przez pojedynczą membranę i bogate w enzymy trawienne, białka występujące w naturze, które przyspieszają procesy biochemiczne. Na przykład, lizosomy mogą zawierać więcej niż trzy tuziny enzymów dla degradacji białek, kwasów nukleinowych i niektórych cukrów nazywanych polisacharydy. Wszystkie te enzymy działają najlepiej w niskim pH, zmniejszając ryzyko, że te enzymy będą trawiły własne komórki gdyby w jakiś sposób wydostały się z lizosomów. Tutaj możemy zobaczyć znaczenie przedziałów komórek eukariotycznych. Komórka nie może zawierać takich enzymów niszczących jeśli nie zostały one zawarte w system otoczony membraną.
Co to jest pH?
Określenie pH wynika z połączenia "p" jako wyraz power - moc i "H" na symbol pierwiastka wodoru. pH jest to ujemny logarytm aktywności jonów wodoru i stanowi "aktywność" jonów wodorowych w roztworze w danej temperaturze. Pojęcie aktywności jest stosowane, ponieważ pH odzwierciedla ilość dostępnych jonów wodoru, a nie stężenie jonów wodorowych. PH roztworów wodnych jest w skali od 0 do 14 jednostek pH, pH 7 jest neutralne. PH poniżej 7 oznacza, że roztwór jest kwaśny, a pH powyżej 7 oznacza, że jest on zasadowy.
Gdzie pasują wirusy?
Wirusy nie są klasyfikowane jako komórki, a tym samym nie są ani jednokomórkowymi organizmami ani wielokomórkowymi. Większość ludzi nawet nie klasyfikuje wirusów jako "żywych", ponieważ brak jest w nich systemu metabolicznego i są one zależne od komórek gospodarza, które zainfekowały do reprodukcji. Wirusy mają genomy, które składają się zarówno z DNA lub RNA, a istnieją przykłady wirusy, które mają albo jedno- lub dwułańcuchowe. Co ważne, ich genomy kodują nie tylko białka potrzebne do pakowania jego materiału genetycznego, ale te białka potrzebne do reprodukcji wirusa w czasie jego cyklu infekcyjnego.
Dla większości jednokomórkowych organizmów, reprodukcja jest prostą sprawą powielania komórek, znany także jako proces replikacji. Ale dla organizmów wielokomórkowych, replikacji komórek oraz reprodukcja to dwa odrębne procesy. Organizm wielokomórkowy zastępuje uszkodzone lub zużyte komórki w procesie replikacji zwanym mitozą, podział jądra komórki eukariotycznej do produkcji dwóch identycznych jąder potomnych. Aby odtworzyć, eukarioty muszą najpierw utworzyć specjalne komórki zwane gametami - jajami i nasienie, które następnie łączą się dając początek nowego organizmu. Gamety to tylko jeden z wielu unikalnych rodzajów komórek organizmów wielokomórkowych, niezbędnych do działanie jako kompletny organizm.
Tworzenie nowych komórek
Większość organizmów jednokomórkowych tworzą ich następną generację przez replikację wszystkich jej części, a następnie podział na dwie komórki, jest to typ aseksualnej reprodukcji zwany binarnym rozszczepieniem. Sprawny proces to nie tylko dwie nowe komórki, ale także dwa nowe organizmy. Wielokomórkowe organizmy replikują nowe komórki w podobny sposób. Na przykład, tworzenie nowych komórek skóry i komórek wątroby powstają w replikacji DNA w komórce, że poprzez mitozę. Jednak produkcja zupełnie nowego organizmu wymaga seksualnej reprodukcji, przynajmniej dla większości organizmów wielokomórkowych. W pierwszym kroku, wyspecjalizowane komórki zwane jajami i nasieniem są one tworzone za pomocą procesu zwanego mejozą. Mejoza służy do zmniejszenia liczby chromosomów dla danego organizmu o połowę. W drugim kroku, plemniki i jaja łączą się aby utworzyć pojedynczą komórkę, która posiada pełną liczbę chromosomów. Ta połączona komórka, następnie dzieli się i różnicuje na różne rodzaje komórek, które ostatecznie stanowią funkcjonalny organizm.
Rysunek 3. Przegląd ważniejszych wydarzeń w mitozie.
Mitoza jest procesem, w którym jądro diploidalne (ma dwa zestawy chromosomów homologicznych) w komórkach somatycznych dzieli się by produkować dwa potomne jądra, które są nadal diploidalne. Po lewej stronie rysunek pokazuje w jaki sposób komórka rodzicielska duplikuje swoje chromosomy (jeden czerwony i jeden niebieski), dostarczając komórki potomne, z pełną kopią informacji genetycznej. Następnie chromosomy układają się w równikowej płaszczyźnie, dostarczając centromerów. Siostrzane chromatydy następnie rozchodzą się, dając dwie diploidalne komórki potomne, z których po jednym czerwonym i jednym niebieskim chromosomie.
Mitoza
Za każdym razem, gdy komórka dzieli się, to musi zapewnić, że jej DNA jest podzielony między dwie komórki potomne. Mitoza to proces "dzielący" genom między komórki potomne. Aby łatwiej opisać ten proces, wyobraźmy sobie komórki tylko z jednym chromosomem. Przed wejściem komórki w mitozę, mówimy że komórka jest w interfazie, stanie komórek eukariotycznych, gdy niepoddawane są podziałowi. Za każdym razem, gdy komórka dzieli się, to trzeba najpierw replikować całe jego DNA. Ponieważ chromosomy są po prostu DNA owiniętym w białka, komórka replikuje także jej chromosomy. Te dwa chromosomy, umieszczone obok siebie, są nazywane siostrzanymi chromatydami, są to identycznymi kopiami siebie. Przed tym zanim komórka może się dzielić, musi rozdzielić te dwie siostrzane chromatydy od siebie. Aby to zrobić, chromosomy trzeba skondensować. Ten etap mitoza nazywa się profazą. Następnie membrana jądrowa rozpada się, a duże sieci białkowe, zwane wrzecionami, przywiązuje się do każdej z siostrzanych chromatyd. Chromosomów są teraz ułożone prostopadle do wrzeciona w procesie o nazwie metafaza. Dalej, "molekularnych silniki" ciągną chromosomy od płytki metafazowej do biegunów komórki. Jest to tzw. anafaza. Kiedy ten proces się zakończy, komórki dzielą otoczkę jądrową i chromosomy relaksują się i dekondensują się podczas telofazy. Komórka może replikować jego DNA ponownie w interfazie i przejść przez mitozę jeszcze raz.
Kontrola cyklu komórkowego i rak komórki
W cyklu komórkowym podczas interfazy i mitozy, system nadzoru monitoruje uszkodzenia DNA komórek i niepowodzenia, aby wykonać krytycznych procesów. Jeśli system wyczuje ten problem, sieć cząsteczek sygnałowych zobowiązuje do zatrzymania podziałów komórki. Te tak zwane "punkty kontrolne" dają komórce informację, czy komórki potrzebują naprawy uszkodzenia lub wszczęcia zaprogramowane śmierci komórkowej, procesu zwanego apoptozą. Zaprogramowana śmierć komórek zapewnia, że uszkodzone komórki nie są dalej propagowane. Naukowcy wiedzą, że niektóre białka, zwane p53, akceptuje sygnały wysyłane przez uszkodzenia DNA. Odpowiada on poprzez pobudzanie produkcji białka hamującego, następnie zatrzymuje procesu replikacji DNA. Bez odpowiedniej funkcji p53, uszkodzenia DNA mogą się gromadzić. Bezpośrednią konsekwencją jest to, że uszkodzony gen wstępuje w stan nowotworowy. Dzisiaj, wady P53 są związane z różnego rodzaju nowotworów, w tym raka piersi i okrężnicy.
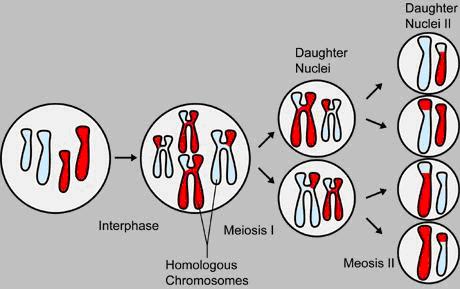
Rysunek 4. Przegląd ważniejszych wydarzeń w mejoza.
Mejoza, typu jądrowego podziału, występuje tylko w komórkach rozrodczych i z diploidalnej komórki (po dwa zestawy chromosomów) powstają cztery haploidalne komórki (posiadające jeden zestaw chromosomów). Każda komórka haploidalne może następnie łączyć się z gametą przeciwnej płci podczas rozmnażania płciowego. Na tej ilustracji, dwie pary homologicznych chromosomów wprowadzonych w mejoza I, której początkowym wynikiem są dwa siostrzane jądra, z których każde ma dwie kopie każdego z chromosomów. Te dwie komórki a następnie wchodzą w mejozę II produkującą cztery potomne jądra, z których każde posiada jeden egzemplarz każdego chromosomu.
Mejoza
Mejoza jest wyspecjalizowanym rodzajem podziału komórki, która ma miejsce w trakcie tworzenia gamet. Choć może wydawać że mejoza jest znacznie bardziej skomplikowana niż mitoza, to są to tak naprawdę tylko dwa podziały komórek zachodzące po sobie. Każdy z tych podziałów wykazuje silne podobieństwo do mitozy.
Mejoza I odnosi się do pierwszego z dwóch podziałów i często nazywana jest podziałem redukcyjnym. Dzieje się tak, ponieważ jest to ten proces, w którym komplet chromosomów została zmniejszony z diploidalnego (dwie kopie) do haploidalnego (jedna kopia). Interfaza w mejozie jest identyczna jak w interfaza mitozy. Na tym etapie nie ma możliwości określenia, jakiemu rodzajowi podziału komórki będą poddane. Mejotyczny podział następuje tylko w komórkach związanych z męskimi lub żeńskimi narządami rozrodczymi. Profaza I jest niemal identyczna z profazą w mitozie, dotyczy wyglądu chromosomów, rozwoju wrzeciona podziałowego oraz podziału membrany jądrowej. W metafazie I występuje znacząca różnica pomiędzy mejozą i mitozą. W mitozie wszystkie chromosomy układają się w płytkę metafazową w nieokreślonym porządku. W metafazie I, pary chromosomów są ułożone po obu stronach płytki metafazowej. Podczas tego procesu układania, ramiona chromatyd mogą nakładać się na siebie i łączyć się tymczasowo, w procesie zwanym crossing-over. Podczas Anafazy I włókna wrzeciona przyłączają się i ciągną homologiczne pary od siebie nawzajem i do każdego bieguna komórki. W telofazie I, powstają typowe formy, zaraz po cytokinezie, zmianach, które występują w cytoplazmie komórki w trakcie podziału jądrowego, ale otoczka jądrowa jest zazwyczaj niezreformowana, a chromosomy nie znikają. Pod koniec telofazy I każda komórka potomna ma jeden zestaw chromosomów, z czego połowa całkowitej liczby komórek w oryginalnym, to jest, podczas gdy pierwotna komórka była diploidalną; potomne komórki są obecnie haploidalne.
Mejoza II jest po prostu całkiem podziałem mitotycznym każdej z komórek haploidalnych, które są produkowane w mejozie I. Nie ma interfazy między mejozą I oraz mejozą II, a druga zaczyna się od Profazy II. Na tym etapie nowy zestaw form włókien wrzeciona i chromosomy zaczynają się poruszać w kierunku równika do komórki. Podczas metafazy II, wszystkie z chromosomów w dwóch komórkach układają się w płytkę metafazową. In. Anafaza II, dzieli centromery, a włókna wrzeciona się skracają, prowadząc chromosomy prosto do biegunów komórki. W telofazie II, rozwijają się bruzdy, zaraz po cytokinezie i formowaniu otoczki jądrowej. Chromosomów rozpoczynają gasnąc i przemieszczają się w chromatynę, charakterystyczna dla interfazy. Gdy mejoza II dobiegnie końca, będą w sumie cztery komórki potomne, w których połowa całkowitej liczby chromosomów jest z oryginalnej komórki. W przypadku struktur męskich wszystkie cztery komórki ostatecznie rozwijają się w komórki nasienia. W przypadku cyklu kobiet w organizmach wyższych, trzy komórki zazwyczaj są usuwane, pozostawiając pojedynczej komórki do rozwinięcia się w komórkę jajową, która jest znacznie większa niż komórki nasienia.
Rekombinacja - Fizyczna Wymiana DNA
Wszystkie organizmy zostają poddane pewnej liczbie drobnych mutacji lub losowym zmianom w sekwencji DNA, w procesie replikacji DNA. Są to spontaniczne mutacje i występuje w tempie charakterystycznym dla tego organizmu. Genetyczna rekombinacja odnosi się bardziej do dużych modyfikacji DNA cząsteczki. Ten proces wymaga utworzenia par między dwoma komplementarnymi obszarami rodzicielskiego dupleksu lub podwójnej helisy DNA, oraz powoduje fizyczną wymianę materiału chromosomów.
Stanowisko, na którym znajduje się gen na chromosomie nazywa się locus. W danej osoby, można znaleźć dwie różne wersje tego genu w danym locus. Alternatywne formy genu nazywane są allelami. Podczas mejozy I, kiedy chromosomy się układają się wzdłuż płytki metafazowej, dwa elementy z pary chromosomów mogą fizycznie się krzyżować. Może to spowodować że ramiona rozdzielą się w crossing-over i ponownie połączą z innym chromosomem, powodując wymianę części chromosomu.
Rekombinacja powoduje powstanie nowego układu alleli matki i ojca w tym samym chromosomie. Chociaż te same geny występują w tej samej kolejności, allele są różne. Proces ten wyjaśnia, dlaczego potomstwo z tych samych rodziców mogą wyglądać inaczej. W ten sposób, teoretycznie możliwa jest dowolna kombinacja rodzicielskich alleli w potomstwie, oraz fakt, że dwa allele pojawiają się razem w jednym potomstwie nie ma żadnego wpływu na statystyczne prawdopodobieństwo, że inne potomstwo będzie miało samą kombinację. Ta teoria "niezależnego asortymentu" alleli ma podstawowe znaczenie dla dziedziczenia genetycznego.
Częstotliwość rekombinacji faktycznie nie jest taki sam dla wszystkich kombinacji genów. Dzieje się tak dlatego, że na rekombinację znacząco wpływa bliskość jednego genu i drugiego. Jeśli dwa geny zlokalizowane są blisko siebie na chromosomie, prawdopodobieństwo, że będą rekombinacja oddzieli te dwa geny jest mniejsze niż gdyby były dalej od siebie. Sprzężenie opisuje tendencję genów by były dziedziczone razem ze względu na ich położenie w tym samym chromosomie. Brak równowagi sprzężenia opisuje sytuację, w której niektóre kombinacje genów lub markery genetyczne występują mniej lub bardziej często w populacji, niż można było oczekiwać od ich odległości od siebie. Naukowcy zastosowali tą koncepcję przy poszukiwaniu genów, które mogą wywoływać choroby. Robią to poprzez porównanie występowania konkretnej sekwencji DNA wraz z pojawieniem się choroby. Gdy znajdą wysoką korelację pomiędzy tymi dwoma, wiedzą, że są coraz bliżej znalezienia odpowiedniej sekwencji genu.
Binarne Rozszczepienie - Jak Bakterie się reprodukują?
Bakterie reprodukują się poprzez dość prosty proces zwany binarnym rozszczepienia lub rozmnażaniem żywych komórek przez podział na dwie równe, lub w przybliżeniu równe części. Jak zauważono, ten typ rozmnażania nie płciowego teoretycznie powoduje powstanie dwóch identycznych komórek. Jednak bakteryjne DNA ma stosunkowo wiele mutacji. Szybka ocena genetycznych zmian jest tym, co sprawia, że bakterie zdolne są do rozwoju oporności na antybiotyki i pomaga im wykorzystać to w inwazji w szerokim zakresie środowisk.
Podobnie do bardziej złożonych organizmów, bakterie również mają mechanizmy wymiany materiału genetycznego. Chociaż nie są równoważne do rozmnażania płciowego, końcowy wynik jest taki, że bakteria zawiera kombinację cech z dwóch różnych komórek rodzicielskich. Trzy różne sposoby wymiany do tej pory stwierdzono u bakterii.
Koniugacja polega na bezpośrednim połączeniu dwóch bakterii, co pozwala ich okrągłym DNA do poddania się rekombinacji. Bakterie mogą również zostać poddane transformacji przez absorpcję pozostałości martwych DNA z bakterii i integracji tych fragmentów DNA, do swoich własnych. Wreszcie, bakterie mogą wymieniać materiał genetyczny na drodze procesu zwanego transdukcją, w których geny są transportowane do komórki bakteryjnej przez wirusy, zwane bakteriofagami lub plazmidami, autonomicznym samo-replikującymi się chromosomami kolistego DNA.
Wirusowa Reprodukcja
Ponieważ wirusy są bez komórkowe i nie używają ATP, muszą one korzystać z maszyn i metabolizm komórek gospodarza do reprodukcji. Z tego powodu wirusy nazywane są wewnątrzkomórkowymi pasożytami. Zanim wirus dostanie się do komórki gospodarza, to nazywa się wirionem - pakiet wirusowego materiału genetycznego. Wirionów – wirusowa cząstka może być przekazywane z gospodarza do gospodarza albo poprzez bezpośredni kontakt lub za pośrednictwem wektora, lub przewoźnika. Wewnątrz organizmu, wirus może wejść do komórek na różne sposoby. Bacteriofagi - wirusów bakteryjne -przywiązują się do powierzchni ściany komórkowej w określonych miejscach. Po przyłączeniu, enzymy dokonują niewielkiej dziury w ścianie komórkowej, a wirus wbija jego DNA do komórek. Inne wirusy (np. HIV), wprowadzają do gospodarza za pośrednictwem endocytozy, procesu, w którym komórki pobierają materiał z środowiska zewnętrznego. Po wejściu do komórki, materiał genetyczny wirusa rozpoczyna destrukcyjny proces przejmowania komórki i zmusza ją do produkcji nowych wirusów.
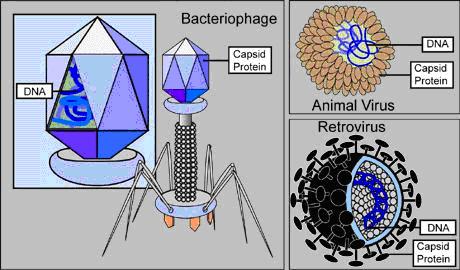
Rysunek 5. Typy wirusów.
Ta ilustracja przedstawia trzy typy wirusów: wirusy bakterii, inaczej zwane bakteriofagami (po lewej w środku); wirusy zwierzęce (u góry po prawej), retrowirusy (na dole po prawej). Wirusy zależą od komórki gospodarza i infekują ją żeby się reprodukować. Jeśli wirus znajdzie się na zewnątrz komórki gospodarza, wirusy, w najprostszej postaci, składają się tylko z genomicznego kwasu nukleinowego, albo DNA lub RNA (przedstawiani jako niebieski), otoczony przez białko płaszcza lub kapsyd.
Istnieją trzy różne sposoby w jaki informacja genetyczna zawartej w genomie wirusowym może być powielana. Forma materiału genetycznego zawartego w wirusowym kapsydzie, białku płaszcza, który otacza kwas nukleinowy, określa dokładny proces replikacji. Niektóre wirusy mają DNA, które po wejściu do wnętrza komórki gospodarza jest replikowana przez gospodarza wraz z własnym DNA. Następnie są dwa różne procesy replikacji wirusów zawierających RNA. W pierwszym procesie, wirusowe RNA jest bezpośrednio skopiowane przy użyciu enzymu o nazwie RNA replikaza. Enzym ten następnie jest używa RNA kopii jako matrycę do zrobienia setek duplikatów oryginału RNA. Druga grupa wirusów zawierających RNA, zwany retrowirusami, używa enzymu odwrotnej transkryptazy do syntezy DNA aby wirusowa genetyczna informacji zawartych była w cząsteczce DNA. Wirusowe DNA mogą być następnie dalej replikowane przy użyciu komórki gospodarza.
Kroki związane z wirusową reprodukcją
Gdy wirus ma przejętą kontrolę nad komórką, to natychmiast kieruje rozpoczęciem produkcji białek niezbędnych do reprodukcji wirusa. Gospodarz produkuje trzy rodzaje białek: wczesne białka, enzymy stosowane w replikacji kwasu nukleinowego; późnych białek, białka wykorzystane do skonstruowania płaszcza wirusa i lityczne białka, enzymy stosowane aby otworzyć komórki i uwolnić wirusa. Końcowy wirusowy produkt jest zmontowany spontanicznie, to jest, części są robione oddzielnie przez gospodarza i są połączone przez przypadek. Ten samo-montaż jest często wspomagany przez molekularne chaperony lub białek produkowane przez gospodarza, które pomagają kapsydowi połączyć się.
Nowe wirusy następnie opuszczają komórkę albo przez egzocytozę lub lizę. Otoczone błoną wirusy zwierząt instruują retikulum endoplazmatyczne gospodarza do produkcji pewnych białek, zwane glikoproteinami, które następnie zbierają się w „clumps” wzdłuż membrany komórek. Wirus jest następnie uwalniany z tych komórek w miejscach wyjścia, o których mowa w egzocytozie. Z drugiej strony, bakteriofag musi złamać albo doprowadzić do lizy komórkę, aby wyjść. W tym celu fagi mają gen, który koduje enzym o nazwie lizozymu. Ten enzym rozkłada ściany komórkowe, co powoduje, że komórka puchnie i rozrywa się. Nowe wirusy są uwalniane do środowiska naturalnego, zabijając komórki gospodarza w tym procesie.
Dlaczego badamy Wirusy?
Jedna rodzina wirusów zwierzęcych, zwanych retrowirusami, zawiera RNA genomowe w cząsteczce wirusa, ale syntetyzuje DNA kopii ich genomu w zakażonych komórkach. Retrowirusy stanowią doskonały przykład tego, w jaki sposób wirusy mogą odgrywać istotną rolę jako modele do badań biologicznych. Badania nad tymi wirusami, co pokazało pierwszy raz syntezę DNA z RNA pokazują podstawowy szablon do przenoszenia materiału genetycznego, który występuje w obu eukariotów i prokariotów.
Wirusy są istotne dla analizy biologii molekularnej i komórkowej, ponieważ przewidują one proste systemy, które mogą być wykorzystywane do manipulowania i zbadanie funkcji wielu typów komórek. Omówiliśmy właśnie jak replikacja wirusowa zależy od metabolizmu zainfekowanej komórki. Dlatego też badania wirusów mogą dostarczyć podstawowych informacji na temat aspektów biologii komórki i metabolizmu. Szybki wzrost oraz mały rozmiar genomu bakterii czynią z nich doskonałe narzędzia do badań w biologii. Wirusy bakteryjne także uproszczają badania mad genetyką bakterii i pogłębiają naszą wiedzę na temat podstawowych mechanizmów genetyki molekularnej. Ze względu na złożoność genomu komórki zwierzęcia, wirusy stały się jeszcze bardziej istotne w badaniach nad komórkami zwierząt niż badania komórki bakterii. Liczne badania wykazały użyteczność zwierzęcych wirusów jako sond do śledzenia różnych działań komórek eukariotycznych. Inne przykłady, w których zwierzęce wirusy dostarczyły ważnych modeli badań biologicznych ich komórek gospodarza obejmują badania DNA replikacji, transkrypcji, RNA, przetwarzania i transportu białek.
Powstające nowe rodzaje komórek
Popatrz uważnie na organizm ludzki, i to jest oczywiste, że nie wszystkie komórki są podobne. Na przykład, komórki, które tworzą naszą skórę są oczywiście różne od komórek, które tworzą nasze wewnętrzne narządy. Jednak wszystkie z różnych typów komórek w naszym organizmie, są otrzymane lub powstają z jednego, zapłodnionego jaja poprzez różnicowanie. Różnicowanie jest procesem, w którym niewyspecjalizowane komórki stają się wyspecjalizowane jak jedna z wielu komórek, które tworzą nasze ciało, takich jak komórki serca, wątroby i mięśni. Podczas różnicowania pewne geny są włączone, lub stają się aktywne, podczas gdy inne geny są wyłączone lub inaktywowane. Proces ten jest w dużym stopniu regulowany. W rezultacie, zróżnicowane komórki rozwijają się w konkretne struktury i pełnią niektóre funkcje.
Rodzaje komórek ssaków
Trzy podstawowe kategorie komórek ssaków tworzące ciało: komórki rozrodcze, komórki somatyczne i komórki macierzyste. Każda z około 100000000000000 komórek dorosłego człowieka ma własną kopię lub kopie genomu, jedynym wyjątkiem są niektóre rodzaje komórek z brakiem jądra w pełni zróżnicowanym stanie, takie jak czerwone krwinki. Większość z tych komórek jest diploidalny, lub ma dwie kopie każdego z chromosomów. Komórki te nazywane są komórkami somatycznymi. Ta kategoria obejmuje większość komórek, które tworzą nasze ciało, takie jak komórki skóry i mięśni. Komórki linii zarodkowej są każdą linią komórek, które dają podstawę do gamet – komórek jajowych i nasienia i są kontynuowane poprzez pokolenia. Komórki macierzyste, z drugiej strony, mają możliwość podziału na okres nieokreślony i mogą prowadzić do wyspecjalizowanych komórek. Są to najlepiej opisane komórki w kontekście normalnego rozwoju człowieka.
Rozwój człowieka zaczyna się, gdy nasienie zapłodni komórkę jajową i utworzy pojedynczą komórkę, która ma potencjał do uformowania całego organizmu. W ciągu pierwszych godzin po zapłodnieniu, ta komórka dzieli się na identyczne komórki. Około 4 dni po zapłodnieniu i po kilku cyklach podziału komórki, te komórki zaczynają się specjalizować, tworząc puste przestrzenie komórek, tzw. blastocysty. Blastocysta posiada zewnętrzną warstwę komórek, a wewnątrz pustą przestrzeń, istnieje grupa komórek zwanych wewnętrzną masą komórek. Komórki wewnętrznej masy komórkowej będą formować praktycznie wszystkie tkanki organizmu ludzkiego. Chociaż komórki wewnętrznej masy komórkowej mogą tworzyć praktycznie każdy rodzaj komórek w organizmie ludzkim, nie mogą stanowić organizmu. Dlatego też te komórki są nazywane polipotencjalnymi, to znaczy, że mogą one stanowić podstawę do wielu typów komórek, ale nie całego organizmu. Polipotencjalne komórki macierzyste poddawane są dalszej specjalizacji w komórki macierzyste, które zobowiązały się do utworzenia komórek, które mają szczególną funkcję. Przykłady obejmują macierzyste komórki krwi, które dają podstawę do krwinek czerwonych, krwinek białych i płytek krwi oraz komórki macierzyste skóry, które dają podstawę do różnych typów komórek skóry. Te bardziej wyspecjalizowanych komórek macierzystych są nazywane multipotencjalnymi - dającego podstawę do kilku rodzajów komórek, tkanek lub struktur.
Rysunek 6. Różnicowanie tkanek ludzkich.
Rozwój człowieka zaczyna się, gdy nasienie zapłodni komórkę jajową i utworzą pojedynczą komórkę, która ma potencjał formowania całego organizmu, zwanej zygota(górnym panelu, fiołkoworóżowy). W ciągu pierwszych godzin po zapłodnieniu, ta komórka dzieli się na identycznych komórek. Te komórki następnie rozpoczynają specjalizację, tworząc puste przestrzenie komórek, tzw. blastocystę (drugi zespół, fioletowy). Blastocysta posiada zewnętrzną warstwę komórek (żółty), a wewnątrz jej pustą przestrzeń, istnieje grupa komórek o nazwie wewnętrznej masy komórkowej (światło niebieskie). Wewnętrzna masa komórkowa może dawać wzrost zarodkowej komórce – komórce jajowej i nasieniu, jak również komórkom pochodzącym z wszystkich trzech warstw zarodkowych (ektoderma, błękitny; mezoderma, jasnozielony i endoderma, jasno żółte), umieszczonych w dole obrazka w tym komórkom nerwowym, komórkom mięśniowym, komórkom skóry, komórkom krwi, komórkom kości i chrząstki.
Replikacja DNA
Replikacji DNA lub proces podwajania genomu komórki, wymagany jest za każdym razem, gdy komórka się dzieli. Replikacja, jak wszystkie działania komórkowe wymaga specjalistycznych białek do realizacji zadania. W pierwszym kroku replikacji, specjalne białka, tzw. helikazy, rozwidlają się część podwójnej helisy rodzicielskiego DNA. Następnie cząsteczki DNA polimerazy- wspólna nazwa dla dwóch kategorii enzymów, które wpływają na syntezę DNA - wiąże się jednej linii DNA. Polimeraza DNA zaczyna się poruszać wzdłuż ramiona DNA w 3 'do 5' kierunku, korzystając z pojedynczego łańcucha DNA jako matrycy. Ten nowo syntetyzowany łańcuch jest wiodącą nicią i jest niezbędna dla dodawania nowych nukleotydów i reformowania podwójnej helisy. Ponieważ synteza DNA może nastąpić jedynie w 5 'do 3' kierunku, druga polimeraza DNA jest stosowany do wiązania się na drugiej nici jak zostanie otwarta podwójna helisa. To cząsteczka polimerazy syntetyzuje nieciągłe segmenty polinukleotydów, zwane fragmentami Okazaki. Kolejny enzym, zwany DNA ligazą, jest odpowiedzialny za połączenie tych fragmentów razem w coś co nazywane jest nicią opóźnioną.
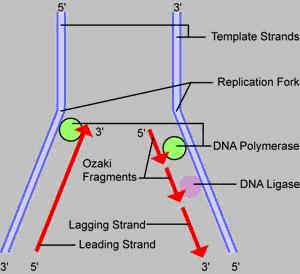
Rysunek 7. Przegląd replikacji DNA.
Średni ludzki chromosom zawiera ogromną liczbę par nukleotydów, które są kopiowane z prędkością około 50 par zasad na sekundę. Jednak cały proces replikacji trwa tylko około godziny. Dzieje się tak, ponieważ istnieje wiele miejsc początku replikacji na eukariotycznych chromosomach. Dlatego replikacja może rozpocząć się wcześniej niż w niektórych takich miejscach niż w innych. Jak replikacja dobiega końca, "bańka" nowo replikowanego DNA, spotyka się i odłącza, tworząc dwie nowe cząsteczki.
Z wielu miejsc pochodzenia replikacji, można zapytać, w jaki sposób komórka wie, które DNA, zostało już replikowane a które wciąż czeka na replikację? Do tej pory, dwa mechanizmy kontroli replikacji zostały zidentyfikowane: jeden pozytywny i jeden negatywny. Dla DNA, które ma być replikowane, każde miejsce początku replikacji musi być związane z zestawem białek o nazwie Origin Recognition Complex. Te pozostają dołączone do DNA w całym procesie. Szczególne akcesoria białek zwanych czynnikami licencji, muszą być również obecne aby rozpocząć replikację. Zniszczenie tych białek po rozpoczęciu replikacji zapobiega dalszej replikacji. Dzieje się tak, ponieważ czynniki licencji produkowane są jedynie w przypadku gdy jądrowa otoczka komórki jest przerwana podczas mitozy.
Transkrypcja - tworzenie mRNA
Transkrypcja DNA odnosi się do syntezy RNA na matrycy DNA. Proces ten jest bardzo podobny do replikacji DNA. Oczywiście, istnieją różne białka, które kierują transkrypcją. Najważniejszym jest enzymem jest polimeraza RNA, enzym, który wpływają na syntezę RNA na matrycy DNA. Aby była rozpoczęta transkrypcja, polimeraza RNA musi być w stanie rozpoznać początek sekwencji genu, tak, że wie od czego zacząć syntetyzujący mRNA. Jest on skierowany do rozpoczęcia przez możliwość jednego z podjednostek rozpoznania konkretnej sekwencji DNA znalezione na początku genu, zwanego promotorem sekwencji. Promotor sekwencji jest kierownikiem sekwencji znajdującym się na jednej nici DNA, który instruuje polimerazę RNA, gdzie zacząć syntezę i w którym kierunku powinna być kontynuowana. Polimeraza RNA następnie rozdziela podwójną helisy w tym punkcie i rozpoczyna syntezę RNA komplementarnego do jednej z nici DNA. Ta nić jest nazywana antysensową lub matrycą, natomiast druga nić jest określana jako nić sensowna lub nić kodująca. Synteza następnie postępuje w sposób ukierunkowany.
Chociaż wiele wiadomo na temat transkrypcji, sygnały i zdarzenia, które kierują polimerazą RNA, aby zatrzymać transkrypcję i opuścić matrycę DNA pozostają niejasne. Eksperymenty w ciągu ostatnich lat wykazały, że eukariotyczne wiadomości zawierają poli (A) dodatkowy sygnał (AAUAAA) na swoim 3 'końcu, po którym następuje ciąg adeniny. Ten sygnał poli (A), również nazywany poli (A) stroną, przyczynia się nie tylko do dodania do poli (A) ogona, ale także do zakończenia transkrypcji i uwolnienia polimerazy RNA z matrycy DNA. Jednak transkrypcja nie kończy się tutaj. Przeciwnie, kontynuowana jest na kolejne 200 do 2000 zasad po tej stronie, zanim zostanie przerwana. Jest to przed lub w trakcie procesu terminacji, że powstający transkrypt jest przecięty w miejscu poli (A), prowadząc do utworzenia dwóch cząsteczek RNA. Część nowo utworzonych lub powstających RNA, a następnie poddawany dalszej modyfikacji, zwaną obróbką po-transkypcyjną, i staje się mRNA.. Dalsza część RNA staje się niestabilna i szybko jest degradowana.
Chociaż znaczenie poli (A) dodatkowego sygnału zostało ustalone, znaczenie sekwencji odrzucanej pozostaje niepewne. Ostatnie badania wskazują, że określony region, zwany zakończenie regionu, jest niezbędny do prawidłowego zakończenia transkrypcji. Badanie to również świadczy o tym, że zakończenie transkrypcji odbywa się w dwóch odrębnych etapach. W pierwszym kroku, powstający RNA jest pocięty na konkretne podjednostki regionu terminacji, mogącą prowadzić do jego uwolnienia od polimerazy RNA. W kolejnym kroku, polimeraza odrywa się od DNA. Stąd polimerazy RNA kontynuują transkrypcję DNA, przynajmniej na krótkie odległości.
Translacja białkowa – Jak kierować RNA do syntezy białek?
Komórkowa maszyna odpowiedzialna za syntezę białek to rybosomy. Rybosomów składają się ze strukturalnych RNA i około 80 różnych białek. W jego stanie nieaktywnym, istnieją w dwóch podjednostkach: dużej i małej podjednostce. Gdy mniejsza podjednostka napotka mRNA, proces translacji mRNA do białka się zaczyna. W dużej podjednostce, istnieją dwa miejsca wiązania dla aminokwasów, są więc wystarczająco blisko siebie, aby stworzyć więź. W miejscu „A” akceptowany jest nowy transfer RNA lub tRNA - cząsteczka, która działa jako tłumacz między mRNA i transportuje aminokwasy tworzące białka. Miejsce „P” wiąże tRNA, który dołączony do rosnącego łańcucha.
Jak tylko omówiliśmy, tRNA, która działa jako tłumacz między mRNA i białko jest specyficzną cząsteczką RNA. Każdy tRNA ma specyficzne miejsce akceptorowe, które wiąże konkretny tryplet nukleotydów, nazywany kodonem i miejsce antykodonowe, które wiąże sekwencję nukleotydów niesparowanych, anty-kodon, który następnie może być wiązany w kodonie. Każdy tRNA ma również specyficzne białka ładujące, tzw. syntetazę aminoacyl tRNA. To może tylko białko wiąże się z tym szczególnym tRNA i dołączyć prawidłowe aminokwasów do miejsca akceptorowego.
Sygnałem startu translacji jest kodon ATG, który koduje metioninę. Jednak, nie wszystkie białka zaczynają się metioniną. Często ten pierwszy aminokwas zostanie usunięty w późniejszych procesach. tRNA połączony z metioniną wiąże się do sygnału startu translacji.. Duża podjednostka wiąże się z mRNA oraz małą podjednostką i tak rozpoczyna się elongacja, formowanie łańcucha polipeptydowego. Po pierwszym związanie tRNA w miejscu A, rybosomy przesuwają się w taki sposób, że tRNA jest teraz w miejscu P. Nowe tRNA, odpowiadające kodonom w mRNA, wprowadzane jest w miejsce A i tworzy się więź pomiędzy dwoma aminokwasami. Pierwszy tRNA jest już uwolniony, a rybosomy zmieniają się znowu więc, tRNA niosące dwa aminokwasy jest teraz w miejscu P. tRNA następnie wiąże się z miejscem A. Ten proces wydłużania trwa do tego aż rybosomy osiągnie to, co nazywa się kodonem stop, jest to tryplet nukleotydów, który jest sygnałem zakończenia translacji. Gdy rybosom dociera do kodonu stop, żadne aminoacylowe tRNA nie wiąże się w puste miejsce A. Jest to sygnał dla rybosomów aby rozdzielić się w dużą i małą podjednostkę, uwalniając nowe białka i mRNA. Jednak to nie zawsze jest koniec historii. Białka często poddawane są dalszej modyfikacji, zwanej modyfikacją po-translacyjną. Na przykład, może zostać rozszczepiona przez białka-cięcia czyli enzym, nazywany proteazą, w określonym miejscu lub zostaje zmienionych kilka jej aminokwasów.
Mechanizmami naprawy DNA
Utrzymanie dokładność kodu genetycznego DNA jest krytyczne zarówno dla długo- i krótkoterminowego przeżycia komórek i gatunków. Czasami normalnej aktywności procesy, takie jak duplikacja DNA i tworzenie nowych gamet, wprowadza zmiany lub mutacje w DNA. Pozostałe zmiany są spowodowane narażeniem DNA na działanie substancji chemicznych, promieniowanie lub inne niekorzystne warunki środowiskowe. Bez względu na źródło, mutacje genetyczne mają potencjał zarówno dla pozytywnych i negatywnych skutków na osobników, jak również ich gatunek. Pozytywnymi rezultatami zmian w nieco inną wersję genu, są te które mogą ostatecznie okazać się korzystne w obliczu nowej choroby lub zmiany warunków środowiska. Takie korzystne zmiany są podstawą ewolucji. Inne mutacje są uważane za szkodliwe, lub mogą spowodować uszkodzenie komórki lub osoby. Na przykład, błędy sekwencji DNA mogą zakończyć się przed utworzeniem witalnego białka albo utworzeniem wadliwego białka. Są to często tego rodzaju błędy, które prowadzą do różnych chorób.
Potencjał uszkodzeń DNA zatrzymywany przez energiczny nadzoru i system naprawy. W ramach tego systemu, istnieje kilka enzymów zdolnych do naprawy uszkodzenia DNA. Niektóre z tych enzymów są specyficzne dla danego rodzaju uszkodzenia, podczas gdy inne mogą obsługiwać szereg rodzajów mutacji. Systemy te różnią się stopniem, w jakim są w stanie przywrócić normalne lub dzikiego typu sekwencje.
Kategorie systemu naprawy DNA
Od komórek do genomów
Zrozumienie tego, co tworzy komórki i jak ona działa ma podstawowe znaczenie dla wszystkich nauk biologicznych. Docenianie podobieństw i różnic pomiędzy rodzajami komórek, jest szczególnie ważne w dziedzinie biologii komórkowej i molekularnej. Te podstawowe podobieństwa i różnice przedstawiają jednoczący temat, pozwalając zasadom wyciągniętym z jednego rodzaju komórki do ekstrapolacji i uogólnienia do innych typów komórek.
Być może najbardziej podstawowe własnością wszystkich żywych rzeczy jest ich zdolność do reprodukcji. Wszystkie komórki powstają z wcześniej istniejących komórek, a ich materiał genetyczny musi być powielany i przekazywany z rodzica na potomstwo. Podobnie, wszystkie organizmy wielokomórkowe dziedziczą ich informacje genetyczne określające strukturę i funkcje od swoich rodziców. W kolejnej sekcji z genetyki czym jest genom, są szczegóły jak informacja genetyczna jest replikowana i przekazywana z komórki do komórki i z organizmu do organizmu.
Życie jest określane przez genomy. Każdy organizm, w tym człowieka, ma genom, który zawiera wszystkie informacje biologiczne potrzebne do budowy i utrzymania życia tego organizmu. Biologiczne informacje zawarte w genomie są zakodowane w jego kwasie dezoksyrybonukleinowym (DNA) i dzielą się na dyskretne jednostki zwane genami. Geny kodują białka które przyłączają się do genomu w odpowiedniej pozycji i włączają szereg reakcji nazywanych ekspresją genów.
W 1909 roku, duński botanik Wilhelm Johanssen użył słowa gen dla dziedziczna jednostki znajdującej się na chromosomie. Prawie 50 lat wcześniej, Gregor Mendel scharakteryzował jednostki jako czynniki dziedziczne - zauważalnych różnic, które przeszły od rodzica do potomstwa. Dzisiaj wiemy, że pojedynczy gen składa się z niepowtarzalnych sekwencji DNA, która zawiera pełne instrukcje, aby wyprodukować funkcjonalny produkt, o nazwie białko. Geny kierują komórkami każdego typu, takich jak skóry, mózgu, wątroby, aby wytworzyć zestawy białek w odpowiednim czasie, a to dzięki temu, że powstała specyfika unikalnych organizmów.
Jądrowe DNA
Wewnątrz każdej z naszych komórek znajduje się jądro, a otaczająca je membrana stanowi region, który prowadzi do sanktuarium informacji genetycznej. Jądro zawiera długie nici DNA, które kodują te informacje genetyczne. Łańcuch DNA składa się z czterech chemicznych zasad: adeniny (A) i guaniny (G), które nazywane są purynami i cytozyny (C) i tyminy (T), o których mowa jako pirymidynach. Każda zasada ma nieco inny skład, lub kombinację tlenu, dwutlenku węgla, azotu i wodoru. W łańcuchu DNA, każda zasada jest dołączona do cząsteczki cukru (deoksyrybozy) i fosforanu, w wyniku których powstają nukleotydy lub kwasów nukleinowych. Poszczególne nukleotydy są ze sobą powiązane poprzez grupy fosforanowe, i to jest dokładny cel, lub sekwencji nukleotydów, która określa, że produkt wykonany jest z genów.
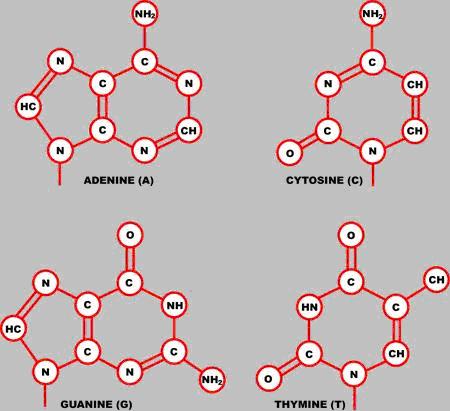
Rysunek 1. Cztery zasady DNA.
DNA, które składa się z genów jest dwukrotnie skręconą cząsteczką składającą się z dwóch łańcuchów ułożonych w przeciwnych kierunkach. Charakter chemiczny zasad w podwójnej helisie DNA tworzy niewielkie siły skręcania, który daje życie DNA charakterystyczny delikatnie skręconą strukturę, znaną jako podwójna helisa. Te dwa łańcuchy są ze sobą połączone przez chemiczne parowanie każdej zasady z jednym konkretnym partnerem na drugim łańcuchu. Adenina (A) paruje się z tyminą (T) oraz guanina (G) tworzy pary z cytozyną (C).. Tak więc, pary AT i GC mówi się, że są komplementarne. Takie parowanie komplementarna zasad jest tym, co sprawia, że DNA jest dopasowaną cząsteczką do niesienia naszej informacji genetycznej - jedna nić DNA może działać jako matryca do bezpośredniej syntezy komplementarnej nici. W ten sposób, informacje w sekwencji DNA jest łatwo kopiowane i przekazywane do następnej generacji komórek.
Organelle DNA
Zarówno rośliny jak i zwierzęta posiadają organelle - "niewielkie organy" w komórce - o nazwie mitochondrium. Każde mitochondrium ma swój własny zestaw genów. Rośliny również mają drugą organelle, chloroplast, który również posiada własne DNA. Komórki często mają wiele mitochondriów, szczególnie komórki wymagające dużo energii, takie jak aktywne komórki mięśniowe. Jest tak dlatego, że mitochondria są odpowiedzialne za konwersję energii zmagazynowanej w makrocząsteczkach w formę użytkową dla komórki, czyli trójfosforan adenozyny (ATP). Tak, więc są one często określane jako generatory energii komórki.
W odróżnieniu od jądrowego DNA (DNA znajdującego się w jądrze komórki), z czego połowa pochodzi z matki, a połowa z ojca, mitochondrialny DNA jest dziedziczone tylko od matki. Jest tak dlatego, że mitochondria są tylko w żeńskich gametach lub komórkach jajowych u zwierząt rozmnażających się drogą płciową, a nie w męskich gametach lub nasieniu. Mitochondrialny DNA również nie rekombinuje; nie ma przekazania genów z jednego pokolenia do drugiego, jak to występuje w genach jądrowych.
Duża liczba mitochondriów znajduje się w tyle plemnika, dostarczając im silnik, który generuje energię potrzebną do płynięcia w kierunku komórki jajowej. Jednakże, gdy wchodzi do nasienia podczas zapłodnienia jaj, ogon odpada, pozbywając się ojcowskich mitochondriów.
Dlaczego istnieje oddzielny mitochondrialny genom?
Proces konwersji energii, który odbywa się w mitochondriach odbywa się w obecności tlenu. Inne procesy przemiany energii w komórce odbywają się bez tlenu. Niezależna tlenowa funkcji tych organelli jest uważana, że ewoluowała od bakterii, które mieszkały wewnątrz innych prostych organizmów odnosząc wzajemne korzyści, lub na zasadzie symbiozy, relacji, dostarczającej im możliwości tlenowych. Poprzez procesu ewolucji tych maleńkich organizmów zostały one włączone do komórek, z ich genetycznym systemem i funkcjami komórkowymi stały się zintegrowane do tworzenia funkcjonalnej jednostki komórkowej. Ponieważ mitochondria posiadają własne DNA, RNA i rybosomy, scenariusz ten jest całkiem możliwy. Ta teoria jest również wspierany przez istnienie eukariotycznych organizmów, zwanych Amoeba, które nie mają mitochondriów. Dlatego Amoeba muszą zawsze być w symbiozie z bakteriami tlenowymi.
Dlaczego badamy mitochondria?
Istnieje wiele chorób spowodowanych przez mutacje w mitochondrialnego DNA (mtDNA). Ze względu na produkcję energii w mitochondriach komórek, objawy chorób mitochondrialnych często są zwyrodnieniem lub awarią tkanki. Na przykład, mutacje mtDNA zidentyfikowano w niektórych formach cukrzycy, głuchoty oraz niektórych odziedziczonych chorobach serca. Oprócz mutacje w mtDNA są w stanie kumulować się podczas całego życia. Inaczej jest z mutacjami w DNA jądrowym, które mogą być naprawiane przez wyrafinowane mechanizmy ograniczania akumulacji mutacji. Mutacje mitochondrialnego DNA mogą także koncentrować się w mitochondriach określonych tkanek. Różnorodność śmiercionośnych chorób jest związane z dużą liczbą zgromadzonych mutacji w mitochondriach. Istnieje nawet teoria, teoria mitochondrialnego starzenia się, które sugerują, że nagromadzenie się mutacji w mitochondriach, lub dyski, odpowiada za proces starzenia. Te wady są związane z chorobą Parkinsona i chorobą Alzheimera, choć nie wiadomo, czy faktycznie są one powodowane przez wady lub są bezpośrednim wynikiem chorób. Jednak dowody wskazują, że mutacje przyczyniają się do rozwoju obu chorób.
Oprócz krytycznych komórkowych funkcji związanych z energią, genów mitochondrialnych są użyteczne dla biologów ewolucyjnych, ponieważ jest to materiał ich matek i zawiera wysokiego stopnia mutacje. Poprzez badanie wzorów mutacji, naukowcy są w stanie zrekonstruować wzór migracji i rozwoju w obrębie i między gatunkami. Na przykład analizy mtDNA został wykorzystany do śledzenia migracji ludzi z całej Azji przez Cieśninę Beringa do Ameryki Północnej i Południowej. Było ono także wykorzystywane do identyfikacji matek starożytnej linii, z których współczesny człowiek ewoluował.
Kwasy rybonukleinowe
Oprócz mRNA, DNA koduje inne formy RNA, w tym rybosomalne RNA (rRNA), transportowe RNA (tRNA) oraz małe jądrowe RNA (snRNA). rRNA i tRNA uczestniczy w budowie białka, a snRNA pomaga w procesie o nazwie splicing - proces edycji mRNA zanim będzie mógł być używany jako matryca do syntezy białek.
Podobnie jak DNA, kwas rybonukleinowy to łańcuch lub polimer nukleotydów z tym samym kierunkiem 5 'do 3' jego nici. Jednakże, ryboza cukru składnika RNA jest nieco inna chemicznie niż w DNA. RNA posiada atom 2’tlenu, który nie występuje w DNA. Istnieją inne podstawowe strukturalne różnice. Na przykład, uracyl zajmuje miejsce tyminy w nukleotydach DNA oraz RNA, jest w przeważającej części, jednonicową molekułą. DNA kieruje syntezą różnych cząsteczek RNA, z których każda posiada wyjątkową rolę w funkcjach komórkowych. Na przykład, wszystkie geny które kodują białka są najpierw utworzone w jądrze komórkowym w nić RNA, o nazwie messanger RNA (mRNA). mRNA przenosi informację zakodowaną w DNA z jądra do maszyn montażu białek, zwanych rybosomami, w cytoplazmie. Kompleks rybosomów używa mRNA jako matrycy do syntezy białka kodowanego dokładnie przez ten gen.
Białka
DNA tworzy RNA, RNA tworzy białka, a białka tworzą nas. (Francis Crick).
Mimo, że DNA jest nośnikiem informacji genetycznej w komórce, białka wykonują większość prac. Białka są długimi łańcuchami zawierającymi aż 20 różnych rodzajów aminokwasów. Każda komórka zawiera tysiące różnych białek: enzymy, które sprawiają, że powstają nowe cząsteczki i katalizują niemal wszystkich procesów chemicznych w komórkach, elementy konstrukcyjne, które dają kształt komórkom i pomagają im się poruszać; hormony przekazują sygnały, że w całym ciele; przeciwciała, które rozpoznają obce molekuły i cząsteczki transportujące, które przenoszą tlen. Kod genetycznego niesiony przez DNA jest tym co określa kolejność i liczbę aminokwasów, a tym samym kształt i funkcję białek.
"Centralny dogmat" - podstawowa zasada biologii molekularnej - stwierdza przepływ informacji genetycznej z DNA na RNA do białka. Ostatecznie jednak, że kod genetycznym występuje w DNA, ponieważ tylko DNA jest przekazywane z pokolenia na pokolenie. Jednak w procesie tworzenia białka zakodowane informacje muszą być przekazywane wiernie na RNA następnie na białka. Przeniesienie kodu z DNA do RNA jest dość prostym procesem nazywanym transkrypcją. Rozszyfrowaniem kodu w powstającym mRNA jest nieco bardziej skomplikowane. To pierwsze wymaga, aby mRNA opuściło jądro i stworzyło duży kompleks z specjalistycznymi RNA i białkami, które wspólnie nazywane są rybosomem. Tutaj mRNA jest tłumaczona na białka przez dekodowanie sekwencji mRNA w blokach trzech zasad RNA, zwanych kodonami, gdzie każdy kodon określa poszczególny aminokwas. W ten sposób, rybosomalny kompleks buduje białko z aminokwasów, kolejność aminokwasów precyzyjnie określona jest przez kolejność kodonów w mRNA.
Dany aminokwas może wystąpić w więcej niż jednym kodonie. Te dodatkowe kodony zwykle różnią się na trzeciej pozycji. Na przykład, aminokwasu seryny jest zakodowany przez UCU, UCC, UCA, i / lub UCG. To jest klucz do uniknięcia mutacji, które występują naturalnie w DNA i które są powielane i nowe komórki są produkowane. Zezwalając na kilka losowych zmian w DNA, aby nie mieć wpływu na ostateczną sekwencję białka, tworzona jest sieć bezpieczeństwa. Niektóre kodony nie kodują aminokwasów, ale kierują rybosomy, kiedy zakończyć dodawanie nowych aminokwasów.
Sedno genów: introny i eksony
Geny stanowią około 1 procent całkowitego DNA w genomie. W ludzkim genomie, część kodująca genu, zwana jest eksonem, są one przerywane przez interweniujące sekwencje, zwane intronami. Ponadto, geny eukariotyczne nie kodują białka w jeden ciągły odcinek DNA. Zarówno eksony i introny są "transkrybowane" na mRNA, w nim są transportowane do rybosomów, ale przed transportem pierwotne mRNA jest redagowany. Ten proces redagowania usuwa introny, łączy eksony razem, i dodaje, unikalne cechy do każdego końca transkryptu tworząc "dojrzałe" mRNA. Można zapytać, jaki jest cel intronów jeśli są one łączone po ich transkrypcji? Jest to jeszcze niejasne, jakie są wszystkie funkcje intronów, ale naukowcy uważają, że niektóre służą jako miejsce rekombinacji, procesu, w którym zachodzi wycięcie i zamiana genów, powodując że potomstwo posiada geny różne od jednego z rodziców, w wyniku powstania nowych genów z nowych kombinacjami eksonów mogą być one kluczem do ewolucji.

Rysunek 2. Rekombinacja.
Przewidywanie genów
Kiedy kompletna sekwencja mRNA dla genu jest znana, programy komputerowe są wykorzystywane do dostosowania sekwencji mRNA z odpowiednimi regionami genomu w sekwencji DNA. Daje to wiarygodne wskazanie początku i końca kodowania tego regionu genu. W przypadku braku pełnej sekwencji mRNA, granice mogą być oszacowane przez coraz poprawniejsze, ale nadal nieprecyzyjne, przewidywanie genów. Problemem jest brak jednolitego wzorca sekwencji, który wskazuje na początek lub koniec genu eukariotycznego. Na szczęście, środek genu, określany jako podstawowa sekwencja genów - ma wystarczająco spójne funkcje umożliwiające bardziej wiarygodne prognozy.
Jak właśnie omówiono, podróż z DNA na mRNA do białka wymaga, aby określić, gdzie gen zaczyna się i kończy. Musi to być wykonane zarówno podczas transkrypcji i translacji.
Transkrypcja
Transkrypcja, jest syntezą RNA z sekwencji DNA, prowadzoną przez enzym o nazwie polimeraza RNA. Cząsteczka ta rozpoznaje sekwencje DNA, gdzie transkrypcja jest inicjowana, w miejscu zwanym promotorem. Ogólnie rzecz biorąc, istnieją dwa "promotory" sekwencji początkowej od początku każdego genu. Lokalizacja i podstawowa sekwencja promotora jest różna dla prokariotów (bakterie) i eukariotów (wyższe organizmy), ale są one rozpoznawane, które może następnie dołączyć do sekwencji i prowadzić produkcję mRNA.
Komórki eukariotyczne mają trzy różne RNA polimerazy, każdy rozpoznaje trzy klasy genów. Polimeraza II RNA jest odpowiedzialny za syntezę białka mRNA. To wymaga polimerazy sekwencji zawierających TATAA, popularnie zwane TATA box, które znajduje się 25-30 nukleotydów w górę od początku genu, określanego jako inicjator sekwencji.
Transkrypcja kończy, gdy polimeraza natknie się na sygnał terminacji lub stop. U eukariotów proces ten nie jest w pełni zrozumiały. Prokarioty jednak wykazują tendencję do posiadania krótkich regionów składających się z G i C, które są w stanie fałdować się i nakładać na siebie i formować komplementarne pary zasad, tworząc pary w nowym mRNA. Te pary następnie powodują wędrówkę polimerazy i uwolnienie nowo utworzonego mRNA.
Translacja
Początek translacji, procesu, w którym kod genetyczny niesiony przez mRNA kieruje syntezą białek z aminokwasów, różni się nieco u prokariotów i eukariotów, chociaż oba procesy zawsze inicjonowane są w kodonie dla metioniny. Dla prokariotów, rybosom rozpoznaje i przywiązuje sekwencję AGGAGGU na mRNA, zwaną sekwencją Shine-Delgarno, która pojawia w górę zaraz od kodonu metioniny (AUG). Ciekawe, u eukariotów brak jest rozpoznania tej sekwencji i po prostu inicjacja translacji ma miejsce na aminokwasie metioniny, zwykle kodowanej przez zasady AUG, ale czasami GUG. Translacja jest zakańczana zarówno u prokariotów i eukariontów, gdy rybosom zatrzyma się na jednym z trzech kodonów stop.
Geny strukturalne, śmieciowe DNA i regulacja sekwencji
Ponad 98 procent genomu ma nieznane funkcje. Chociaż często określane jako "śmieci" DNA, naukowcy zaczynają odsłaniać funkcji wielu z tych intrygujących sekwencji DNA pomiędzy genów.
Geny Strukturalne
Sekwencje, które kodują białka są nazywane strukturalnymi genami. Chociaż prawdą jest to, że białka są najważniejszymi elementami strukturalnymi składników komórki, białka są również pracownikami komórki. Spełniają one funkcje transportu substancji odżywczych do komórek; syntetyzują nowe DNA, RNA i cząsteczki białek i przekazują sygnały chemicznych z zewnątrz do wnętrza komórki, jak również z całej komórki w procesie tworzenia białek.
Sekwencje regulacyjne
Klasa sekwencji zwanych regulacyjnymi sekwencjami stanowią nieznaczną część genomu, lecz prowadzą znaczące funkcje. Na przykład niektóre sekwencje wskazują początek i koniec genów rozpoczęcia replikacji i rekombinacji lub świadczą miejsca przyczepienia się białek, które z kolei włączają lub wyłączają geny. Jak geny strukturalne, sekwencje regulacyjne są dziedziczone, jednak nie są one powszechnie nazywane genami.
Inne regiony DNA
Czterdzieści do czterdziestu pięciu procent naszego genomu składa się z krótkich sekwencji, które są powtarzane, czasami setki razy. Istnieje wiele form tego "powtarzalnego DNA", a kilku są już znane funkcje, takie jak stabilizacja struktury chromosomów lub unieszkodliwianiu jednego z dwóch chromosomów X kobiet w rozwoju, proces o nazwie X-inaktywacji. Większość najbardziej powtarzających się sekwencji znalezionych do tej pory u ssaków jest nazywanych "satelitarnym DNA", ponieważ ich niezwykły skład pozwala im być łatwo oddzielanymi od innych DNA. Sekwencje te są związane z strukturą chromosomu i występują na centromerach (lub centrach) i telomerach (końcach) chromosomów. Choć nie odgrywają one rolę w kodowaniu białek, odgrywają one istotną rolę w strukturze chromosomów, powielaniu i podziale komórki. Wysoce zmienny charakter tych sekwencji czyni je doskonałymi "znacznikami" przez które osoby mogą być identyfikowane na podstawie ich niepowtarzalnego wzoru ich satelitarnego DNA.
Rysunek 3. Chromosom.
Chromosom składa się z bardzo długiej cząsteczki DNA i związanych z nim białek, które niosą dziedziczną informację. Centromery, przedstawione w centrum tego chromosomu, są wyspecjalizowaną strukturą, która pojawia się w trakcie podziałów komórkowych i zapewnia prawidłowe rozmieszczenie duplikowanych chromosomów do komórek potomnych. Telomery są strukturami na końcu chromosomów. Telomery odgrywają kluczową rolę w replikacja chromosomów i przeciwdziałaniu tendencji do skracania chromosomów w każdej rundzie replikacji.
Inną klasą niekodującego DNA są "pseudogeny", nazwany tak, ponieważ uważa się za resztę realnych genów, które poddane zostały mutacji i nie są funkcjonalne. Pseudogeny mogły powstać poprzez powielanie funkcjonalnego genu, a następnie przez inaktywację jednej kopii. Porównywanie obecności lub nieobecności pseudogenów jest metodą stosowaną przez genetyków ewolucyjnych do grupowania gatunków i określenia ich powiązań. Więc, sekwencje te są myślą do wniesienia zapisu naszej ewolucyjnej historii.
W lutym 2001, dwa w dużej mierze niezależne projekty ludzkiego genomu zostały opublikowane. W obu badaniach szacuje się, że istnieją 30000 do 40000 genów w ludzkim genomie, około jednej trzeciej liczby wcześniejszych szacunków. Ostatnio naukowcy szacują, że znajduje się mniej niż 30000 ludzkich genów. Jednak wciąż są to domysły nad rzeczywistą liczbą genów, ponieważ nie wszystkie z sekwencji ludzkiego genomu są opisane oraz nie wszystkich znanych sekwencji zostały przypisane danej pozycji w genomie.
Tak więc, w jaki sposób naukowcy szacują liczbę genów w genomie? W większości przypadków szukają sygnałów genów w sekwencji DNA. Należą do nich: otwarte ramki odczytu, odcinki DNA, zwykle większe niż 100 zasad, które nie są zakłócone przez kodon stop taki jak TAA, TAG lub TGA; kodony start jak ATG, specyficzne sekwencje znalezione na miejscach śmieci, lokalizację w sekwencji DNA, gdzie RNA, gdzie usuwa obszary niekodujące aby utworzyć ciągły transkrypt genów do translacji białko; i sekwencje genów regulacyjnych. Proces ten jest zależny od programów komputerowych, które poszukują tych wzorów w różnych bazach danych sekwencji, a następnie dokonują prognozy o istnieniu genu.
Z jednego genu - jednego białka do bardziej globalnej perspektywy
Tylko niewielki procent z 3 miliardów zasad ludzkiego genomu staje się wyrażone w produkcie genu. Jednakże, z około 1 procenta naszego genomu, który ulega ekspresji, 40 procent to alternatywnie łączone do wytwarzania wielu białek z jednego genu. Alternatywny splicing odnosi się do cięcia i wklejanie z pierwotnego transkryptu mRNA do różnych kombinacji dojrzałego mRNA. Dlatego teoria jeden gen- jedno białko, oryginalnie brzmiąca jako "jeden gen - jeden enzym", nie jest prawdziwa.
Przy tak dużej zawartości DNA w genomie, dlaczego transkrypcja ogranicza się do tak małej porcję, i dlaczego tak niewielka porcja pracuje ponad-czasem aby wyprodukować wiele alternatywnych transkryptów? Ten proces może ewoluować jako sposób na ograniczenie szkodliwych skutków mutacji. Genetyczne mutacje występują losowo, a efekt małej liczby mutacji jednego genu może być minimalny. Jednakże indywidualne posiadanie wielu genów każdego z niewielkimi zmianami może osłabić osobnika, a tym samym gatunek. Z drugiej strony, jeśli pojedyncza mutacja dotyczy kilku alternatywnych transkryptów na raz, jest to bardziej prawdopodobne, że efekt będzie destruktywny - osobnik może nie przetrwać do następnej generacji. Tak więc, alternatywne transkrypty z jednego genu mogą zmniejszyć szanse, że zmutowany gen będzie przekazany.
Przełączanie genów: włączanie i wyłączanie genów
Szacunkowa liczba genów ludzi, mniejsza niż 30000, nie jest tak różna od znanych genów 25300 Arabidopsis thaliana, powszechnie nazywanej trawą musztardową. Jednak wydaje się nam, przynajmniej na pierwszy rzut oka, że jesteśmy dużo bardziej skomplikowanym organizmem. Osoba, może się zastanawiać, jak ten wzrastająca złożoność została osiągnięta. Jedna odpowiedź jest w systemie regulacji genów, który włącza i wyłącza geny. System ten również precyzyjnie kontroluje ilość produktu genu, który jest produkowany i dalsze jego modyfikacje. Ta znakomita kontrola wymaga wielu punktów regulacyjnych wejściowych. Jednym z bardzo wydajnych punktów występującym w transkrypcji, jest taki, że mRNA wytwarzany jest tylko wtedy, gdy gen jest potrzebny. Komórki również regulują ekspresję genów przez zmiany po-transkrypcyjne; zezwalając jedynie podzestawom mRNA aby przejść do translacji lub poprzez ograniczenie translacji określonych mRNA do produktu tylko wtedy, gdy jest potrzebny. Na innych poziomach, komórkowa regulacja ekspresji genów poprzez fałdowanie DNA, chemiczną modyfikację zasad nukleotydów i skomplikowane "mechanizmy informacji zwrotnej", w których niektóre z białek, produkowane przez ten gen kierują komórkę do zaprzestania dalszej produkcji tego białka.
Kontrola transkrypcji
Promotorzy i regulatory sekwencji
Transkrypcja jest procesem, w którym RNA powstaje z DNA. Jest on rozpoczynany gdy enzym, polimeraza RNA, wiąże się z miejscem na DNA nazywanym sekwencją promotora. W większości przypadków, polimeraza jest wspomagana przez grupę białek zwanych czynnikami transkrypcyjnymi, które wykonują wyspecjalizowane funkcje, takie jak rozpoznanie sekwencji DNA i regulację aktywności enzymu polimerazy. Inne sekwencje regulacyjne obejmują aktywatory, represorzy i wzmacniacze. Te sekwencje mogą być cis działające (dotyczące genów, które są w sąsiedztwie sekwencji) lub trans działające (wpływających na ekspresję genu w odległym miejscu), nawet na innym chromosomie.
Wpływ struktury DNA i domeny wiążące
Sekwencje, które są ważne w regulacji transkrypcji niekoniecznie kodują czynniki transkrypcyjne lub inne białka. Transkrypcja może również być regulowana przez subtelne różnice w strukturze DNA i chemiczne zmiany w zasadach, do których wiążą się czynniki transkrypcyjne. Jak stwierdzono wcześniej, chemiczne właściwości czterech zasad DNA różnią się nieco, zapewniając każdej zasadzie unikalną możliwość chemicznej reakcji z innymi cząsteczkami. Jedna chemiczna modyfikacja DNA, zwane metylacją, zakłada wprowadzenie grupy metylowej (-CH 3). Metylacja często występuje w cytozynie, która jest poprzedzona przez guaninę, często w pobliżu sekwencji promotora. Metylacja DNA często koreluje z aktywnością funkcjonalną, gdzie nieaktywne geny są zwykle w większym stopniu metylowane. Dzieje się tak, ponieważ grupa metylowa służy do zahamowania transkrypcji poprzez przyciąganie białek, które wiążą się w szczególności do metylowanego DNA, tym samym zakłócając wiązanie polimerazy. Metylacja także odgrywa ważną rolę w imprinting genomu, które występuje wtedy, gdy zarówno allele matki i ojca są obecne, ale tylko jeden allel ulega ekspresji podczas gdy drugi pozostaje nieaktywny. Innym sposobem myślenia o imprinting genomu są różnice pochodzenia macierzystego w ekspresji dziedziczonych cech. Znacznie intrygujące skutki metylacji DNA i wielu naukowców pracuje nad odblokowaniem tajemnicy tej koncepcji.
Kontrola translacji
Translacja jest procesem, w którym kod genetyczny niesiony przez mRNA kieruje syntezą białek. Regulacja translacji występuje poprzez wiązanie specyficznych cząsteczek, zwanymi represorami białek, do sekwencji na cząsteczce RNA. Białka represorzy zapobiegają ekspresji genu. Jak już wyjaśnialiśmy, stanem domyślnym genu jest ekspresja przez rozpoznanie promotora przez polimerazę RNA. Blisko do regionu promotora jest inne miejsce cis działające nazywane operator, celem jego jest represja białka. Gdy represor białka wiąże się z operatorem, polimeraza RNA nie może inicjowanie transkrypcji i ekspresja genów jest wyłączona.
Kontrola translacji odgrywa znaczącą rolę w procesie rozwoju zarodkowego i różnicowania komórek. Po zapłodnieniu, komórka jajowa zaczyna mnożyć się do produkcji komórek, tak że wszystkie są takie same. W pewnym momencie jednak, te komórki zaczynają się różnicować, lub zmienia się w poszczególne typy komórek. Niektóre będą krwinkami lub komórkami nerek, podczas gdy inne mogą stać się komórkami nerwowymi lub komórkami mózgu. Kiedy wszystkie komórki tworzone są podobnie, te same geny są włączone. Jednakże różnicowanie powoduje, że różne geny w różnych komórkach muszą stać się aktywne dla spełnienia potrzeb tego typu komórek. W niektórych organizmach, komórki jajowe przechowują niedojrzałe mRNA, które staje się aktywne translacyjnie dopiero po zapłodnieniu. Zapłodnienie następnie służy do uruchomienia mechanizmów efektywnego rozpoczęcia translacji mRNA do białka. Podobne mechanizmy służą do aktywacji mRNA na innych etapach rozwoju i różnicowania, np. gdy konkretne produkty białkowe są potrzebne.
Czy wszyscy mają takie same geny?
Kiedy patrzysz na gatunek człowieka widać dowody procesu zwanego zmiennością genetyczną, to jest natychmiastowego rozpoznawania różnic w ludzkich cechach, takich jak włosy i kolor oczu, pigmentu skóry i wzrost. Następnie, nie są tak oczywiste różnice genetyczne, takie jak typ krwi. Te ulegające ekspresji lub fenotypowe, cechy są przypisane do zmienności genotypowej sekwencji DNA. Kiedy dwie osoby posiadają różne fenotypy tej samej cechy, są to dwa allele tego samego genu. Oznacza to, że w sekwencja genów jest nieco inna u dwóch osób, a o genie mówi się, że jest polimorficzny, "poli", co oznacza wiele i "morficzny", co oznacza, kształt lub formę. Dlatego, choć na ogół ludzie mają takie same geny, geny nie mają dokładnie takiej samej sekwencji DNA. Te polimorficzne miejsca wpływają na ekspresję genów, a także służą jako markery genomicznych wysiłków badawczych.
Zmienność genetyczna
Najczęściej zmienność genetyczna występuje w fazach cyklu komórkowego, gdy DNA jest powielany. Mutacje w nowej nici DNA mogą uwidaczniać się jako substytucje zasad, tak jak kiedy pojedyncza zasada zostaje zastąpiona inną; delecje, gdzie jedna lub więcej zasad jest opuszczone; lub insercja, jeżeli jedna lub więcej zasad zostaje dodana. Mutacje mogą być synonimami, w których zmiana ciągle powoduje że kodon dotyczy tego samego aminokwasu lub nie synonimami, w których zmienność powoduje wystąpienie kodonu innego aminokwasu. Mutacje mogą również powodować zmiany ramki odczytu, która pojawia się gdy zmiana powoduje zmianę punktu odniesienia dla odczytywania kodu genetycznego i powoduje utratę części lub czasami całości produktu genu. Mutacje DNA mogą być również wprowadzone przez toksyczne chemikalia oraz w szczególności w komórkach skóry narażenie na promieniowanie ultrafioletowe.
Mutacji, które występują w komórkach somatycznych, w dowolnej komórce ciała z wyjątkiem gamet i ich prekursorów, nie zostaną przeniesione do następnego pokolenia. Nie oznacza to jednak, że mutacje komórki somatycznej, czasami nazywane mutacjami nabytymi są łagodne. Na przykład, jak przygotowane komórki skóry przygotowane do podziału i produkcji nowych komórek skóry, błędy mogą być wprowadzone, gdy DNA jest powielany, co dało w efekcie że komórka potomna zawiera błąd. Chociaż większość wadliwych komórek umiera szybko, niektóre mogą się utrzymywać, a nawet mogą stać się nowotworowymi jeśli mutacja wpływa na zdolność do regulacji wzrostu komórek.
Mutacje i nowe pokolenia
Istnieją dwa miejsca, gdzie mutacje mogą być wprowadzone i przeprowadzane do następnej generacji. W pierwszych etapach rozwoju, komórki nasienia i komórki jajowe łączą się. Następnie zaczynają się dzielić, dając wzrost komórkom różnicowania komórek w tkankach dając specyficzne rodzaje komórek. Jednym wczesnego typem różnicowane komórki linii zarodkowej, które mogą ostatecznie przekształcić się w dojrzałe gamety. Jeśli mutacja występuje w rozwijającej się linii zarodkowej komórek, to może się ona utrzymać do momentu, aż osobnik osiągnie wiek reprodukcyjny. Teraz mutacja może mieć potencjał by być przekazywana następnemu pokoleniu.
Mutacje mogą być także wprowadzone podczas mejozy, trybie replikacji komórek do tworzenia nasienia i komórek jajowych. W tym przypadku linii zarodkowej komórki zdrowe i zmutowane są wprowadzone podczas rzeczywistego procesu replikacji gamet. Po raz kolejny komórki nasienia lub jaj będą zawierać mutacje oraz podczas procesu reprodukcyjnego, te mutacje mogą być następnie przekazywane do potomstwa.
Należy pamiętać, że nie wszystkie mutacje są złe. Mutacje również dają gatunkowi możliwość dostosowania do nowych warunków, jak również chronić gatunki od nowych czynników chorobotwórczych. Mutacje są, co leży za popularnym powiedzeniem, "przetrwania doboru naturalnego ", podstawową teorią ewolucji zaproponowaną przez Karola Darwina w 1859. Ta teoria proponuje, że podczas wzrostu nowych środowisk, osobnicy przenoszące niektóre mutacje, które niosą ewolucyjne korzyści przetrwają do przekazania tej mutacji na jego potomstwa. To nie wskazuje na to, że mutacja pochodzi ze środowiska, ale przetrwanie w tym środowisku jest wzmocniona w przypadku niektórych mutacji. Niektóre geny, a nawet niektóre organizmy ewoluowały aby tolerować mutacje lepiej niż inne. przykład niektóre geny wirusowe są znane, z tego że mają wysokie stopnie mutacji. Mutacji służy wirusowi również poprzez umożliwienie adaptacyjnych cech, takich jak zmiany w białku płaszcza zewnętrznego, tak aby mógł on uniknąć wykrycia, a tym samym zniszczyć systemu odpornościowy gospodarza. Wirusy także produkcją niektórych enzymów, które są niezbędne do zakażenia komórek gospodarza. Mutacje w obrębie takich enzymów mogą prowadzić do nowych form, które pozwalają na zakażenie wirusem jego gospodarzem, ale nie jest już zablokowany przez anty-wirusowe leki. Pozwoli to wirusom propagację w swoim środowisku.
Prawa Mendla – jak dziedziczymy nasze geny?
W roku 1866, Gregor Mendel badał przekazywanie siedmiu różnych cech groszku poprzez staranne badanie - krzyżowanie wielu odrębnych odmian grochu. Badanie grochu ogrodowego może wydawać się banalne do tych z nas, którzy żyją w nowoczesnym świecie klonowanych owiec i transfer genów, ale proste podejście Mendla doprowadziło do podstawowego spojrzenia na dziedzictwo genetyczne, znane dziś jako prawa Mendla. Mendel nie znał czy rozumiał mechanizmów komórkowych, które produkują wyniki, które zauważył. Niemniej jednak prawidłowo zauważył on zachowanie cech i matematyczne prognozy ich przekazywania, niezależną segregację alleli w trakcie produkcji gamet, a także niezależny asortyment genów. Być może tak niesamowity jak odkrycie Mendla był fakt, że jego praca została w dużej mierze ignorowane przez naukowców ponad 30 lat!
Podstawowe prawa dziedziczenia Mendla
Prawo segregacji: Każdy z dwóch dziedziczonych czynników (alleli) posiadanych przez rodziców będzie podlegać segregacji i przekazanie do oddzielnych gamet (jaj lub plemników) podczas mejozy, która będzie nieść tylko jeden z czynników.
Prawo niepodległego asortymentu: W gametach, allele jednego genu oddzielone niezależnie od tych innego genu, a więc wszystkie możliwe kombinacje alleli są jednakowo prawdopodobne.
Prawo dominacji: Każda cecha jest określona przez dwa czynniki (alleli), odziedziczone po jednym z każdego rodzica. Czynniki te wykazują cechy dominacji, współdominacji lub o recesywnej ekspresji, i te, które są dominujące będą maskować wypowiedzi tych, które są recesywne.
Jak działa dziedziczenie?
Nasza dyskusja jest tu ograniczona do płciowo rozmnażających się organizmów gdzie każdy gen w osobniku jest reprezentowany przez dwie kopie, zwany allelami - jednym dla każdej pary chromosomów. Może być więcej niż dwa allele lub warianty, w odniesieniu do danego genu w populacji, ale tylko dwa allele można znaleźć w osobniku. Dlatego prawdopodobieństwo, że poszczególne allele będą dziedziczone jest 50: 50, to allele losowo i niezależnie segregują się do komórek potomnych, choć istnieją pewne wyjątki od tej reguły.
Termin diploid opisuje stan, w którym komórka ma dwa zestawy chromosomów homologicznych, lub dwa chromosomy, które są takie same. Dojrzewanie komórek macierzystych linii zarodkowej do gamet wymaga diploidalnej liczby każdego chromosomu aby została zmniejszona o połowę. Stąd, gametami są haploidalne - posiadają tylko pojedynczy zestaw chromosomów homologicznych. Redukcja ta zachodzi za pomocą procesu zwanego mejoza, gdzie jeden chromosom w parze diploidalnej wysyłana jest do każdej gamety potomnej. Ludzkie gamety, dlatego zawierają 23 chromosomy, połowę liczby komórek somatycznych, wszystkich innych komórek ciała.
Ponieważ chromosom w jednej parze oddzielony jest niezależnie od wszystkich innych chromosomów, każda nowa gamet ma potencjał dla całkowicie nowych kombinacji chromosomów. U ludzi, niezależna segregacja 23 chromosomów może prowadzić aż od 16 do 17 milionów różnych kombinacji w jednej gamecie osobnika. Tylko jedna z tych gamet będzie łączyć z jednym z blisko 17 milionów możliwych kombinacji z drugim rodzicem, generując potencjalne rozłożenia na indywidualne odmiany. Jednak to dopiero początek. Jeszcze więcej zmienności jest możliwych, jeśli wziąć pod uwagę rekombinację pomiędzy działami chromosomów podczas mejozy jak też losowych mutacji, które mogą wystąpić podczas replikacji DNA. Z takim wachlarzem możliwości, to jest niesamowite, że rodzeństwo wygląda tak podobnie!
Ekspresja dziedziczonych genów
Ekspresja genów, znajduje odzwierciedlenie w fenotypie organizmów, jest oparta na specyficznych warunkach dla każdej kopii genu. Jak tylko omówiliśmy, dla każdego genu człowieka istnieją dwie kopie, a dla każdego genu może być kilka wariantów lub alleli. Jeśli oba allele są takie same, o genie mówi się, że jest homozygotą. Jeśli allele są różne, są one uważane za heterozygoty. Dla niektórych alleli, ich wpływ na fenotyp ma pierwszeństwo przed wszystkimi innymi alleli. Dla innych, zależy od tego, czy ekspresja genu zależy czy jest on homozygotą lub heterozygotą. Jeszcze inne cechy fenotypowe są kombinacją kilku alleli kilku różnych genów. Determinacja kondycji alleli może być osiągnięta wyłącznie poprzez analizę rodowodów, w podobny sposób jaki Mendel przeprowadzał eksperymenty na grochu. Jednak ta metoda może zostawić wiele pytań bez odpowiedzi, zwłaszcza w przypadku cech, które są wynikiem interakcji pomiędzy kilkoma różnymi genami. Obecnie istnieją techniki genetyki molekularnej, które mogą pomóc naukowcom w śledzeniu przekazywania cech poprzez identyfikowanie lokalizacji poszczególnych genów, identyfikacji wariantów alleli i identyfikacji tych cech, które są powodowane przez wiele genów.
Wyjątki od praw Mendla
Istnieje wiele przykładów dziedzictwa, które wydają się być wyjątkami praw Mendla. Zazwyczaj te okazują się one reprezentować skomplikowane interakcje pomiędzy różnymi warunkami alleli. Na przykład współdominujące allele oba wpływają na fenotyp. Żaden nie dominuje nad pozostałymi. Kontrola systemu grupy krwi ludzkiej stanowi dobry przykład współpracy dominujących alleli.
Cztery podstawowe grupy krwi
Istnieją cztery podstawowe rodzaje krwi, a są to O, A, B i AB. Wiemy, że nasz typ krwi jest określany przez "allele", które dziedziczymy od naszych rodziców. Do tych typów krwi, istnieją trzy podstawowe allele krwi: A, B i O. Wszyscy mamy dwa allele, jeden odziedziczony od każdego z rodziców. Możliwych kombinacji trzech alleli jest: OO, AO, BO, AB, AA i BB. Typy krwi A i B są współdominującymi allelami, a O jest allelem recesywnym. Co-dominacja allelu pojawia się nawet jeśli obecny tylko jeden; recesywny allel pojawia się tylko jeśli dwa recesywne allele są obecne. Ponieważ typ krwi O jest recesywny, to nie jest on widoczny, jeśli osoba dziedziczy allel A lub B wraz z nim. Tak, możliwe kombinacje alleli powodują poszczególne typy krwi w ten sposób:
Plejotropizm lub plejotrofia, odnosi się do zjawiska, w których pojedynczy gen jest odpowiedzialny za tworzenie wielu odrębnych i pozornie niepowiązanych cech fenotypowych, czyli osobnik może wykazywać różne fenotypowe wyniki. Jest tak dlatego, że gen jest aktywny w wielu miejscach w organizmie. Przykładem jest Marfan's syndrome, gdzie występują wady genu kodującego białka tkanki łącznej. Osoby z syndromem Marfan wykazują nieprawidłowości w ich oczach, systemie szkieletowym i układzie sercowo-naczyniowym.
Niektóre geny maskują ekspresję innych genów, podobnie jak w pełni dominujący allel maskuje ekspresję jego recesywnego odpowiednika. Gen, który maskuje fenotypowy efekt innego genu nazywany jest genem epistatycznym; gen podwładne to geny hipostatyczne. Gen odpowiedzialny za bielactwo u ludzi jest epistatycznym genem. To nie jest częścią interakcji na genów koloru skóry. Przeciwnie, jej dominujące allele są niezbędne dla rozwoju wszelkich pigmentu skóry, a jego stan homozygoty recesywnej powoduje stan albinotyczny, niezależnie od tego, jak wiele innych genów barwnika może być obecnych. Z powodu skutków epistatycznego genu, niektóre osoby, które dziedziczą dominujący allel, choroby powodowane przez genu pokażą tylko częściowe objawy choroby. Niektóre z nich, w rzeczywistości, nie wykazują ekspresji choroby powodującej przez gen, warunek, o którym mowa w nonpenetrance. Osobniki, w których taki nonpenetrant zmutowanego genu istnieje będą fenotypowo normalne, ale wciąż zdolny do przepuszczenia szkodliwych genów na potomstwo, które mogą powodować pełną chorobę.
Następnie mamy cechy, które są wielogenowe, to te które wynikają z kilku różnych ekspresji genów. To jest prawdziwe w odniesieniu do ludzkiego koloru oczu, w których co najmniej trzy różne geny są odpowiedzialne za określenie koloru oczu. Geny brązowe / niebieskie i brązowy są zarówno na chromosomie 15, gdzie zielony/niebieski gen znajduje się na chromosomie 19. Interakcja pomiędzy tymi genami nie jest dobrze zrozumiana. Jest spekulacja, że mogą istnieć inne geny, które kontrolują inne czynniki, takie jak wielkość pigmentu złożonego na tęczówkę. Ten wielogenowy system wyjaśnia, dlaczego dwóch błękitnookich osobników może mieć brązowo-okie dziecko.
Mówiąc o kolor oczu, czy kiedykolwiek widziałeś ktoś z jednym zielonym okiem i jednym brązowym? W tym przypadku, somatyczny mozaicyzm może być winny. Jest to prawdopodobnie łatwiej opisać, niż wyjaśniać. W wielokomórkowych organizmach, każda komórka u dorosłego ostatecznie pochodzi od pojedynczych komórek zapłodnionych jaj. W związku z tym, każda komórka dorosłej osoby zwykle zawiera tą samą informację genetyczną. Jednak, co by się stało, jeśli mutacja występowała tylko w jednej komórce na dwie komórki w etapie rozwoju? Wtedy dorosły składałby się z dwóch rodzajów komórek: komórki z mutacją i komórek bez mutacji. Jeżeli mutacja wpływa na produkcję melaniny występującej w jednej z komórek w linii komórek z jednego oka, ale nie w innym, to oczy miałyby odmienny genetyczny potencjał syntezy melaniny. Mogłoby to powodować oczy dwóch różnych kolorów.
Penetracja odnosi się do stopnia, w jakim dany allel jest wyrażony w fenotypie populacji. Jeżeli każdy człowiek niosący dominujący zmutowany genu wykazuje fenotyp mutanta, mówi się że gen wykazuje pełną penetrację.
Genetyka molekularna: Badania dziedziczności, genów i DNA
Jak się dowiedzieliśmy DNA prowadzi projekt, który kieruje wszystkimi działaniami komórki oraz określa plan rozwoju organizmów wielokomórkowych. Dlatego zrozumienie DNA, struktury genów i podstawowych funkcji ma zasadnicze znaczenie dla oceny w biologii molekularnej komórki. Jednak ważne jest, aby uznać, że postęp w dziedzinie naukowej zależy od dostępności eksperymentalnych narzędzi, które pozwalają naukowcom do nowych obserwacji i prowadzenia nowatorskich eksperymentów. Ostatni odcinek tego działy kończy się omówieniem niektórych narzędzi laboratoryjnych i technologii, które umożliwiają naukowcom badania komórek i ich DNA.
Genetyka molekularna jest badaniem czynników, które przekazują informacji z pokolenia na pokolenie. Te cząsteczki, nasze geny, są długimi polimerami kwasu dezoksyrybonukleinowego lub DNA. Zaledwie cztery chemiczne cegiełki -guanina (G), Adenina (A), tyminy (T) i cytozyny (C) tworzą unikalny kod dla wszystkich genów we wszystkich organizmach żywych.
Geny określają dziedziczne cechy, takie jak kolor włosów lub oczu. Robią to poprzez dostarczenie instrukcji dla każdej działalności, jaka w każdej komórce naszego ciała powinna zostać przeprowadzona. Na przykład geny mogą nakazać komórkom wątroby usunąć nadmiar cholesterolu z naszej krwi. Jak geny to robią? Będą kierowały komórką aby wyprodukowała konkretne białka. Jest to białko, które następnie wykonuje rzeczywistą pracę. W przypadku nadmiaru cholesterolu we krwi, jest to białko receptora na zewnątrz z komórek wątrobowych, które wiążą się i usuwają cholesterol z krwi. Cząsteczka cholesterolu może następnie być transportowana do komórek, gdzie będzie przetwarzana przez inne białka.
Wiele chorób spowodowanych jest mutacjami, lub zmianami w sekwencji DNA genu. Gdy informacja kodowana przez zmieniony gen, powoduje to że białko może nie działać prawidłowo lub może nie być go w ogóle. W każdym przypadku, komórki zawierające zmiany genetyczne mogą nie zachowywać się zgodnie z oczekiwaniami. Teraz już wiemy, że mutacje w genach kodujące białko receptora cholesterolu związanego z chorobą o nazwie rodzinna hipercholesterolemia. Komórki osobnika z tą chorobą kończą się zmniejszoną funkcją receptora i nie może zostać usunięta wystarczająca ilość lipoprotein o niskiej gęstości (LDL), lub złego cholesterolu, z ich krwi. Osoba może wówczas mieć niebezpiecznie wysoki poziom cholesterolu, narażając się na zwiększone ryzyko zarówno ataku serca jak i udaru mózgu.
Jak naukowcy badają i znajdują te mutacje genetyczne? Mają dostęp do wielu narzędzi i technologii do porównywania sekwencji DNA izolowanych ze zdrowej osoby i tej samej sekwencji DNA od chorych osób. Zaawansowane technologie komputerowe, w połączeniu z wybuchem danych genetycznych uzyskanych z różnych projektów sekwencjonowania całego genomu pozwalają naukowcom na korzystanie z tych narzędzi genetyki molekularnej do diagnozowania chorób i projektowania nowych leków i terapii. Poniżej znajduje się przegląd pewnych powszechnych metod laboratoryjnych, których genetycy – naukowcy, którzy badają dziedziczenia struktury szczególnych cech - mogą używać w celu uzyskania i pracy z DNA, a następnie omówić niektóre z aplikacji.
Narzędzia i techniki laboratoryjne
Metody stosowane przez genetyków molekularnych pozwalające uzyskać i badać DNA zostały opracowane przez pragnie obserwacji i śledzenia reakcji chemicznych i procesów biologicznych, które występują naturalnie we wszystkich komórkach. Wiele z tych enzymów, które kopiują DNA, tworzą RNA z DNA i syntezują białka z matrycy RNA zostały po raz pierwszy w scharakteryzowane u bakterii. Te podstawowe wyniki badań stały się podstawą do zrozumienia funkcji komórek ludzkich i doprowadziły do ogromnego praktycznego zastosowania badań genu i jego białka. Na przykład, produkcja na dużą skalę białek jest obecnie niedrogim sposobem, aby uzyskać obfite ilości niektórych leczniczych czynników, takich jak insulina w leczeniu cukrzycy. Jako że następuje postęp nauki, tak też liczba dostępnych narzędzi, które mają zastosowanie do badań genetyki molekularnej.
Uzyskanie DNA do badań laboratoryjnych
Izolowanie DNA z pojedynczej komórki zapewnia kompletny zestaw wszystkich genów danej osoby, czyli są to dwie kopie każdego genu. Jednak wiele technik laboratoryjnych wymaga, aby naukowiec miał dostęp do setek tysięcy egzemplarzy danego genu. Jednym ze sposobów uzyskania tych wielu kopii jest izolacja DNA z milionów komórek sztucznie wyhodowanych w laboratorium. Inną metodą, zwaną klonowaniem, które wykorzystuje procedury manipulacji DNA do produkcji wielu kopii pojedynczego genu lub odcinka DNA. Łańcuchowa reakcja polimerazy (PCR) jest trzecią metodą, w której specyficzna sekwencja podwójnej nici DNA jest kopiowana lub amplifikowana. Amplifikacja PCR stała się niezastąpionym narzędziem w wielu różnych zastosowaniach.
Izolowanie DNA i mRNA z komórki
Hodowla komórkowa
Hodowla komórkowa obejmuje wzrost komórek w sztucznych warunkach, takich jak w laboratorium, gdzie dołącza się do pewnego rodzaju sztucznej nawierzchni lub zawieszona jest w specjalnym roztworze. W obu przypadkach, komórki kąpią się w płynach zawierających substancje odżywcze, które są produkowane syntetycznie lub pochodzącą z podobnych organizmów. Niektóre rodzaje komórek wykazują lepszy wzrost w kulturze niż inne. Na przykład, fibroblasty, rodzaj komórek skóry, były hodowane w laboratorium, przez dziesięciolecia, natomiast niuanse uprawy innych typów komórek, np. komórek nerwowych i komórek macierzystych dopiero niedawno zostały wyjaśnione. Warunki, które służą do podtrzymywania komórki jednego typu nie są stosowane się do innych typów komórek lub nawet tego samego typu komórek z innego gatunku. Warunki niezbędne do uprawy komórek ludzkich i wielu innych ssaków i roślin, na których nam zależy, zostały ustalone ogólnie, że warunki hodowli komórek z egzotycznych zwierząt i roślin wciąż wymagają doświadczenia dla każdego nowego gatunku.
Hodowla komórkowa jest przydatną techniką, gdyż stanowi odnawialne źródło komórek do izolowania DNA. Ponadto, naukowcy mogą użyć komórki hodowane w kulturach do badań, jak różne substancje chemiczne i leki wpływają na niektórych komórek i przez ekstrapolację, cały organizm. Proces wzrostu komórek poza żywym organizmem, tak jak w probówce, określany jest jako in vitro. Efekty czynników na komórki zostały dokładnie ocenione w badaniach in vitro, w poszukiwaniu bezpiecznego i skutecznego leczenia mogą być też badane w żywym organizmie, w procesie o nazwie badania in vivo.
Niektóre komórki zwykle tracą cenne właściwości lub mogą nawet umrzeć, jeśli trzymane będą w kulturze zbyt długo. Aby naprawić ten problem, naukowcy zamrażają dół linii komórkowej, tak aby mógł on być rozmrożony w późniejszym terminie do późniejszego wykorzystania. Proces ten wymaga stosowania chemikaliów krio-protektrantów, którzy chronią komórki i zapobiegają pęknięciu podczas procesu zamrażania i rozmrażania.
Izolacja DNA
Izolacji DNA odnosi się do procesu ekstrakcji DNA z komórki w stosunkowo czystej postaci. Obejmuje ona oddzielenie DNA od innych składników komórkowych, takich jak białka, RNA i lipidy. Komórki wykorzystywane do uzyskania i izolacji DNA mogą pochodzić bezpośrednio z tkanek lub mogą być hodowane z laboratoryjnych linii komórkowych pozyskanych przy pomocy metod opisanych wcześniej. Niezależnie od źródła DNA jest izolowane poprzez umieszczenie komórki w probówce zawierającej specjalny roztwór, zwany "koktajl", i mechanicznie lub chemicznie działając by ją otworzyć. Powoduje to, że zawartość komórki zostaje do uwolniona do koktajlu zawierającego enzymy, chemikalia i sole. Enzymy są wykorzystywane do trawienia białek; chemikalia do zniszczenia obecnych wszelkich RNA i sole do wyciągnięcia DNA z roztworu. W tym momencie DNA będzie występował jako długie nici, które wytrącają się w roztworze. DNA jest następnie zebranych przez wirowanie próby w maszynie zwanej wirówką. Podczas wirowania, DNA gromadzi na dnie probówki. Roztwór jest następnie wylewany, a DNA jest rozpuszczone w drugim roztworze, który ułatwi pracę w kolejnych procedurach. Skutkiem tego jest skoncentrowany DNA w próbce zawierającej tysiące kopii każdego genu. Dla analiz DNA na dużą skalę metody, takich jak te, które są wymagane do sekwencji ludzkiego genomu, izolacji DNA jest wykonywane przy użyciu robotów.
Izolacja mRNA
Wielu naukowców chce pracować z ekspresyjnym DNA lub DNA, które kierowane jest bezpośrednio na syntezę białek. Ten specjalny rodzaj DNA jest otrzymywana przez pierwsze izolowanie messenger RNA (mRNA), jest ono pośrednie między fragmentami ekspresyjnego DNA i produktem białkowym. Metody laboratoryjnej izolacji mRNA korzystają z normalnej komórkowej modyfikacji mRNA - dodanie do jednego końca cząsteczki mRNA 200 nukleotydów – jest to tzw. poli (A) ogon. W pierwszym kroku izolacji mRNA, komórka pęka i treści komórkowe są wystawiane na działanie syntetycznych kulek pokrytych ciągami nukleotydów tyminy.
Rysunek 2. Przykład izolacji mRNA.
Ten rysunek pokazuje, jak poli (A) RNA może być oddzielony od innych RNA przez wydzielenie na specjalnym stałym materialne. W tym przykładzie, materiał składa się z paciorków szklanych, do którego przyczepione są cząsteczki tyminy. Jak widać po lewej stronie diagramu, roztwór zawierający RNA różnych populacji, w tym mRNAs z poli (A) ogony (czerwony), jak również inne RNA i komórki (fioletowy), są nakładane na kolumnę rozdzielającą. Tylko poli (A) RNA jest zachowane, ponieważ zostaje unieruchomione na stałym materiale. Pozostałe RNA i komórek przechodzą przez kolumnę. Po prawej, związana poli (A) mRNA są usuwane przez traktowanie kolumny specjalnym roztworem buforowym, który rozdziela wiązanie nukleotydów tyminy z AAA nicią. mRNA mogą być gromadzone w probówce do dalszych eksperymentów.
Ponieważ Adenina i tyminy łatwo łączą się z sobą, poli (A) mRNA jest selektywnie zatrzymywane na kulkach natomiast pozostałe składniki komórkowe są wymywane. Wyizolowane, oczyszczone mRNA jest konwertowane do jednonicowego DNA przy użyciu enzymu odwrotnej transkryptazy a następnie do stabilnego dwuniciowego DNA przy użyciu enzymu polimerazy DNA. DNA produkowane w ten sposób jest komplementarnym DNA (cDNA), ponieważ jego sekwencja, przynajmniej pierwsza nic, jest komplementarna z mRNA, z którego on powstał. Dlaczego naukowcy natrafiają na kłopoty w tworzeniu cDNA? cDNA jest znacznie bardziej stabilne niż mRNA i, co ważniejsze, ponieważ zostało ono wygenerowane z mRNA, w którym nie ma regionów kodowania, zostały usunięte, cDNA reprezentuje jedynie sekwencja DNA podlegające ekspresji.
Odwrotna transkryptaza, enzym wymagany do formowania komplementarnej sekwencji DNA z sekwencji RNA, ma zasadnicze znaczenie dla przetrwania grupy wirusów o nazwie retrowirusy. Retrowirusy zawierają RNA zamiast DNA, jako ich materiał genetyczny. Odwrotna transkryptaza jest stosowana do produkcji kopii DNA wirusowego materiału genetycznego, niezbędnego elementu integracji w genom organizmu gospodarza. Chociaż większość retrowirusów nie jest uważana za korzystne dla ludzi, w laboratorium odwrotna transkryptaza jest nieocenionym narzędziem dla badaczy i leczenia niektórych z dolegliwości spowodowanych przez te wirusy.
Metody amplifikacji DNA
Klonowanie DNA w bakteriach
Klonowanie zrewolucjonizowało badania biologiczne w 1970 przez umożliwienie badania poszczególnych genów.
Słowo "klonowanie" może być wykorzystywane na wiele sposobów. W tym dokumencie odnosi się do uzyskania wielu dokładnych kopii danej sekwencji DNA. Aby stworzyć klon, sekwencja DNA jest dodawana się do tego, co nazywa się wektorem klonowania. Wektor klonowania jest cząsteczką DNA pochodzącą od wirusa, plazmidu lub komórki organizmu wyższego, w którym inny fragment DNA o odpowiedniej wielkości może zostać włączony bez ingerencji, musi on posiadać zdolność do samodzielnej replikacji. Docelowe i wektorowe fragmenty DNA są następnie ligowane lub łączone w całość, aby stworzyć to, co nazywa się rekombinantem DNA. Rekombinant DNA jest zwykle wprowadzany do Escherichia coli lub E. coli - powszechnym laboratoryjnym szczepem bakterii - przez transformację, naturalny mechanizm absorpcji DNA posiadany przez bakterie. W bakterii, wektor kieruje namnażanie rekombinowanego DNA, produkując kilka identycznych egzemplarzy. Wektorowy proces replikacji jest taki, że tylko jedno rekombinowane DNA może rozprzestrzenić się w ramach jednej bakterii, dlatego każdy powstający klon zawiera wiele kopii jednego insertu DNA. DNA może zostać odizolowane przy użyciu technik opisanych wcześniej.
Enzym restrykcyjny jest białkiem, który wiąże się z cząsteczką DNA o określonej sekwencji i czyni cięcie podwójnej nici na lub w pobliżu tej sekwencji. Enzymy restrykcyjne mają szczególne zastosowanie w różnych dziedzinach technik naukowych, takich jak manipulowanie cząsteczkami DNA podczas klonowania. Te enzymów mogą ciąć DNA na dwa różne sposoby. Wiele dokonuje prostych cięć podwójnej nici, dając sekwencję, która jest nazywana tępe lub równe końce.. Inni tną DNA dwuniciowe w różnych miejscach, zwykle tylko kilka nukleotydów obok siebie, co powoduje że DNA ma krótkie fragmenty jednoniciowego końca, zwanego lepkie lub spójne końce. Dzięki starannie wybieranie odpowiednich enzymów restrykcyjnych, pracownik może wyciąć docelową sekwencję DNA, otworzyć wektor do klonowania i połączyć dwa fragmenty DNA w celu utworzenia rekombinanta DNA.
Więcej o wektorach klonowania
Ogólnie rzecz biorąc, bakteryjny genom składa się z jednego, okrągłego chromosomu. Może on także zawierać znacznie mniejsze pozachromosomalne elementy genetyczne, zwane plazmidami, które różnią się od zwykłych genomów bakterii i nie są niezbędne dla przeżycia komórki w normalnych warunkach. Plazmidy są zdolne do kopiowania się niezależnie od chromosomów i łatwo przemieszczają się z jednej bakterii do innej. Ponadto, niektóre plazmidy są zdolne do integracji genomem gospodarza. To czyni je doskonałymi pojazdami lub wektorami, do transportowania docelowego DNA do gospodarza bakterii. Poprzez cięcie zarówno docelowego i plazmidowego DNA tym samym enzymem restrykcyjnym, komplementarne pary zasad są formowane na każdym fragmencie DNA. Fragmenty te mogą następnie łączyć się ze sobą, tworząc nowy kołowy plazmid zawierający docelowego DNA. Rekombinowany plazmid jest następnie przyjmowany przez gospodarza bakterii, gdzie jest kopiowany lub powielane, jakby to były normalne plazmidy.
Plazmidy bakteryjne były pierwszymi wektorami wykorzystywanymi do przekazywania informacji genetycznej i nadal są szeroko stosowane. Jednak ich stosowanie jest ograniczone czasem o wielkość docelowego DNA, mogą one przyjąć około 15000 zasad lub 15 KB. Z wielkością sekwencji DNA, sprawność wektora zmniejsza się, ponieważ ma problemy z wejściem w komórkę i replikowaniem się. Jednakże, zostały odkryte lub stworzone inne wektory, które mogą przyjąć większe docelowe DNA w tym: bakteriofagi, wirusy bakteryjne, które akceptują wstawki do 20 kB; kosmidy, zrekombinowane plazmidy z elementami bakteriofagów, które akceptują wstawki do 45 Kb; sztuczne chromosomy bakteryjne (BACS) przyjmują wstawki do 150 Kb; drożdże i sztuczne chromosomy (YACs), które akceptują wstawki do 1000 kb. Wiele wirusów zostało zmodyfikowanych do wykorzystania ich jako wektory klonowania.
Metoda łańcuchowej reakcji polimerazy (PCR)
Łańcuchowa reakcja polimerazy (PCR) jest zadziwiająco prostą technika, która prowadzi do wykładniczej amplifikacji niemal każdego regionu wybranego DNA. Działa ona w sposób, który jest podobny do replikacji DNA w przyrodzie. Podstawowym materiałem lub odczynniki wykorzystywanymi w PCR są:
Przygotowanie DNA do analizy doświadczalnej
Elektroforeza żelowa: Oddzielenie Cząsteczek DNA o różnych długościach
Początkowo białka były oddzielone na żelu wykonanego ze skrobi ziemniaczanej. Dzisiaj, żele są z agarozy lub syntetycznych polimerów, takich jak poliakrylamid.
Żele są zwykle wykonane z agarozy - łańcuchów cząsteczek cukru ekstrahowanych z wodorostów - lub innych syntetycznych cząsteczek. Oczyszczona agarowa jest ogólnie zakupywana w proszku, i rozpuszcza się ją we wrzącej wodzie. Kiedy roztwór jest jeszcze gorący, jest on wylewany w specjalnej aparaturze do odlewania, która zawiera trzy podstawowe części: tacę, podstawę i grzebień. Podstawa służy jako forma, która zapewnia kształt i rozmiar żelu. Zapobiega wyciekaniu agarozy z formy w czasie procesu zestalania. Jak tylko ciecz agarozy zaczyna być w zimnie, to podlega procesowi znanemu jako polimeryzacja. Zamiast pozostać rozpuszczony w wodzie, polimery cukru tworzą krzyżowe połączenia ze sobą, powodując że roztwór staje się żelem - półstałą matrix podobną do żelek, tylko bardziej zbitą. Podstawa także pozwala aby polimeryzowany żel został usunięty z formy bez zerwania. Zadaniem grzebienia jest generowanie małych dołków, do których próbki DNA zostaną załadowane.
Po tym jak żelu spolimeryzuje, jest odczepiany od tacy do odlewania, umieszczony w zbiorniku do elektroforezy i zanurzony w specjalnym wodnym buforze, zwanym bufor running. Aparatura w której znajduje się żel jest podłączana do źródła zasilania za pośrednictwem dwóch elektrod. Każda elektroda prowadzi do cienkiego drutu na przeciwnych końcach zbiornika. Ponieważ jedna elektroda jest pozytywna, a druga jest negatywna, silny prąd elektryczny będzie przepływał przez zbiornik, kiedy zasilanie będzie włączone.
Następnie próbki są rozpuszczone w niewielkiej objętości płynu zawierającego niewielką ilość glicerolu. Ponieważ glicerol ma większą gęstość niż woda, to służy do zwiększenia masy próbki i zatrzymuje ją na dole, kiedy próby zostaną załadowane do studzienki. Ponadto, ponieważ jest to pomocne, aby móc monitorować próbki DNA, gdy migruje w żelu, widoczne cząsteczki, zwane barwnikami, są również dodawane do próbki buforu. Te barwniki mają zwykle dwa różne kolory i dwie różne wagi molekularne lub rozmiary. Jeden z barwników jest zwykle mniejszy niż większość, jeśli nie wszystkie, fragmenty próby DNA i migrują szybciej niż najmniejsze próby DNA. Pozostały barwnik jest zwykle duży i będzie migrować z większymi fragmentami próbek DNA. Zakłada się, że większość fragmentów DNA będzie migrować gdzieś pomiędzy tymi dwoma barwnikami. Dlatego, gdy mały barwnik osiągnie koniec żelu, elektroforeza zwykle zostaje zatrzymana.
Gdy żel zostanie przygotowany i załadowany, zasilanie jest włączane. Prąd elektryczny przepływa przez żel i powoduje, że fragmenty DNA migrują w kierunku dołu lub pozytywnie naładowanego końca żelu. Dzieje się tak, ponieważ DNA jest negatywnie naładowane ze względu na połączenie cząsteczek w jej strukturze. Mniejsze fragmenty DNA mają mniej utrudnioną drogą przez sito utworzone w polimeryzowanym żelu niż większe cząsteczki. Oznacza to, że mniejsze fragmenty DNA zazwyczaj szybciej i dalej poruszają się w danym czasie. Skutkiem tego jest smuga lub gradient większych do mniejszych fragmentów DNA. W tych przypadkach, gdy wiele kopii DNA mają tę samą długość, koncentracja DNA występuje na tej pozycji w żelu, zwanej prążkiem. Prążki mogą wynikać z trawienia enzymem restrykcyjnym jednej próbki zawierającej tysiące kopii plazmidu DNA lub sekwencji DNA po amplifikacji PCR. Prążki DNA są następnie wykrywane przez namoczenie żelu krótko w roztworze zawierającym barwnik zwany bromkiem etydyny (EtBr). EtBr czynnikiem interkalującym, co oznacza, że jest zdolny do wciskania się w rowki DNA, gdzie on pozostaje. Im więcej par obecnych w fragmentach DNA, tym większa liczba dostępnych rowków dla EtBr. EtBr także fluoryzuje pod wpływem światła ultrafioletowego (UV). Dlatego też, jeśli żel zanurzymy w roztworze zawierającym EtBr pod źródłem promieniowania UV, to pracownik faktycznie może wykryć DNA przez wizualizację gdzie EtBr fluorescencji. Ponieważ naukowiec zawsze ładuje i uruchamia "kontrolną" próbę, która zawiera wiele fragmentów DNA o znanej wielkości, wielkość próby fragmentu DNA można oszacować porównując prążki kontroli i próby.
DNA Blotting
Porowaty i cienki charakter żelu jest idealny do oddzielania fragmentów DNA przy użyciu elektroforezy, ale jak wspomniano wcześniej, te delikatne żele są rzadko wykorzystywane do innych technik. Z tego powodu, DNA, które zostało rozdzielone przez elektroforezę jest przeniesione z żelem do łatwo obsługiwanej obojętnej membrany w procesie zwanym blotting. Określenie "blotting" opisuje pokrycie żelu membraną i stosowania z płytki, aby zapewnić kontakt, bez naruszania miejsc fragmentów DNA. W pierwszym kroku, DNA unieruchomione w żelu jest denaturowane - dwuniciowe DNA jest podzielony na pojedyncze nici przez moczenie żelu w roztworze alkalicznym. To pozwala DNA na hybrydyzację z sondą, fragmentem DNA, który jest komplementarny do sekwencji objętej dochodzeniem. Membrana, zwykle wykonana z mieszanki zwanej nitrocelulozą, jest umieszczana w górnej części żelu i skompresowana w wadze ciężkiej. DNA jest przekazywana z żelu na membranę zwykłymi siłami kapilarnymi. Procedura ta odtwarza dokładną strukturę DNA wytworzoną w żelu na membranę. Membrana może być następnie umieszczana z markerem DNA, w celu sprawdzenia obecności docelowej sekwencji.
Southern blotting jest nazwą procedury przekazywania denaturowanego DNA z żelu agarozowego do silnej stabilnej membrany. Procedura ta wykorzystuje specjalną własność nitrocelulozy, jego zdolność do wiązania bardzo silnie do jednoniciowego DNA, ale nie dwuniciowego DNA. Z drugiej strony, nothern blotting odnosi się do wszelkich procedur blottingu, w których elektroforeza odbywa się z użyciem RNA.
Tworzenie bibliotek DNA
Aby śledzić miliony lub nawet miliardy, nukleotydów w genomie organizmu, naukowcy często tworzą tzw. biblioteki DNA lub duży zbiór fragmentów DNA. Istnieje wiele różnych rodzajów bibliotek. Biblioteka genomiczna zawiera wszystkie różne rodzaje sekwencji DNA znajdujące się w genomie - introny, eksony i niekodujące i powtarzalne sekwencje DNA. Naukowcy również tworzą biblioteki wyłącznie genów, które są ekspresyjne, lub tych genów, które są przepisywane na messenger RNA i następnie przetłumaczone na białka. Ta biblioteka jest nazywana biblioteką komplementarnego DNA (cDNA) i często jest wykonana z mRNA ekspresyjnego w poszczególnych rodzajach tkanki. Biblioteka jest nazywana chromosomowo specyficzna, jeżeli początkowe DNA pochodzi z tylko jednego chromosomu.
Metody analizy DNA
Gdy DNA zostanie wyizolowany i oczyszczony, może być dalej analizowany na różne sposoby, np. w celu określenia obecności lub braku poszczególnych sekwencji nukleotydów lub zlokalizowania zmiany, zwanej mutacją, w określonej sekwencji.
Autoradiografia:: badanie DNA
Aby zlokalizować konkretną sekwencje DNA, naukowcy opierają się na parowaniu zasad lub hybrydyzacji krótkich fragmentów DNA, które są komplementarne do sekwencji ich interesujących. Ten krótki, jednoniciowy fragment DNA nazywa się "sondą" i może być oznaczony łagodnie promieniotwórczymi nukleotydami lub nukleotydy, które są związane z substancją, która emituje światło, gdy narażona jest na niektóre substancje chemiczne.
Jeśli odniesiemy się z powrotem do blottingu procedury opisanej wcześniej, wspomnieliśmy, że gdy docelowe DNA zostaje uwięzione w nylonowej membranie, membrana jest inkubowane w roztworze, który zawiera sondy. W tym przypadku, sondy były znakowane promieniotwórczo. Gdziekolwiek sekwencja sondy uzupełnia sekwencję na membranie, to będą się one łączyć, tworząc obszar dwuniciowego DNA.. Membrana jest później płukana, aby usunąć wszystkie niezwiązane sondy, a następnie nakładana na kawałku X-ray filmu. Wykrycie znakowanych promieniotwórczo cząsteczek przez ekspozycję na promieniowanie rentgenowskie wrażliwych filmów fotograficznych jest określane jako autoradiografia. Gdziekolwiek promieniotwórczo znakowana sonda związała się z badanym DNA, czarne miejsca będą pojawiać się na filmie.
Metoda ta jest przydatna do różnorodnych zastosowań. Na przykład, załóżmy, że znasz sekwencję DNA danego genu (allel), który powoduje choroby. Teraz chcesz wiedzieć, czy konkretny osobnik jest nosicielem tego allelu. Można to zrobić postępując zgodnie z procedurą opisaną powyżej. Izolując niektóre jego DNA. Oddzielając je na żelu. Następnie wykonać Southern blot, a później autoradiografię. Jeżeli czarne miejsca pojawią się w filmie, to wskazuje to na obecność choroby powodowanej przez ten allel.
Analiza RFLP: Wykrywanie chorób genów
Każdy osobnik ma niewielkie różnice lub sekwencje polimorficzne, które sprawiają, że ich sekwencja DNA jest niepowtarzalna. Te różnice są często pojedynczą zmianą zasady w parze, które występują w regionach DNA, które nie kodują genu, ale które są rozpoznawane przez enzymy restrykcyjne. Enzymy restrykcyjne są białkami, które wiążą się cząsteczki DNA o określonej sekwencji i tną podwójną nić na lub w pobliżu tej sekwencji. Tak więc, gdy DNA od dwóch różnych osób, będzie cięte jednym enzymem restrykcyjnym, będą produkowane DNA o różnej długości. Dzieje się tak, ponieważ nie wszystkie miejsca restrykcyjne istnieją u każdego DNA. Te różnice w długości fragmentu są określone jako ograniczenie długości fragmentu polimorficznego (RFLPs) oraz wzór fragmentów jest unikalny dla każdej osoby. Jeśli ograniczony polimorfizm może być związany z określonym fenotypem, takim jak kolor oczu, to jest to nazywane markerem restrykcji. Markery te są ważne, ponieważ oferują procedury diagnostyczne do wykrywania choroby i mogą pomóc naukowcom w izolacji genu.
Sekwencjonowanie DNA
Proces określania kolejności nukleotydów wzdłuż nici DNA nazywa się sekwencjonowaniem. W 1977 roku, 24 lata po odkryciu struktury DNA, dwie oddzielne metody sekwencjonowania DNA zostały opracowane: metoda terminacji łańcucha i degradacji chemicznej. Obie metody były równie popularne na początku, ale z wielu powodów, zakończenie łańcucha metoda terminacji łańcucha jest metodą powszechniej stosowaną. Metoda ta opiera się na zasadzie, że jednoniciowe, które różnią się długością tylko jednym nukleotydem mogą być oddzielone od siebie za pomocą elektroforezy na żelu poliakrylamidowym, opisanej wcześniej.
DNA które ma być sekwencjonowane, nazywa się matrycą DNA, jest najpierw przygotowywane jako jednoniciowe DNA. Następnie krótki oligonukleotyd jest dołączany lub przyłączany do tego samego miejsca na nici matrycy. Oligonukleotyd działa jak primer dla syntezy nowej nici DNA, która będzie komplementarna do matrycy DNA. Ta technika wymaga czterech reakcji nukleotydo-specyficznych – jedna dl każdej G, A, C i T - zostanie przeprowadzona w czterech identycznych próbach DNA. Cztery reakcje sekwencjonowania wymagają dodania wszystkich niezbędnych składników do syntezy i określania nowego DNA, w tym:
Po tym jak zostanie dodany pierwszy deoksynukleotyd do rosnącej komplementarnej sekwencji polimeraza DNA przemieszcza się wzdłuż matrycy i kontynuuje dodawanie zasad. Reakcja syntezy nici jest kontynuowana dopóki dideoksynukleotyd zostanie dodany, blokując dalsze wydłużenie. Dzieje się tak, ponieważ brakuje dideoksynukleotydowi specjalnej grupy w cząsteczce, tzw. 3'-hydroksylowej grupy, potrzebnej do stworzenia wiązania z kolejnym nukleotydem. Jedynie niewielka liczba zostaje dodanych dideoksynukleotydów do każdej reakcji, umożliwiając reakcje różnej długości czasu, przez szanse że polimeraza w różnym czasie wstawi dideoksynukleotyd, kończąc reakcję. Dlatego wynikiem jest szereg nowych łańcuchów, wszystkie o różnych długościach.
Aby przeczytać nowo generowaną sekwencję, cztery reakcje są uruchamiane side-by-side na sekwencjonującym żelu poliakrylamidowym. Rodziny cząsteczek wygenerowanych w obecności ddATP są ładowany do jednego paska żelu, a pozostałe trzy rodziny, generowane z ddCTP, ddGTP i ddTTP, są ładowane na trzy przylegające pasy. Po elektroforezie, sekwencje DNA mogą być odczytywane bezpośrednio z pozycji prążków w żelu.
Rysunek 3. Sieć zakończenie sekwencjonowania DNA.
Sekwencjonowanie metodą terminacji łańcucha powoduje syntezę nowych nici DNA komplementarnych do jednoniciowej matrycy (etap I). Matryca DNA jest dostarczana z mieszaniną wszystkich czterech deoksynukleotydów (każdy oznaczony sondą o innym kolorem fluorescencyjnym), polimerazą DNA (etap II). Ponieważ wszystkie cztery deoksynukleotydy są obecne, wydłużenie łańcucha trwa aż przez przypadek polimeraza DNA wstawi dideoksynukleotyd. Rezultatem jest nowy zestaw łańcuchów DNA, wszystkie o różnych długościach (etap III). Fragmenty te są następnie oddzielone ze względu na rozmiar za pomocą elektroforezy na żelu (etap IV). Ponieważ każdy znakowany fragment DNA posiada detektor na dole żelu kolor jest rejestrowany. Sekwencja DNA jest następnie rekonstruowana z wzorca kolorów reprezentujących każdy nukleotyd sekwencji (etap V).
Odmiany tej metody zostały opracowane do automatycznego sekwencjonowania wykorzystującego maszyny. W jednej metodzie, zwanej cykliczne sekwencjonowanie, dideoksynukleotydy, a nie primery, są oznaczone w różnymi kolorowymi barwnikami fluorescencyjnymi, zatem wszystkie cztery reakcje przebiegają w tej samej probówce i oddzielane są w tym samym pasie na żelu. Ponieważ każdy oznakowany fragment DNA przekazuje detektor na dole żelu, kolor jest rejestrowany, a sekwencja jest rekonstruowana z wzorca kolorów reprezentujących każdy nukleotyd w sekwencji.
Analiza chromosomu
Cytogenetyka to dziedzina nauki zajmująca się relacji między ludzkimi komórkami i ich chemicznymi blokami i dziedzicznością. Kluczem dla połączenia chromosomów do objawów i cech jest kariotyp, pokazujący kolejność par chromosomów pod względem wielkości. Pierwsze takie wysiłki mające na celu ułożenie par chromosomów, jednak były dość surowe. Po 1959 r., o wszystkim co mogłoby rozsądzić został dodatkowy chromosom lub jego brak. Przez rok 1960, pionierscy cytogenetycy gromadzili techniki przechwytywania chromosomów w ich najbardziej widocznym stanie. Dla większości komórkowych istnień materiał chromosomalny nie jest stanie wchłonąć barwników. Tylko w trakcie podziałów komórkowych chromosomy kondensują i stają się wykrywalne. Naukowcy dowiedzieli się, że traktowanie komórek hipotonicznym roztworem spowodowałoby ich zwinięcie, rozłożenie i zwinięcie w chromosom. Inny chemiczny znacznik, kolchicyna, był odkryty, że zatrzymuje podział komórkowy, kiedy chromosomy były w najbardziej zwiniętym stanie. Trzeci chemiczny związek, fituhemaglutynina, stwierdzono, że nęci limfocyty, komórki krwi najbardziej dostępne dla chromosomalnych badań, aby się podzielić. Z tymi narzędziami w ręku, sztuka kariotypowania został wkrótce przekształcona w prawdziwą naukę.
Ale wciąż, pary chromosomów nie zawsze było łatwo odróżnić i badacz miał polegać na takich dużych i subiektywnych wskazówkach jak wielkość chromosomu i położenie centromeru, charakterystycznie położonego w każdym chromosomie. Nawet barwienia chromosomów pozwalały jednoznacznie rozróżnić tylko 4 z 23 par chromosomów. Pary te zostały następnie pogrupowane według wielkości i zauważyć można było tylko duże działy dodatkowych lub brakujących chromosomów.
Przez 1970, przez łączenie plam z enzymami trawiennymi dawała dużo bardziej subtelne cienie wzorców, ujawniając charakterystyczne cechy każdego chromosomu. Różne zabiegi zostały również rozwinięte, i pozwoliło to naukowcom dalsze określenie wzorców każdego chromosomu. Teraz małe inwersje (odwrócenia w wąskim wzorcu), powielania, braki i translokacje (chromosomów, które utraciły części) mogą być wykryte. Ale budowanie kariotypu wymaga wielu godzin wykwalifikowanej pracy. Procedura kariotyping związana z uzyskaniem krwi lub innych odpowiednich tkanek, oddzielenie dzielących się komórek, ich wzrost w kulturze, mieszanie ich, a następnie umieszczanie na szkiełku mikroskopowym. Następnie przy użyciu mikroskopu świetlnego, naukowiec musiał znaleźć komórkę, w której wszystkie z niezwinięte chromosomy były obecne i zrobić zdjęcia. Zdjęcie następnie było rozwinięte, a poszczególne chromosomy zostały wycięte i rozmieszczone w parach według porządku wielkości w tabeli, określanej kariotypem. Dosłownie nożyczki i taśmy, oraz, wierz lub nie, wiele laboratoriów cytogenetyki nadal zależy głównie od tej metody analizy chromosomów. Ale teraz automatyczny analizator chromosomów - system, który obejmuje kamery, komputer, i mikroskop - może radykalnie poprawić szybkość i dokładność wyglądu chromosomów.
Fluorescencyjna hybrydyzacja in situ
Fluorescencyjna hybrydyzacja in situ (FISH), nowa metod analizy chromosomów, wykorzystuje fluorescencję cząsteczek, zwanych barwnikami, aby "malować" geny na chromosomie. Technika ta jest szczególnie przydatna dla mapowania genów oraz wykrycie różnych nieprawidłowości chromosomowych. W tej procedurze, krótkie sekwencje DNA komplementarne do sekwencji zainteresowania, zwane sondami hybrydyzują do próbki DNA. Ponieważ próby są oznaczone sondą fluorescencyjną, pracownik może zobaczyć dokładną lokalizację sekwencji DNA na chromosomie. Dodatkowym atutem jest to, że FISH może być wykonywane w niedzielących się komórkach, dzięki czemu ta metoda jest znacznie bardziej uniwersalna niż tradycyjny kariotyping.
Naukowcy mogą stworzyć trzy rodzaje sond FISH, z których każda ma inne zastosowanie. Sondy locus specyficzne hybrydyzują do danego regionu chromosomu i są przydatne do wykrywania lokalizacji genu na chromosomie. Alphoid lub sonda powtórzenia centromerowego, jest generowana w powtarzających się sekwencjach znajdujących się na centromerach chromosomów. Ponieważ każdy chromosom może być pomalowany na inny kolor, naukowcy korzystają z tych sond w celu ustalenia, czy dana osoba ma prawidłową liczbę chromosomów. Sondy całego chromosomu są faktycznie zbiorami mniejszych sond, zwanych bibliotekami, z których każda hybrydyzuje do innej sekwencji wzdłuż tego samego chromosomu. Korzystając z tych bibliotek, naukowcy mogą malować cały chromosom różnymi kolorami, wytwarzając to, co nazywane jest spektralnym kariotypem. Tego typu sondy są przydatne dla rozpatrywania dużych i małych nieprawidłowości chromosomowych.
Hybrydyzacja komórki somatycznej
Określenie "somatyczne" komórki odnosi się do wszystkich komórek w organizmie, które są zróżnicowane na konkretny rodzaj komórek, z wyłączeniem komórki zarodkowej, komórek macierzystych i gamet. Hybrydyzacja komórki somatycznej jest techniką połączenia dwóch komórek z różnych tkanek lub gatunków w hodowli komórkowej, zazwyczaj u ludzi i gryzoni, z zamiarem połączenia różnych linii komórkowych, z których każda posiada inną kombinację chromosomów. Hybrydyzowane komórki są połączone, ale ich jądra zazwyczaj pozostają odrębne. Jednak podczas podziału komórki, jedno wrzeciono jest tworzone tak, że każda komórka potomna ma jedno jądro zawierające zestawy chromosomów z obu linii rodzicielskich. Podczas wzrostu i podziałów komórek hybrydowych, mają one tendencję do losowej utraty wielu swoich chromosomów, dopóki nie osiągną stabilnego punktu. Odtąd obecnie, komórka będzie utrzymywała tą samą liczbę i gatunek chromosomów w kolejnych podziałach. Niewiele wiadomo na temat mechanizmów odpowiedzialnych za ten proces, ale z jakiegoś powodu, hybrydy między ludźmi i gryzoniami zazwyczaj gubią większość ludzkich chromosomów, dopóki tylko od 8 do 12 oryginalnych 46 ludzkich chromosomów pozostanie. Jednak w jakiś sposób te komórki mogą jeszcze przeżyć. Dzięki starannej izolacji i hodowli różnych hybrydowych linii komórkowych, naukowcy mogą tworzyć szereg komórek somatycznych mieszańców, które łącznie zawierają cały zestaw chromosomów człowieka. Naukowcy mogą następnie korzystać z tych linii komórkowych do przeszukania na obecność lub brak genu lub produktu genu (białka). Na przykład, naukowiec może przetestować możliwości komórek do metabolizowania danej substancji lub badać cechy odporności na antybiotyki. Jeśli linii komórkowa wykazuje efekt, naukowiec może następnie badać chromosomy obecne w danej linii komórkowej do identyfikacji genu, który daje pożądany efekt.
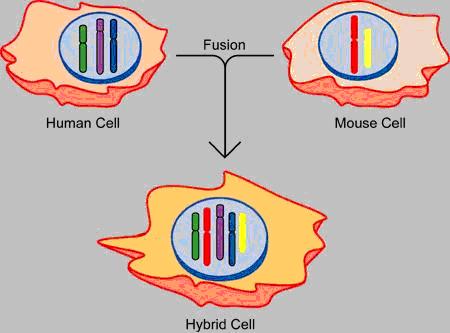
Rysunek 4. Hybrydyzacja komórek somatycznych.
Komórki ludzkie (z lewej) i komórki myszy (po prawej), uprawiane w hodowli, mogą ulec fuzji przez traktowanie wirusem lub związkiem chemicznym tworząc coś co nazywa się komórką hybrydową (u dołu), zawierającą zarówno chromosomy ludzkie i myszy. W tym przykładzie trzy ludzkie chromosomy (zielony, fioletowy i niebieski, z zarysie) i dwa chromosomy myszy (czerwony i żółty, bez nakreśla) są pokazane, a komórka hybrydowa jest miksem pięciu chromosomów.
Wpływ genetyki molekularnej
Większość technologii sekwencjonowania i analizy została opracowana z badań nie ludzkich genomów, zwłaszcza bakterii Escherichia coli, drożdży Saccharomyces cerevisiae, muszki owocowej Drosophila melanogaster, obleńca Caenorhabditis elegans, oraz myszy laboratoryjnej Mus musculus. Te prostsze systemy dostarczają doskonałych modeli rozwoju i testów niezbędnych procedur do badań znacznie bardziej złożonego ludzkiego genomu.
Duża ilość informacji genetycznej już otrzymanych z tych organizmów, dostarczyła cennych danych do analizy regulacji normalnych ludzkich genów, chorób genetycznych i procesów ewolucyjnych. Na przykład, naukowcy już określili pojedyncze geny związane z licznymi chorobami, takimi jak Cystic Fibrosis. W toku badania, badacze również odsłonią mechanizmy chorób spowodowanych przez kilka genów lub interakcje pojedynczych genów z czynnikami środowiskowymi. Genetyczne podatności zostały wzięte pod uwagę w udziale w wielu poważnych, śmiertelnych chorobach w tym chorób serca, udaru mózgu, cukrzycy i wielu rodzajów nowotworów. Identyfikacja tych genów i białek utoruje drogę do bardziej skutecznej terapii i środków zapobiegawczych. Badacze podkreślają biologię organizacji genomu i regulacji genów, które również zaczynają rozumieć, w jaki sposób wpływają na ludzi, dlaczego ten proces jest czasem na opak, a jakie zmiany następują w jakim wieku.
Niekorzystne reakcje na lek. Te trzy proste słowa przekazują trochę horroru o ciężkiej negatywnej reakcji na przepisany lek. Takie negatywne reakcje mogą jednak występować. 1998 badanie hospitalizowanych pacjentów opisane w Journal of the American Medical Association poinformowało, że w 1994, niewłaściwa reakcja na lek spowodowała więcej niż 2,2 miliona poważnych przypadków i ponad 100 000 śmierci, powodując iż niewłaściwa reakcja na lek (adverse drug reactions – ADRs) stała się jedną z głównych powodów hospitalizowania i śmierci w USA. Obecnie, nie istnieje prosty sposób, aby ustalić czy ludzie będą dobrze, źle czy wcale reagować na lek, dlatego firmy farmaceutyczne są ograniczone do opracowywania leków używając systemu „jeden pasuje do wszystkich. Ten system pozwala na opracowanie leków, na który „przeciętny” pacjent będzie reagował. Jednak jak pokazują statystki, jeden rozmiar nie pasuje do wszystkich, czasami z druzgocącymi skutkami. Potrzebnym jest opracowanie drogi do rozwiązania problemu z ADRs, zanim one się pojawią. Rozwiązanie jest w zasięgu wzroku i jest nazywane farmakogenomiką.
Sposób w jaki dana osoba reaguje na leki (w tym zarówno pozytywna i negatywna reakcja) to kompleksowa reakcja, która jest powodowana przez wiele różnych genów. Nie znając wszystkich genów zaangażowanych w odpowiedź na leki, naukowcy odkryli, że trudno opracować testy genetyczne, które mogłyby przewidzieć odpowiedź osoby, na poszczególne leki. Kiedy naukowcy odkryli, że ludzkie geny wykazują drobne wahania (lub zmiany) w zawartości nukleotydów, wszystko to się zmieniło – genetyczne testy w przewidywaniu odpowiedzi na lek są teraz możliwe. Farmakogenomika jest nauką, która analizuje odziedziczalne zmiany w genach, które dyktują odpowiedź na leki i wykorzystują drogi tych zmian, aby przewidywać czy pacjent będzie odpowiadał dobrze na leki, źle, czy wcale.
Czy istnieje różnica między farmakogenetyka a farmakogenomiką?
Farmakogenomika odnosi się do ogólnych badań wielu różnych genów, które określają zachowanie się leków.
Farmakogenetyka odnosi się do badania odziedziczalnych zmian w metabolizmie leków i odpowiedzi na nie.
Rozróżnienie między tymi dwoma dziedzinami jest arbitralne, jednak oba te terminy są używane zamiennie.
Jak zmienność genów może być wykorzystana w przewidywaniu odpowiedzi na leki?
Sekwencjonowanie DNA to określenie kolejności nukleotydów w DNA.
Już teraz, istnieje wyścig do katalogu wszystkich znalezionych zmienności genetycznych w genomie ludzkim. Zmienności te, albo SNPs (wymawiane „snips”), jak są powszechnie nazywane, mogą być wykorzystane jako narzędzie diagnostyczne do przewidywania reakcji osoby na lek. Aby SNP mogły być użyte w ten sposób, DNA osoby, musi zostać zanalizowane (zsekwencjonowane) na obecność specyficznych SNP. Problemem jest jednak to, że tradycyjne sekwencjonowanie genów jest bardzo powolne i drogie i dlatego utrudnia powszechne użycie SNP jako narzędzia diagnostycznego. Mikromacierze DNA (lub DNA chipy) są rozwijającymi się technologiami, które powinny sprawić aby dla lekarzy możliwa i szybka stała się analiza pacjentów na obecność specyficznych SNP. Jedna mikromacierz może być użyta do wykrycia 100 000 SNP znalezionych w genomie pacjenta w ciągu kilku godzin. Technologia mikromacierzy jest nadal rozwijana, wyszukiwanie SNP w gabinecie lekarza aby określić reakcję pacjenta na lekarstwo, przed przepisaniem recepty, stanie się powszechne.
Jak rozwój leków i testowanie mogą być korzyścią dla farmakogenomiki?
Wstępne badania kliniczne powinny być mniejsze, szybsze i mniej kosztowne, po to, aby konsument mógł korzystać z obniżonych kosztów leków.
Wyszukiwanie SNP skorzysta z rozwoju leków i testowania, ponieważ firmy farmaceutyczne mogą wyłączyć badania kliniczne tych ludzi, które wyszukiwanie farmakogenomiczne mogło by pokazać że testowane leki byłyby nieskuteczne lub szkodliwe dla ludzi. Bez tych ludzi, zwiększy się szansa, że leki okażą się użyteczne dla poszczególnych grup populacji oraz tym samym wzrasta szansa, że te same leki zostaną wprowadzone na rynek. Wreszcie, dzięki zdolności do indywidualnej oceny reakcji na leki, przed tym zanim zostaną przepisane, wzrośnie pewność lekarzy do przepisywania leków i pewność pacjentów w braniu leków, co z kolei powinno zachęcić do opracowania nowych leków testowanych w podobny sposób.
Jaka jest rola NCBI w farmakogenomice?
Eksplozja obu – SNP i danych mikromacierzy wygenerowane z projektu ludzkiego genomu, wymusiły opracowanie sposobu katalogowania i opisu (krótko opisujące) tych danych, tak aby naukowcy mogli mieć łatwiejszy dostęp i używać ich w swoich badaniach. NCBI, zawsze na topie w badaniach bioinformatycznych, opracowała bazę danych repozytoriów dla SNP (dbSNP) oraz danych mikromacierzy (GEO). Te bazy danych zawierają informacje opisowe o danych na stronie internetowej oraz linki do NCBI oraz zasoby zewnętrznych informacji. Dostęp do tych danych oraz zasobów informacji pozwoli naukowcom łatwiej interpretować dane, które są używane nie tylko w pomocy określenia reakcji na lek, ale do badań wrażliwości chorób i prowadzenie podstawowych badań w dziedzinie genetyki populacji.
Obietnica farmakogenomiki
W tej chwili, w gabinetach lekarzy na całym świecie, pacjenci dostają leki, które nie działają lub mają efekty uboczne. Często zdarza się, że pacjent musi wracać ciągle do lekarza aż doktor znajdzie lek, który będzie dla niego odpowiedni. Farmakogenomika oferuje bardzo atrakcyjną alternatywę. Wyobraź sobie dzień, kiedy idziesz do gabinetu lekarza i po prostym i szybkim teście Twojego DNA, lekarz zmienia swoje zdanie co do leku rozpatrywanego dla Ciebie, ponieważ Twój test genetyczny pokazał, że możesz ponieść poważne negatywne skutki tych leków. Jednakże, przez dalszą analizę Twoich badań, lekarz stwierdza, że możesz odnieść korzyści stosując nowy lek, i że istnieje niewielkie prawdopodobieństwo, że będziesz reagować na nie negatywnie. Dzień jak ten opisany zawita niedługo w gabinecie Twojego lekarza, dzięki farmakogenomice.
Rozpoczęcie
Potrzebujesz pomocy z dbSNP lub Map Viewer? Co powiesz na szybki „jak” aby używać innych narzędzi danych NCBI? Spróbuj „Getting Started”, nowych zasobów NCBI zaprojektowanych aby pomóc początkującemu użytkownikowi:
- krótki opis danych, które mogą być znalezione/ manipulowane używając poszczególnych narzędzi NCBI
- skróty aby dostać się gdzie każdy potrzebuje się dostać
- zwięzłe opisy narzędzi graficznych NCBI z poufnymi technikami powiązanymi z bazą danych
- proste przykłady użycia narzędzi
Klasyfikacja organizmów
Czy kiedykolwiek zauważyłeś, że kiedy widzisz owada lun ptaka, istnieje prawdziwa przyjemność w nazywaniu go oraz niewygodna niepewność kiedy nie możesz?Wzdłuż tych samych linii, rozważ zawrotną liczbę i różnorodności organizmów, które żyją albo żyły, na tej planecie. Jeśli nie wiesz jak nazywać te organizmy, jak można by komunikować pomysły na ich temat, nie mówiąc już o historii życia? Dzięki taksonomii, dziedzina nauki, która klasyfikuje organizmy żywe w grupy, możemy dyskutować o każdym organizmie, od bakterii do człowieka.
Karol Linnaeusz był pionierem w grupowaniu organizmów, opartym na naukowych nazwach z wykorzystaniem łaciny. Jego system nazywania organizmów składał się z dwóch części, czasami więcej, nazywany jest dwumianową nomenklaturą albo „nazywaniem dwu-słownym”. Jego program był oparty na fizycznych podobieństwach i różnicach, referowanych jako znaki. Dziś, klasyfikacja taksonomiczna jest dużo bardziej skomplikowana i bierze pod uwagę typy komórkowe i organizację biochemicznych i genetycznych podobieństw. Taksonomia jest tylko jednym z tematów, dla dużo większej dziedziny nazywanej systematyką.
Taksonomiczna klasyfikacja
Taksonomiczne szeregi przybliżają ewolucyjną odległość pomiędzy grupami organizmów. Na przykład gatunki należące do dwóch różnych nadkrólestw, bardziej związanymi (ich wspólny przodek odbiegał w ich odległej przeszłości, oraz stopniowe bardziej ekskluzywne grupy wliczając gromadę, klasę i tak dalej, niżej do mniej specyficznych szeregów lub szeregów występujących z gatunkami. Mniej specyficzne szeregi, takie jak podgatunek, odmiana i forma, oznacza najbliższy związek ewolucyjny. Zobacz uproszczoną klasyfikację człowieka poniżej.
Taksonomiści, naukowcy którzy klasyfikują żywe organizmy, określają gatunek jak każdą grupę bliskich powiązań organizmów, która może wydawać potomstwo. Dwa organizmy są bardziej podobne, jak znajdują się w tym samym gatunku, co znaczy że mają więcej wspólnych genów. Poziom gatunku może być podzielony na mniejsze segmenty. Populacje są mniejszą jednostką gatunki i są ono tworzone przez organizmy tego samego gatunku. Czasami, populacja będzie fizycznie zmieniać się z upływem czasu, aby odpowiadać potrzebom środowiska. Jest to tzw. Cline i może on sprawić że członkowie tego samego gatunku wyglądają inaczej.
Homo sapiens
Nadkrólestwo: Eukaryota
Królestwo: Metazoa
Typ: Chorwata
Klasa: Mammalia
Rząd: Primata
Rodzina: Hominidae
Rodzaj: Homo
Gatunek: sapiens
Karol Linnaeusz był również pomysłodawcą pionierskiej systematyki, dziedziny nauki zajmującej się różnorodnością życia i powiązań między komponentami życia. Systematyka wykracza poza taksonomię, bo wyjaśnia nowe metody i teorie, które mogą być użyte do klasyfikacji gatunków opartej na podobieństwie cech i możliwych mechanizmach ewolucji, zmiany w populacji genów populacji w czasie.
Filogenetyczna systematyka jest dziedziną biologii, która zajmuje się identyfikacją i zrozumieniem powiązań ewolucyjnych pomiędzy wieloma różnymi formami życia na ziemi, zarówno żywymi (istniejącymi) jak i martwymi (wymarłymi). Teoria ewolucji mówi, że podobieństwa pomiędzy jednostkami a gatunkiem są warunkiem posiadania wspólnego pochodzenia lub dziedziczenia od wspólnego przodka. Tak więc, ustanowione powiązania przez filogenetyczną systematykę często opisują historię ewolucyjną gatunków, a tym samym jego filogenezę, historyczne związki pomiędzy liniami albo organizmami albo ich częściami, takimi jak ich geny.
Charles Darwin był pierwszym, który rozpoznał że systematyczna hierarchia stanowiła duże zbliżenie do historii ewolucji. Jednak dopiero w 1950 roku niemiecki entomolog Will Hennig zaproponował, że systematyka powinna odzwierciedlać znaną historię ewolucyjną linii tak blisko jak to możliwe, podejście to nazwał filogenetyczną systematyką. Naśladowcy Hennig’a pogardliwie określani „kladystami” przez swoich przeciwników, ze względu na nacisk na rozpoznawanie tylko monofiletycznych grup, grupy oraz wszystkich jej potomków, lub klady (organizmy pochodzące od wspólnego przodka). Jednakże, kladyści szybko przyjęli, że pojęcie jako pomocną etykietę, a obecnie, kladystyka podchodzi do systematyki stosunkowo rutynowo.
Zmienność genetyczna: Zmiany w puli genetycznej
Ewolucja nie zawsze jest dyskretna, o jasno zdefiniowanych granicach, aby wskazać pochodzenie nowego gatunku, ani stałą kontynuacją. Ewolucja wymaga genetycznych zmian, które powodują zmiany w puli genetycznej, genetyczne posiadanie w konkretnej populacji. Genetyczna pula jest kombinacją wszystkich alleli – alternatywnych form genetycznego locus – dla wszystkich cech, które populacja może ujawniać. Zmiany w puli genowej, mogą być powodowane przez mutacje – zmiany w pojedynczych genach – albo przez zmiany w częstotliwości genów – proporcje allelu w danej populacji.
Jak występuje zmienność genetyczna?
Każdy organizm posiada genom, który zawiera biologiczną informację niezbędną do zbudowania i utrzymania żywego przykładu tego organizmu. Biologiczna informacja zawarta w genomie jest zakodowana w sekwencji nukleotydowej DNA albo RNA oraz jest podzielona na podjednostki nazywane genami. Informacja przechowywana w genie jest czytana przez białka, które łączą się z genomem i powodują serie reakcji nazywanymi ekspresja genów.
Za każdym razem, gdy komórka dzieli się, musi doprowadzić to do powstania kompletnej kopii genomu, proces ten nazywany jest replikacją DNA. Replikacja DNA musi być bardzo dokładna, aby uniknąć powstania mutacji, albo zmian w sekwencji nukleinowej krótkiego regiony genomu. Z całą pewnością, niektóre mutacje występują, zazwyczaj w dwóch sposobach: albo w skutek pomyłek w replikacji albo w skutek niszczących efektów chemicznych substancji albo promieniowania, które reaguje z DNA i zmienia strukturę pojedynczych nukleotydów. Wiele z tych mutacji jest rezultatem zmian, które nie mają wpływu na funkcjonowanie genomu, są to ciche mutacje. Ciche mutacje obejmują praktycznie wszystkie zmiany które występują w niekodujących składnikach genów i związanymi z genami sekwencjami.
Mutacje w regionach kodujących genów są dużo ważniejsze. Musimy tu rozważyć ważność tych samych mutacji w komórkach somatycznych w porównaniu z komórkami linii zarodkowych. Komórką somatyczną jest każda komórka organizmu inna niż komórki rozrodcze, takie jak nasienie albo komórka jajowa. Linia komórek zarodkowych jest każda komórka, z której powstaje gameta i kontynuuje przez pokolenia. Ponieważ komórka somatyczna nie przechodzi kopiowania swojego genomu do następnego pokolenia, jest ona tylko ważna w organizmie, w którym się znajduje i nie ma potencjalnego wpływu na ewolucję. W rzeczywistości większości somatycznych mutacji nie ma znaczących efektów, ponieważ istnieje wiele identycznych komórek w tej samej tkance.
Z drugiej strony mutacje w komórkach rozrodczych mogą być przekazywane następnym pokoleniom i następnie występować w wszystkich komórkach osoby, która dziedziczy tą mutację. Nawet jeszcze mutacje w linii komórek rozrodczych mogą nie zmieniać fenotypu organizmu w znaczący sposób. Te mutacje, które mają wpływ na ewolucję mogą dzielić się na dwie kategorię, mutacje utraty funkcji i mutacje zyskujące funkcje. Mutacja straty funkcji powoduje zmniejszenie lub zniesienie funkcji białka. Mutacje zyskujące funkcje, które są dużo powszechniejsze, powodują nieprawidłowe działanie białka.
Przypadkowe mutacje mogą powodować znaczącą rolę w biologii oraz są wymogiem Darwina w jego świetle ewolucji, która uważa te zmiany za charakterystykę organizmu powodowanego przez szansę oraz nie mającą wpływu na środowisko, w którym żyje organizm. Korzystne zmiany w organizmie są następnie pozytywnie wybierane, natomiast szkodliwe zmiany są negatywnie wybierane.
Kierowanie ewolucją: selekcja, sens, oraz efekt odkrywcy
Właśnie omówiliśmy, że allele pojawiają się w populacji przez mutacje, które pojawiają się w komórkach rozrodczych organizmu. To znaczy ze wiele genów jest polimorficznych, to znaczy, że dwa lub więcej alleli genów jest obecnych w populacji. Każdy z tych alleli, ma swój własny allel oraz frekwencję genu, jest to miarą jak powszechny jest ten allel w populacji. Częstotliwość allelu różni się w czasie ze względu na dwa warunki, naturalną selekcję oraz losowe zmiany.
Naturalna selekcja
Naturalna selekcja jest procesem, w którym z jednego genotypu, indywidualnej kontynuacji dziedziczenia, daje więcej potomstwa niż inny genotyp, ponieważ ma najwyższe atrybuty życia, określane kondycją. Naturalna selekcja działa na zasadzie zmienności genetyczne przez przyznanie korzyści przetrwania dla tych osobników posiadających daną mutację, powodującą tendencję do przystosowania się do zmieniających się warunków środowiska. Osobniki te, następnie rozmnażają się i przekazują nowy gen, zmieniając pulę genów. Selekcja naturalna, zmniejsza więc frekwencję allelu, który redukuje kondycję organizmy oraz zwiększa frekwencje alleli, które podnoszą kondycję.
Naturalna selekcja jest to zasada według której, każda najmniejsza zmiana, jeśli jest użyteczna, zostaje zachowana.
Ważnym punktem, który powoduje że naturalna selekcja nie zawsze stanowi postęp, tylko adaptację do zmieniającego się otoczenia, jest to, że ewolucje cechuje naturalna selekcja jest pozbawiona intencji – czasami nie powoduje polepszenia, tylko adaptację. Ponieważ środowisko ciągle się zmienia, co było kiedyś korzyścią mutacji, ponieważ mogły się one stać odpowiedzialne za dalsze zmiany ewolucyjnych linii.
Losowe zmiany
Termin losowe zmiany aktualnie obejmuje szereg odległych procesów, czasami określanych jako przybyłe z zewnątrz. Obejmują one masowe próby rodzica, efekt odkrywcy, oraz fluktuacje stopy w procesie ewolucji takie jak selekcja, migracja czy mutacje. Wybieranie rodzica jest procesem determinującym, który organizm jednego pokolenia będzie rodzicem następnego pokolenia. Może on dyskryminować, to znaczy, w odniesieniu do różnic kondycji, albo nie dyskryminować (być przypadkowy), bez względu na różnice kondycji. Dyskryminowanie próby rodzica jest przeważnie powodowane naturalną selekcją, podczas gdy nie dyskryminacja jest powodowana losowymi zmianami.
Co to jest próba (próbkowanie)?
Załóżmy że populacja czerwonych i brązowych wiewiórek dzieli środowisko ze ślepym drapieżnikiem. Chociaż drapieżnik jest niewidomy, brązowe wiewiórki umierają w większej liczbie niż czerwone, sugeruje to że brązowe wiewiórki nie mają po prostu szczęścia i są w kontakcie z drapieżnikiem częściej. W rezultacie, frekwencja brązowych wiewiórek w przyszłym pokoleniu jest zmniejszona. Więcej czerwonych wiewiórek przetrwa aby rozmnożyć się albo próbkować, ale jest to bez względu na jakieś różnice w kondycji między tymi dwoma grupami. Fizyczne różnice tych grup nie odkrywają znaczącej roli w różnicach sukcesu reprodukcyjnego.
Teraz, możemy stwierdzić że drapieżnik nie jest niewidomy i może widzieć czerwone wiewiórki lepiej niż brązowe, rezultatem będzie lepszy wskaźnik przetrwania u brązowych wiewiórek. W tym przypadku było by to dyskryminowanie próby rodzica albo naturalna selekcja.
Efekt odkrywcy
Inna przyczyną zmienności genetycznej jest efekt założyciela, różnica między pulą genów w populacji jako całości, a populacją nowo wyizolowaną tego samego gatunku. Efekt odkrywcy występuje wtedy gdy populacja zaczyna od małej liczby indywidualnych założycieli jednej oryginalnej populacji. Ze względu na mała liczbę osobników, nowa populacja może posiadać więcej różnic genetycznych w stosunku do pierwotnej populacji. Przykładem efektu odkrywcy było by, wtedy gdy populacja roślin wyrasta z jednego materiału siewnego.
Do tej pory, omówiliśmy naturalną selekcję oraz zmienność losową jako wydarzenia które występują w oderwaniu od siebie. Jednakże, w większości populacji, oba procesy będą występowały w tym samym czasie. Co więcej, istnieje wielka debata czy, w poszczególnych przypadkach, czy w ogóle, naturalna selekcja jest bardziej powszechna niż losowa zmienność.
Systematyka opisuje wzór powiązań między taksonami i ma na celu pomoc w rozumieniu historii życia. Ale historia nie jest czymś co możemy zobaczyć – zdarzyła się ona raz i pozostawia jedynie wskazówki co do rzeczywistych wydarzeń. Naukowcy korzystają tych wskazówek do budowania hipotez, albo modeli historii życia. W badaniach filogenetycznych, najbardziej dogodny sposób do wizualizacji powiązań ewolucyjnych wśród grup organizmów jest ilustracja nazywana drzewem filogenetycznym.
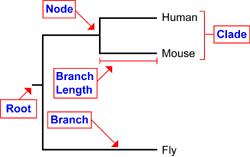
Node (węzeł): reprezentuje taksonomiczną jednostkę. Może to być albo istniejący gatunek albo przodek.
Branch (gałęzie): definiuje powiązania między taksonami w zakresie odległości i pochodzenia.
Topologia: wzór odgałęzień drzewa
Branch length (długość gałęzi): reprezentuje liczbę zmian, które nastąpiły na gałęziach
Root (źródło): wspólny przodek wszystkich taksonów
Skala odległości: skala, która reprezentuje liczbę różnic pomiędzy organizmami a sekwencjami.
Blade: grupa dwóch lub więcej taksonów albo sekwencji DNA, która zawiera zarówno ich wspólnego przodka oraz ich przodków.
Jednostki operacyjne taksonomi: taksonomiczny poziom próbkowania wyszukanych przez użytkownika, które mają być stosowane w badaniach, takich jak osoby, populacje, gatunki, rodzaje lub szczepy bakteryjne.
Drzewo filogenetyczne składa się z nodes (węzłów), każdy reprezentuje jednostkę taksonomiczną (gatunek, populacje, osobniki) oraz branches (gałęzi), które wyjaśniają powiązania pomiędzy jednostkami taksonomicznymi w zakresie odległości i pochodzenia. Tylko jedna gałąź może łączyć dwa sąsiednie węzły. Wzór odgałęzień drzewa nazywany jest topologią, a długość gałęzi zwykle reprezentuje zmiany, które występują na gałęzi. Nazywane jest to skalowaniem gałęzi. Skalowanie drzew jest często skalibrowane tak by reprezentować upływ czasu. Takie drzewa mają teoretyczne podstawy w poszczególnym genie lub genach podczas analizy. Gałęzie mogą być również (unscaled) nieskalowane, co znaczy że długość gałęzi nie jest proporcjonalna do liczny zmian, która miała miejsce, choć liczba rzeczywista mogła być wskazana gdzieś na gałęzi. Drzewa filogenetyczne mogą także być ze źródłem lub bez źródła. W drzewach ze źródłem, są szczególne węzły, nazywane źródłami, reprezentująca wspólnego przodka, z którego niepowtarzalna ścieżka prowadzi do każdego innego węzła. W drzewach bez źródła występują tylko powiązania między gatunkami, bez identyfikacji wspólnego przodka albo ewolucyjnej ścieżki.
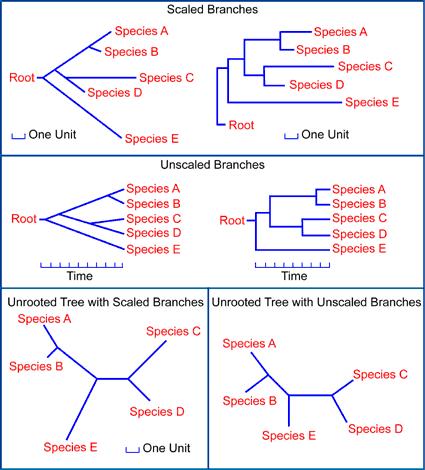
Filogenetyka drzew, wygodnym sposobem do prezentacji powiązań ewolucyjnych wśród grupy organizmów, może być przedstawiany na różne sposoby. Gałęzie na drzewie mogą być skalowane (panel na górze) reprezentuje liczbę zmian ewolucyjnych, czas oraz oba, gdzie jest zegar molekularny, oraz może być nieskalowany (panel środkowy) i nie mają bezpośredniego powiązania do wielkości zmian ewolucyjnych. Drzewa mogą być ze źródłem (środkowe i górne panele) oraz bez źródła (panel na dole). W przypadku drzew bez źródła, związki gałęzi między taksonami są specyficzne w sposób, w który są powiązane ze sobą, ale nie jest zaznaczona pozycja źródła. Na przykład, na drzewie bez źródła jest pięć gatunków, jest więc pięć gałęzi (cztery zewnętrzne, jeden wewnętrzny) na których drzewo może mieć przodka. Źródło na każdej z pięciu gałęzi ma różne implikacje dla powiązań ewolucyjnych.
Dwie główne grupy analizy istnieją w celu zbadania powiązań filogenetycznych: fenetyczne metody i kladystyczne metody. Ważne jest, aby pamiętać, że fenetycy i kladystycy mają niełatwe powiązania w ciągu ostatnich 40 lat. Większość obecnych biologów ewolucyjnych preferuje kladystyków, choć ściśle kladystyczne podejście może powodować nieścisłość wyników.
Fenetyczne metody analizy
Fenetyka, znana zarówno jako numeryczna taksonomia, wiąże się z użyciem różnych miar ogólnego podobieństwa rangi gatunków. Nie ma ograniczeń co do liczby i typu cech, które mogą być użyte, ale wszystkie dane muszą być najpierw konwertowane na numeryczną wartość, bez charakterystyki „wagi”. Każdy organizm może być wtedy porównany z każdym pod względem wszystkich mierzonych cech, a liczba podobieństw i różnic jest obliczana. Organizmy te są, następnie klastrowane w taki sposób, że większość podobieństw jest grupowana blisko razem a większość różnic jest podkreślanych bardziej daleko. Taksonomiczne klastry, nazywane fonogramami, które są wynikiem takich analiz nie musza odzwierciedlać genetycznego podobieństwa albo ewolucyjnych powiązań. Brak ewolucyjnego znaczenia w fenetyce, sprawił że system ten miał niewielki wpływ na klasyfikację zwierząt, a w konsekwencji, zainteresowanie i użycie go spada w ciągu ostatnich lat.
Kladystyczne metody analizy
Alternatywnym podejściem do diagramów związków między taksonami jest kladystyka. Podstawowym założeniem jest to, że członkowie grupy dzielą wspólną ewolucyjną historię. Dlatego, są oni bliżej związani ze sobą, niż do innych grup organizmów. Podobne grupy organizmów są rozpoznawalne, ponieważ dzielą zestaw unikalnych cech (apomorfizmy), które nie były obecne u odległych przodków, ale które są wspólne dla większości lub wszystkich organizmów w grupie. Te wspólne pochodne cechy nazywane są synapomofizmami. Dlatego w przeciwieństwie do fenetyków, kladystyczne grupy nie zależą od tego czy organizm dzieli fizyczne cechy, ale od ich ewolucyjnych związków. Rzeczywiście, kladystyczne analizy dwóch organizmów mogą dzielić liczne cechy, ale są uznawani za członków różnych grup.
Kladystyczna analiza pociąga za sobą szereg założeń. Na przykład, gatunki powstają głownie przez rozwidlenie, lub separację przodków linii, gatunki są często używane do tego aby wygasać po hybrydyzacji, a hybrydyzacja jest rzadka lub nieobecna. Ponadto, kladystyczne grupy muszą posiadać poniższe cechy: wszystkie gatunki w grupie muszą dzieli wspólnego przodka i wszystkie gatunki pochodzą ze wspólnego przodka muszą być wliczone w takson. Stosowanie tych wymogów powoduje w następujących terminach, że stają się używane do opisu różnych sposobów, według których mogą być robione grupy:
Dane makromolekularne, oznaczają geny (DNA) oraz sekwencje białkowe, które są akumulowane w zwiększających się stawkach, ponieważ zwiększają się osiągnięcia w dziedzinie biologii molekularnej. Dla biologów ewolucyjnych, szybkie nagromadzenie danych sekwencji z kompletnych genomów stało się głównym postępem, ponieważ cały charakter DNA pozwala mu być „dokumentem” w historii ewolucji. Porównanie sekwencji DNA różnych genów pomiędzy różnymi organizmami może powiedzieć naukowcom wiele na temat powiązań organizmów, które nie może być inaczej wnioskowane z morfologii, albo zewnętrznych cech organizmów lub jego struktur wewnętrznych. Ponieważ genomy rozwijają się przez stopniowe nagromadzenie mutacji, liczba różnych nukleotydów w sekwencji pomiędzy dwoma genomami z różnych organizmów powinna wskazywać jak często te dwa genomy dzieliły wspólnego przodka. Dwa genomy, które odbiegały w niedawnej przeszłości powinny mieć mniej różnic, niż dwa genomy które miały wspólnego przodka w starszym czasie. Dlatego też, przez porównanie różnych genomów ze sobą, powinno być możliwe do uzyskania ewolucyjnych powiązań między nimi, jest to główny cel filogenetyki molekularnej.
Molekularna filogenetyka próbuje ustalić stawki i modele zmian zachodzących w DNA i białkach oraz zrekonstruować historię ewolucji genów i organizmów. Dwa ogólne podejścia mogą być podjęte w celu uzyskania tych informacji. W pierwszym, naukowcy używają DNA do badań ewolucji organizmu. W drugim, różne organizmy są używane do badań ewolucji DNA. Niezależnie od podejścia, ogólny cel to wywnioskować proces z modelu: proces ewolucji organizmu wynikający z modeli zmienności DNA oraz proces molekularnej ewolucji wywnioskowany z modeli zmian tego DNA.
Molekularna analiza filogenetyczna: podstawowe elementy
Jak właśnie omówiliśmy, makromolekuły, przede wszystkim sekwencje genów i białek, przekroczyły morfologiczne oraz inne cechy organizmu jako najbardziej popularne formy danych do analizy filogenetycznej. W związku z tym następny rozdział będzie koncentrować się wyłącznie na danych molekularnych.
Ważne jest aby podkreślić, że pojedyncze, uniwersalne recepty nie istnieją w analizie filogenetycznej molekularnych danych. Mimo licznych algorytmów, procedur oraz rozwiniętych programów komputerowych, ich niezawodność i praktycznie (w każdym przypadku) zależy od rozmiaru i struktury zestawu danych do analizy. Zasadności i niedociągnięcia tych różnych metod są podstawą naukowej debaty, ponieważ niebezpieczeństwo przekazania niewłaściwych rezultatów jest większe w molekularnej filogenetyczne obliczeniowej niż w wielu innych dziedzinach nauki. Czasami, czynnikiem ograniczającym takie analizy nie jest tyle używana obliczeniowa metoda, co zrozumienie użytkownika co właściwie metoda ta robi z danymi. Dlatego celem tej sekcji jest demonstracja czytelnikowi, że praktyczne analizy powinny być traktowane zarówno jako wyszukiwanie właściwego modelu analizy, jak również do wyszukiwania prawidłowego drzewa.
Modele budowania drzewa filogenetycznego przyjmują ewolucyjne modele. Dla danego zbioru danych, modele te mogą być naruszone, z powodu różnych zdarzeń, takich jak transfer genetycznego materiału między organizmami. Dlatego, przy interpretacji danej analizy, osoba powinna zawsze rozważyć model stosowany i brać pod uwagę możliwe wyjaśnienia otrzymanych wyników. Na przykład, modele stosowane w metodach filogenetyki molekularnej powodują „default” – wartość domyślną założeń, w tym:
Budowanie drzewa: Najważniejsze funkcje drzew filogenetycznych oparte na DNA
Badania ewolucji genów i białek często dotyczą porównania homologów, sekwencje mające wspólnych pochodzenie mogą lub nie mieć podobne działanie. Sekwencje, które dzielą w dowolnym stopniu podobieństwo ustalone przez podstawowe dopasowanie alignment’u są homologami. Te sekwencje są dziedziczone od wspólnego przodka, posiadają podobną strukturę, mimo że przodek może być trudny do ustalenia, ponieważ został zmodyfikowany przez pochodzenie.
Homologi są najczęściej definiowane jako ortologów, paralogów i ksenologów.
Ortologi są to homologi produkowane przez specjację – reprezentują geny pochodzące ze wspólnego przodka, który odbiega, z powodu różnorodności organizmu. Ortologi mają podobne funkcje.
Paralogi są homologami produkowanymi przez duplikację genów i reprezentują genu pochodzące ze wspólnego przodka genu, który zduplikował w organizmie i został rozdzielony. Paralogi mają różne funkcje.
Ksenologi są homologami wynikającymi z horyzontalnego transferu genów miedzy dwoma organizmami. Funkcja ich może być zmienna, zależnie jak wielkie są zmiany w kontekście poziomego ruchu genów. Ogólnie jednak funkcje bywają podobne.
Typowe oparte na genach drzewo filogenetyczne zostało przedstawione poniżej. To drzewo pokazuje relacje między czterema homologami genów: A, B, C i D. Topologia tego drzewa składa się z czterech węzłów zewnętrznych, z których każdy reprezentuje jeden z czterech genów, oraz dwa wewnętrzne węzły (e i f) reprezentujące przodków genów. Długość gałęzi wskazuje stopień różnic ewolucyjnych między genami. To drzewo jest bez źródła – jest to tylko ilustracja powiązań między genami A, B, C i D i nie oznacza nic na temat serii ewolucyjnych zdarzeń, które doprowadziły do tych genów.
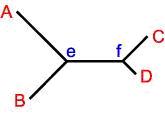
Drugi panel poniżej, przedstawia trzy drzewa ze źródłami, które można wyciągnąć z drzew bez źródeł pokazanych powyżej, każe reprezentuje inną drogę ewolucyjną możliwą między tymi czterema genami. Drzewo ze źródłami jest często określane jako wywnioskowane drzewo. Ma to na celu podkreślenie, że ten typ ilustracji przedstawia tylko serię ewolucyjnych wydarzeń, które są wywnioskowane z danych w ramach badań i mogą nie być takie same jak prawdziwe drzewo albo drzewo, które przedstawia aktualna serię wydarzeń ewolucyjnych, które nastąpiły.
Aby odróżnić ścieżki, analiza filogenetyczna musi zawierać co najmniej jedną poza-grupę, gen, który jest mniej związany z A, B, C i D, następnie geny te są do siebie prostopadłe (panel poniżej). Poza-grupy uniemożliwiają pojawienie się źródła drzewa oraz określenie prawidłowej drogi ewolucji. Powiedzmy, że cztery homologi użyte w poprzednich przykładach drzew pochodzą od człowieka, szympansa, goryla, oraz orangutana. W tym przypadku, poza-grupę może stanowić gen z innych naczelnych, jak pawian, o którym wiadomo że rozgałęzienie daleko od czterech gatunków poniżej przed wspólnym przodkiem tych gatunków.
Drzewo genów przeciwko drzewa gatunków – dlaczego są różne?
Zakłada się że drzewo genów, ponieważ jest oparty na danych molekularnych, będzie bardziej precyzyjne i mniej dwuznaczną reprezentacją drzewa gatunków, niż te oparte na morfologicznych porównaniach. Może to rzeczywiście być powodem, ale to nie znaczy że drzewa genów są tym samym co drzewa gatunków. Aby to było prawdziwe, wewnętrzne węzły w obu drzewach musiałyby być swoimi precyzyjnymi odpowiednikami, a nie są. Wewnętrzny węzeł drzewa genów wskazuje rozbieżności przodków genów w dwa genu z różną sekwencją DNA, zwykle wynikające z mutacji jednego lub innego rodzaju. Wewnętrzny węzeł w drzewie gatunków pokazuje tzw. wydarzenie przypadku, w którym populacja przodka gatunków dzieli się na dwie grupy, które nie są już w stanie się krzyżować. Te dwa wydarzenia, mutacji i specjacji, nie zawsze występują w tym samym czasie.
Terminologia filogenetyki molekularnej
Monofiletyczny: dwie lub więcej sekwencje DNA, które pochodzą ze wspólnego przodka sekwencji DNA
Klad: grupa monofiletycznych sekwencji DNA, która tworzy wszystkie sekwencji w analizie, które pochodzą od wspólnych przodków danej sekwencji.
Oszczędność: podejście, które decyduje między różnymi topologiami drzew przez określenie jednego, które obejmuje najkrótszą drogę ewolucyjną. Jest to droga, która wymaga najmniejszej liczby zmian nukleotydów pochodzących z sekwencji przodka, źródła drzewa, do wszystkich dzisiejszych sekwencji, które zostały porównane.
Hipoteza molekularnego zegara: stwierdza, że substytucje nukleotydów, albo aminokwasów jeśli są porównywane białka, występują w stałej stawce (puli), jest to stopień różnić między dwoma sekwencjami, który może być użyty aby przypisać dane do czasu w którym się rozdzieliły z przodkiem. Stawka molekularnych zmian różni się wśród grup organizmów, genów i nawet wśród różnych fragmentów tego samego genu. Co więcej, molekularny zegar wymaga kalibracji z skamieniałości aby określić harmonogram rzędu kladów, a więc ich dokładność jest w dużej mierze zależna od kopalnego rekordu, lub jej braku, dla badanych grup. Kopalne DNA starsze niż około 25 000- 50 000 lat jest prawie puste filogenetycznie z wyjątkiem rzadkich przypadków, a więc tradycyjne morfologiczne badania wymarłych i żyjących organizmów pozostają istotnym elementem analizy filogenetycznej.
Projekt taksonomii
Celem projektu taksonomii NCBI jest zbudowanie spójnej filogenetycznej taksonomii dla sekwencji baz danych NCBI. W taksonomicznej bazie danych znajdują się opisy i linie każdego organizmu reprezentowanego przynajmniej przez jeden nukleotyd lub sekwencję białkową w genetycznej bazie danych. W lutym 2003, zawierała ta baza ponad 250 000 taksonów. Aby uzyskać informacje aktualne, odwiedź Taxonomy Statistics Web page. Baza danych jest rozpoznawana jako norma odniesienia przez współpracujące międzynarodowe bazy danych sekwencji (GenBank, EMBL, DDJB).
Przeglądarka taksonomii jest w NCBI i pochodzi z wyszukiwania narzędzia, który pozwalałby na przeszukiwanie bazy danych taksonomii. Korzystając z przeglądarki, informacje mogą być pobierane na podstawie dostępnych nukleotydów, białek i struktur dla danego gatunku lub wyższego taksonu. Może być ona też użyta w celu wyświetlenia taksonomicznej pozycji lub do pobrania sekwencji i struktury danych z poszczególnego organizmu lub grupy organizmów. Wyszukiwanie może opierać się na całości, części lub fonetycznej pisowni nazwy, oraz dawać bezpośrednie linki do wspólnych organizmów używanych w biologicznych wyszukiwaniach. Taksonomia systemu entrez ma możliwość wyświetlania niestandardowych taksonomicznych drzew reprezentujących zdefiniowane przez użytkownika podzbiory całej taksonomii NCBI.
TaxPlot, kolejny składnik projektu taksonomii, jest narzędziem do prowadzenia porównań różnych genomów na trzy różne sposoby. Porównania oparte są na sekwencji białek zakodowanych w genomie tych organizmów. Aby korzystać z TaxPlot, jeden wybiera odniesienie dla genomu do którego z dwóch innych genomów będzie porównywany. Narzędzie taxPlot następnie wykorzystuje BLAST, opisuje wynik jako punktu na macierzy dla każdego białka prognozowanego aby być w opisywanym genomie.
BLAST: wykrywanie nowych podobieństw sekwencji
Obecnie, cechami najczęściej używane w analizie filogenetycznej są sekwencje DNA i białek. Sekwencje DNA mogą być porównywane bezpośrednio, lub dla tych regionów które kodują znane białka, przetłumaczone z sekwencji białkowej. Tworzenie filogenetyki z sekwencji nukleotydów lub aminokwasów wymaga wyrównania matrycy, tak aby różnice między sekwencjami były badane w łatwiejszy sposób.
Wprowadzenie przez NCBI BLAST, w 1990 roku sprawiło że przeszukiwanie baz danych w poszukiwaniu homologów albo podobnych sekwencji stało się szybsze i łatwiejsze i możliwa jest ocena statystyczna uzyskanych dopasowań. BLAST działa przez porównywanie nieznanej sekwencji użytkownika do znanych sekwencji baz danych dobranych przez dopasowanie. W ciągu kilku sekund, BLAST porównuje sekwencje użytkownika z milionem znanych sekwencji i określa najbliższe dopasowanie.
Specjalistyczne BLASTy są także dostępne dla ludzi, myszy, bakterii i wiele innych genomów. Jedno wyszukiwanie BLAST może porównać sekwencje do innych sekwencji GenBanku. W tym kroku, naukowiec ma możliwość opcji ograniczenia wyszukiwania do konkretnych grup taksonomicznych. Jeśli pełna nazwa naukowa lub powiązania gatunków nie są znane, użytkownik może wyszukiwać takie dane używając wyszukiwarki taksonomicznej, która dostarcza bezpośrednich linków do niektórych organizmów powszechnie używanych w projektach molekularnych poszukiwań, takie jak zebrafish, fruit fly, baker yeast i wiele innych.
BLAST następnie mówi o różnicach między sekwencjami i przypisuje liczbę punktów opartą na podobieństwie sekwencji. Ocena ta, ma dobrze zdefiniowaną statystyczną interpretację, robiącą prawdziwe dopasowania sekwencji prościej w odróżnieniu od losowego trafienia. Dzieje się tak dlatego, że BLAST korzysta ze specjalnego algorytmu, albo matematycznej formuły, która szuka lokalnego w przeciwieństwie do globalnego alignmentu i dlatego jest w stanie wykryć powiązania wśród sekwencji, które dzielą tylko odizolowane regiony podobieństwa. Taksonomiczne związane rezultaty BLAST są prezentowane w trzech formatach. Organizm Report sortuje porównania BLAST, nazywanego też hitami, przez takie gatunki, że wszystkie hity są zgrupowane razem. Lingeage Report dostarcza widoku powiązań organizmów. Taxonomy Report dostarcza szczegółowych danych o powiązaniach pomiędzy organizmami w liście hitów BLAST.
COG: filogenetyczna klasyfikacja białek
Baza Clusters of Orthologous Groups białek (COGs) stanowi próbę na filogenetyczną klasyfikację białek, system, który wskazuje powiązania ewolucyjne pomiędzy organizmami, od całych genomów. Każdy COG obejmuje białka, które są uznawane za ortologi, albo powiązane przez ewolucyjne pochodzenie. COG mogą być używane do wykrycia podobieństw i różnic między gatunkami, do identyfikacji rodziny białek i przewidywania nowych funkcji białek, oraz aby wskazać potencjalne leki mające znaczenie w chorobach gatunków. Baza danych jest wraz z programem COGnitor, który wyznacza nowe białka, zazwyczaj z nowych sekwencji genomów do istniejących wcześniej COGs. Te podkreślone strony informacyjne zawierają: system klasyfikacji członków COG pod wpływem różnych klasyfikacji; wskazania, który z członków COG (jeśli jakiś) był charakteryzowany genetycznie i biochemicznie; informacje o strukturze domen białek tworzących COG i ich trójwymiarową strukturę jeśli są znane lub przewidywalne; zwięzłe podsumowanie powszechnych struktur oraz cech funkcjonalnych członków COG, jak też specyfikę poszczególnych członków; klucze odniesienia.
HomoloGene
Jest bazą danych, obu wyznaczonych i obliczonych ontologów i homologów dla organizmu znajdującego się w bazie danych UniGene. Wyznaczone ortologi zawierają pary genów z MGD (Mouse Genome Database), ZFIN (Zebrafish Information). Komputerowe ortologi i homologi są identyfikowane z porównywania nukleinowej sekwencji BLAST między wszystkimi klastrami UniGene dla każdej pary organizmów. Zawiera on również zestaw potrójnych klastrów, w których ortologi dwóch organizmów są ontologami tego samego klastra w trzecim organizmie.
Entrez Genome
Całe genomy ponad 1 200 organizmów mogą być znalezione w Entrez Genome. Genomy reprezentują oba kompletnie zsekwencjonowe organizmy i te których sekwencje są w trakcje sekwencjonowania. Trzy główne domeny życia – bakterie, archaea, i eukarioty – są reprezentowane, jak również wirusy, wiroidy, plazmidy i organelle eukariotyczne. Dane mogą być dostępne w hierarchicznym porządku zaczynając od listy alfabetycznej albo filogenetycznych drzew dla całych genomów w każdym z sześciu zasadniczych grup taksonomicznych. Można podążać za hierarchią różnych podglądów graficznych, wliczając całe genomy pojedynczych organizmów, chromosomów lub nawet genów. Na każdym poziomie, można uzyskać wiele podglądów danych, komputerowe przeliczenia i linki do analiz odpowiednich dla tego poziomu. Ponadto, każdy produkt genu (białko), które jest członkiem grupy COG ma odnośnik do COG bazy danych. Podsumowanie funkcjonalnych grup COG jest także prezentowane w tabelarycznych i graficznych formatach na poziomie genomu.
Aby uzyskać pełne genomy bakterii, wliczając ich dystrybucję taksonomiczną i linki do 3D struktury są podane TaxTables i PDBTables. Pairwise sekwencji alignmentu jest prezentowany graficznie i posiada linki do Cn3D wyświetlenia makromolekuł, które pozwala na interaktywne wyświetlenie 3D struktur i sekwencji alignmentu.
PDBeast: Taksonomia w MMDB
Struktura grup NCBI, we współpracy z taksonomistami NCBI, podjęła się adnotacji do taksonomii dla 3D struktur zebranych w MMDB (Molekular Modeling Database). Półzautomatyzowane podejście zostało wdrożone i jest sprawdzane przez ludzkich ekspertów, korygowane i potwierdzane automatycznie w MMDB. Oprogramowanie narzędzia PDBeast zostało opracowane przez NCBI w tym celu. Wyciąga ono opisy „Źródła organizmów” albo z oryginalnej pozycji albo specyficznych informacji użytkownika i szuka dopasowania w bazie danych Taksonomii NCBI w celu rejestrowania taksonomicznych powiązań.
Dziedzina filogenetyki molekularnej wzrosła, zarówno pod względem ważności jak i rozmiarów, od kiedy powstała w 1990 roku, można jej głównie przypisać postęp w biologii molekularnej i bardziej rygorystyczne metody budowania drzew filogenetycznych. Znaczenie filogenetyki również znacznie wzrosło przez udane wdrożenie budowy drzewa, jak również technik filogenetycznych, do bardziej zróżnicowanych i kłopotliwych zagadnień biologii. Dzisiaj, przegląd literatury naukowej pokaże, że biologia molekularna, genetyka, ewolucja, rozwój, zachowanie, epidemiologia, ekologia, systematyka, zachowanie stałości biologii są tylko kilkom egzemplarzami dla wielu odległych dziedzin koncepcyjnie zjednoczonych przez używanie metod i teorii molekularnej filogenetyki. Filogenetyka jest używana zasadniczo w ten sam sposób jak wszystkie te dziedziny, albo przez rysowanie różnic w strukturze drzewa, albo w charakteryzowaniu map na drzewie. Biolodzy mogą następnie korzystać z tych wskazówek do tworzenia hipotez i modeli ważnych wydarzeń w historii. Ogólnie rzecz ujmując, ustanowione powiązania przez drzewa filogenetyczne często opisują historię ewolucji gatunku, a tym samym jego filogenezę – historyczne powiązania wśród linii lub organizmów albo ich części, takich jakimi są geny. Filogenetyka może być traktowana jako naturalny i znaczący sposób na uporządkowanie danych, z wielką liczbą ewolucyjnych informacji zawartych je jej gałęziach. Naukowcy pracujący w tych różnych dziedzinach mogą następnie użyć filogenetyki do badań i wyjaśniania procesów biologicznych występujących na wielu poziomach hierarchicznego życia.